【机器学习:异常值检测】新颖性和异常值检测
【机器学习:异常值检测】新颖性和异常值检测
-
- 异常值检测方法概述
- 新颖性检测
-
- 纵向扩展单类 SVM
- 异常值检测
-
- 拟合椭圆包络
- 隔离林
- 局部异常因子
- 使用局部异常因子进行新颖性检测
许多应用程序需要能够确定新观测值是属于与现有观测值相同的分布(它是异常值),还是应被视为不同的分布值(它是异常值)。通常,此功能用于清理真实数据集。必须做出两个重要的区别:
| outlier detection: 异常值检测: | 训练数据包含异常值,这些异常值被定义为与其他观测值相去甚远的观测值。因此,异常值检测估计器试图拟合训练数据最集中的区域,忽略偏差观测值。 |
|---|---|
| novelty detection: 新颖性检测: | 训练数据不会受到异常值的污染,我们有兴趣检测新的观测值是否为异常值。在这种情况下,anoutlier 也称为新颖性。 |
异常值检测和新颖性检测都用于异常检测,其中人们有兴趣检测异常或异常观察结果。异常值检测也称为无监督异常检测,新颖性检测也称为半监督异常检测。在异常值检测的上下文中,异常值/异常值不能形成密集聚类,因为可用的估计器假设异常值/异常值位于低密度区域。相反,在新颖性检测的上下文中,新奇/异常可以形成密集的聚类,只要它们位于训练数据的低密度区域,在这种情况下被认为是正常的。
scikit-learn 项目提供了一组机器学习工具,可用于新颖性或异常值检测。该策略是通过从数据中以无监督方式进行对象学习来实现的:
estimator.fit(X_train)
然后,可以使用以下predict方法将新观测值排序为异常值或异常值:
estimator.predict(X_test)
Inliers 标记为 1,而异常值标记为 -1。预测方法使用由估计器计算的原始评分函数的阈值。该评分函数可通过该score_samples方法访问,而阈值可以通过contamination参数控制。
该decision_function方法也是从评分函数中定义的,负值是异常值,非负值是异常值:
estimator.decision_function(X_test)
neighbors.LocalOutlierFactor请注意,默认情况下predict不支持 和 score_samples 方法,但只是一个fit_predict方法,decision_function因为此估计器最初用于异常值检测。训练样本的异常分数可通过属性negative_outlier_factor_访问。
如果你真的想用于neighbors.LocalOutlierFactor新颖性检测,即预测标签或计算新未见数据的异常分数,你可以True在拟合估计器之前,使用novelty参数集实例化估计器。在这种情况下,fit_predict不可用。
警告:局部离群因子预警新颖性检测
当新颖性被设置为True注意,您只能使用predict新的看不见的数据,decision_function score_samples而不是训练样本,因为这会导致错误的结果。predict fit_predict训练样本的异常分数始终可以通过属性negative_outlier_factor_访问。
下表总结了其neighbors.LocalOutlierFactor行为。
| 方法 | 异常值检测 | 新颖性检测 |
|---|---|---|
| fit_predict | 还行 | 不可用 |
| predict | 不可用 | 仅用于新数据 |
| dicision_function | 不可用 | 仅用于新数据 |
| score_samples | 使用negative_outlier_factor_ |
仅用于新数据 |
| negative_outlier_factor | 还行 | 还行 |
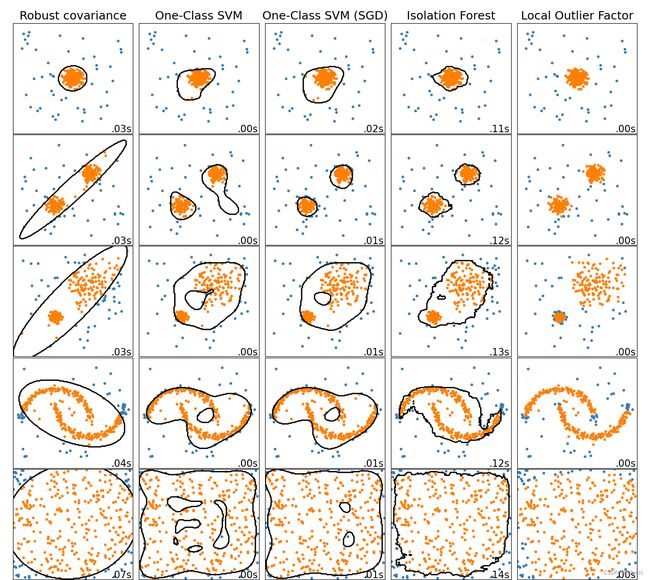
异常值检测方法概述
scikit-learn 中异常值检测算法的比较。LocalOutlier Factor (LOF) 不会以黑色显示决策边界,因为当用于异常值检测时,它没有可应用于新数据的预测方法。
ensemble.IsolationForest并且neighbors.LocalOutlierFactor在这里考虑的数据集上表现相当好。众所周知,它svm.OneClassSVM对异常值很敏感,因此在异常值检测方面表现不佳。话虽如此,在高维中检测异常值,或者对内围数据的分布没有任何假设,是非常具有挑战性的。svm.OneClassSVM仍可与异常值检测一起使用,但需要微调其超参数nu以处理异常值并防止过度拟合。linear_model.SGDOneClassSVM提供非线性单类支持向量的实现,其样本数复杂度呈线性。此实现与内核近似技术一起使用,以获得与默认使用高斯内核类似的svm.OneClassSVM结果。最后,covariance.EllipticEnvelope假设数据是高斯的,并学习一个椭圆。有关不同估计器的更多详细信息,请参阅示例比较玩具数据集上异常值检测的异常检测算法以及以下部分。
例子:
-
请参阅比较玩具数据集上异常值检测的异常检测算法,以比较 、
svm.OneClassSVM、ensemble.IsolationForestneighbors.LocalOutlierFactor和covariance.EllipticEnvelope。 -
请参阅异常值检测估计器的评估,了解如何使用 ROC 曲线评估异常值检测估计器、和
neighbors.LocalOutlierFactorensemble.IsolationForestmetrics.RocCurveDisplay的示例。
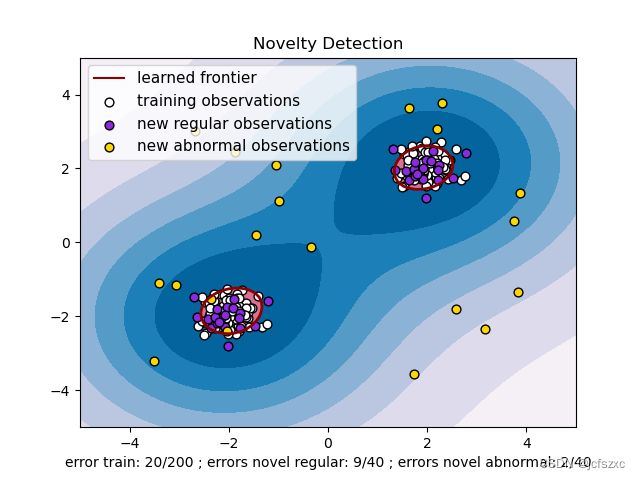
新颖性检测
考虑来自特征描述的相同分布的观测值数据集。 现在考虑向该数据集再添加一个观测值。新的观察结果是否与其他观察结果大相径庭,以至于我们可以怀疑它是否是有规律的?(即它是否来自同一发行版?或者恰恰相反,它是否与另一个如此相似,以至于我们无法将其与原始观察区分开来?这是新颖性检测工具和方法所要解决的问题。
一般来说,它即将学习一个粗略的、紧密的边界,划定初始观测值分布的轮廓,绘制嵌入维空间。然后,如果进一步的观测在边界划定的子空间内,则认为它们来自与初始观测相同的种群。否则,如果它们位于边界之外,我们可以说它们是异常的,并且对我们的评估有一定的信心。
为此,Schölkopf 等人引入了单类 SVM,并在svm.OneClassSVM对象的支持向量机模块中实现。它需要选择内核和标量参数来定义边界。 RBF 内核通常被选择,尽管没有确切的公式或算法来设置其带宽参数。这是 scikit-learn实现中的默认值。该nu参数也称为单类支持向量机的余量,对应于在边界之外找到新的但有规律的观测值的概率。
引用:
- Estimating the support of a high-dimensional distribution Schölkopf, Bernhard, et al. Neural computation 13.7 (2001): 1443-1471.
例子:
- See One-class SVM with non-linear kernel (RBF) for visualizing the frontier learned around some data by a svm.OneClassSVM object.
请参阅具有非线性内核 (RBF) 的单类 SVM,以可视化svm.OneClassSVM对象围绕某些数据学习的前沿。 - Species distribution modeling 物种分布建模
纵向扩展单类 SVM
实现了单类 SVM 的在线线性版本linear_model.SGDOneClassSVM。此实现随样本数线性缩放,并且可以与核近似一起使用,以近似于核化svm.OneClassSVM(其复杂性充其量是样本数的二次)的解。有关详细信息,请参见“在线单类 SVM”部分。
例子:
- 请参阅使用随机梯度下降的单类 SVM 与单类 SVM,了解核化单类 SVM 与核近似
linear_model.SGDOneClassSVM相结合的近似图示。
异常值检测
异常值检测类似于新颖性检测,其目标是将常规观测值的核心与一些污染的观测值(称为异常值)分开。然而,在异常值检测的情况下,我们没有一个干净的数据集来表示可用于训练任何工具的常规观测值的总体。
拟合椭圆包络
执行异常值检测的一种常见方法是假设常规数据来自已知分布(例如,数据是高斯分布的)。根据这个假设,我们通常尝试定义数据的“形状”,并且可以将离远观测值定义为与拟合形状相距足够远的观测值。
scikit-learn 提供了一个对象,该对象covariance.EllipticEnvelope将鲁棒协方差估计拟合到数据,从而将椭圆拟合到中心数据点,忽略中心模式之外的点。
例如,假设内位数据是高斯分布的,它将以稳健的方式估计内位和协方差(即不受异常值的影响)。从该估计中获得的马氏距离用于推导出外围度的度量。此策略如下图所示。
例子:
- 请参阅稳健协方差估计和马氏距离相关性,以说明使用位置和协方差的标准
(covariance.EmpiricalCovariance)或稳健估计(covariance.MinCovDet)来评估观测值的离远程度之间的区别。
引用:
- Rousseeuw, P.J., Van Driessen, K. “A fast algorithm for the minimum covariance determinant estimator” Technometrics 41(3), 212 (1999)
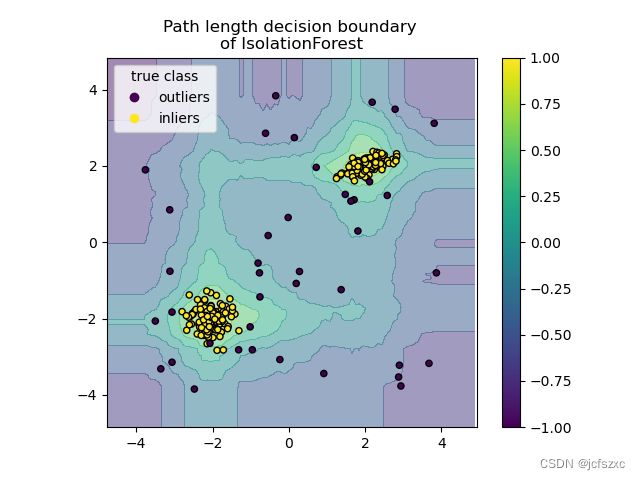
隔离林
在高维数据集中执行异常值检测的一种有效方法是使用随机森林。ensemble.IsolationForest 通过随机选择一个特征,然后在所选特征的最大值和最小值之间随机选择一个分割值来“隔离”观测值。
由于递归分区可以用树结构表示,因此隔离样本所需的拆分次数相当于从根节点到终止节点的路径长度。
这个路径长度,在这种随机树的森林中取平均值,是正态性和我们的决策函数的度量。
随机分区会为异常情况生成明显更短的路径。因此,当随机树的森林共同为特定样本产生较短的路径长度时,它们极有可能是异常。
集成的实现。ensemble.IsolationForest是基于tree. extraterereegressor的集合。根据隔离森林的原始论文,每棵树的最大深度被设置为 ⌈ l o g 2 ( n ) ⌉ \lceil log_2{(n)}\rceil ⌈log2(n)⌉,其中是用于构建树的样本数量(详见(Liu et al., 2008))。
该算法如下图所示。
ensemble.IsolationForest允许您向已拟合的模型添加更多树的支撑warm_start=True:
from sklearn.ensemble import IsolationForest
import numpy as np
X = np.array([[-1, -1], [-2, -1], [-3, -2], [0, 0], [-20, 50], [3, 5]])
clf = IsolationForest(n_estimators=10, warm_start=True)
clf.fit(X) # fit 10 trees
clf.set_params(n_estimators=20) # add 10 more trees
clf.fit(X) # fit the added trees
例子:
- 请参阅 IsolationForest 示例,了解 IsolationForest 的用法说明。
- 请参阅比较玩具数据集上异常值检测的异常值检测算法,以比较
ensemble.IsolationForest和svm.OneClassSVM(neighbors.LocalOutlierFactor调整为执行类似于异常值检测方法),linear_model.SGDOneClassSVM以及 的基于协方差的异常值检测。covariance.EllipticEnvelope
引用:
- Liu, Fei Tony, Ting, Kai Ming and Zhou, Zhi-Hua. “Isolation forest.” Data Mining, 2008. ICDM’08. Eighth IEEE International Conference on.
局部异常因子
对中等高维数据集执行异常值检测的另一种有效方法是使用局部异常值因子 (LOF) 算法。
(LOF)算法计算一个分数neighbors.LocalOutlierFactor(称为局部异常值因子),以反映观测值的异常程度。它测量给定数据点相对于其相邻点的局部密度偏差。这个想法是检测密度明显低于其相邻样本的样本。
在实践中,局部密度是从 k 个最近邻获得的。观测值的LOF得分等于其k最近邻的平均局部密度与其自身局部密度的比值:正常实例的局部密度预期与其相邻实例的局部密度相似,而异常数据的局部密度预计要小得多。
通常选择所考虑的邻居数 k(别名参数 n_neighbors)大于聚类必须包含的最小对象数,以便其他对象可以是相对于该聚类的局部异常值,以及 2)小于可能成为局部异常值的邻近对象的最大数量。在实践中,此类信息通常不可用,takingn_neighbors=20 似乎总体上效果很好。当异常值的比例很高(即大于 10%,如下例所示)时,n_neighbors应更大(在以下示例中为 n_neighbors=35)。
LOF算法的优势在于它同时考虑了数据集的局部和全局属性:即使在异常样本具有不同底层密度的数据集中,它也能很好地表现。问题不在于样本的隔离程度,而在于它与周围社区的隔离程度。
当应用 LOF 进行异常值检测时,没有 predict和decision_function score_samples 方法,而只有一种方法fit_predict。训练样本的异常分数可通过该negative_outlier_factor_属性访问。请注意,predict当 LOF 用于新颖性检测时,即当novelty参数设置为 True时,可以score_samples对新的看不见的数据使用 ,decision_function但结果predict可能与 的结果不同fit_predict。请参阅使用局部异常值因子进行新颖性检测。
此策略如下图所示。
例子:
-
有关使用 的说明,请参见使用局部异常值因子 (LOF) 进行异常值检测
neighbors.LocalOutlierFactor。 -
有关与其他异常检测方法的比较,请参阅比较玩具数据集上异常值检测的异常检测算法。
引用:
- Breunig, Kriegel, Ng, and Sander (2000) LOF: identifying density-based local outliers. Proc. ACM SIGMOD
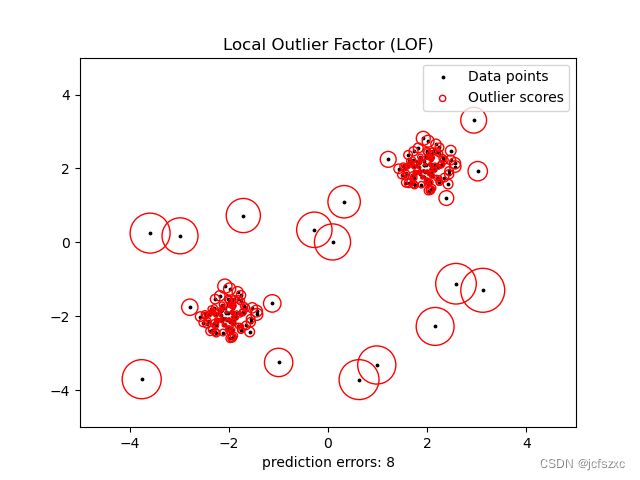
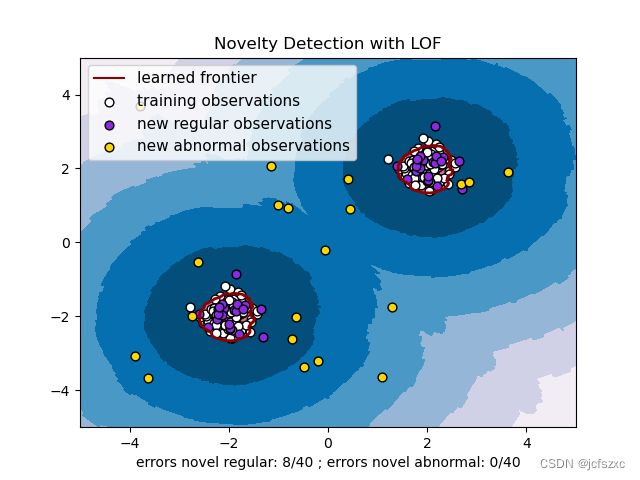
使用局部异常因子进行新颖性检测
要用于neighbors.LocalOutlierFactor新颖性检测,即预测标签或计算新的未查看数据的异常分数,您需要True在拟合估计器之前使用novelty参数集实例化估计器:
lof = LocalOutlierFactor(novelty=True)
lof.fit(X_train)
请注意,fit_predict为避免不一致,在这种情况下不可用。
局部离群因子预警新颖性检测
当新颖性被设置为True注意,您只能使用predict新的看不见的数据,decision_function score_samples而不是训练样本,因为这会导致错误的结果。predict fit_predict训练样本的异常分数始终可以通过属性negative_outlier_factor_访问。
使用局部异常值因子进行新颖性检测如下图所示。