如何用R的绘制高颜值的列线图、校准曲线和DCA曲线?
继上次绘制模型验证,列线图、校准曲线、DCA图这种已经不在话下,看的一些文献也是这种的图片,如下:
 这种简单线条,最下面一条就是相应患病风险了,那现在我想做点高级点的,像这种
这种简单线条,最下面一条就是相应患病风险了,那现在我想做点高级点的,像这种
不仅是有线条的变化,同个指标上的不同结局还给画的不一样,哪怕是简单的分类变量,也会根据评分的高低绘制不同的形状;有些高级列线图,还会对连续型变量采用直方图的形式表示,或者是在预测概率上添加不同颜色表示中、低、高范围,这些操作其实是让图片看起来更加容易解读,当然,也看起来逼格高些。闲着也是闲着,不如就试试看,至少画出个彩色的列线图、ADC图也好呀,换个不同的风格,尝试用不同的R包。
第一步:CSV格式的数据展示
这里模拟了A、B、C、D、E共计5个变量,结局变量是因变量,本次要研究的结局了。注意我这里的数据展示仍然是二分类,且代表结局的数值是0和1,不能是汉字或者其他的数字,不然在后续的操作中会报错。当然,为了方便,有些大佬会输入字符转换的代码,但我把那步骤给删掉了,就还是按照0和1的步骤来。
第二步:输命令
Mydata<-read.csv(file.choose() ,header = TRUE,
fileEncoding = "GBK") #我习惯打开文件的命令,大多数文件都能打开
head(Mydata) #初步展示前六行的数据
View(Mydata) #查看下我的数据,习惯所致,这步可要可不要
library(regplot) #用regplot绘制列线图
预测模型<-glm(结局~A+B +C+ D+E,
family=binomial(link = logit), data=Mydata) #用glm拟合函数
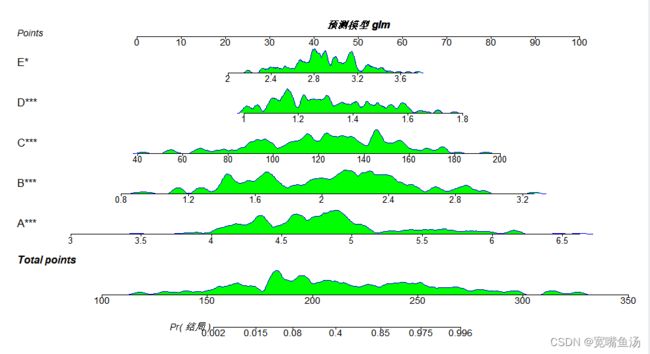
regplot(预测模型, plots=c("density","boxes")
,observation=FALSE
,points=TRUE,dencol="green",boxcol="green") #我自己选择的green绿色,也可以选择其他颜色。
第三步:列线图的结果如下
看上去还可以
第四步:彩色校准曲线方法一
当然,完成了好看的列线图后,校准曲线的命令+ADC决策曲线的都安排上
library(Hmisc)
library(rms)
dd<-datadist(Mydata) #开始打包数据
options(datadist="dd")
f_lrm <-lrm(HUA~A+B+C +D+ E
, data=Mydata) #用lrm构建函数
fit2 <- lrm(f_lrm, data = Mydata, x=TRUE,y=TRUE) #注意这里的f_lrm是在我前面的命令中设置的
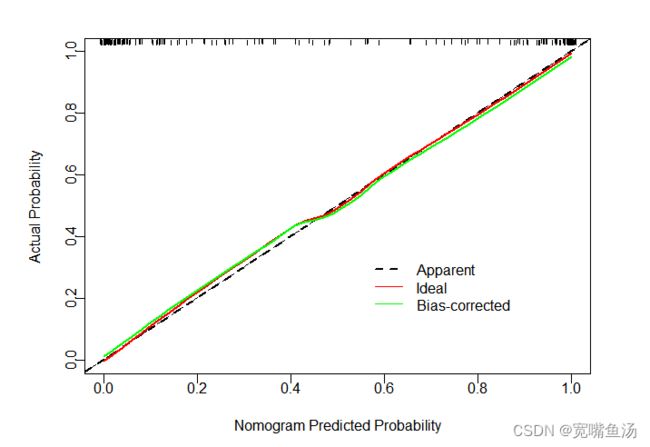
cal1 <- calibrate(fit2, method = "boot", B=1000)
par(mgp=c(2.4,0.3,0),mar=c(7,7,3,7)) #设置画布的命令,我会提前设置好,不然画面会很大很大,mgp和mar都是设置画面以及坐标轴参数的相关命令,需要的话可以直接搜这两个命令的含义
plot(cal1,
xlim = c(0,1),
xlab = "Nomogram Predicted Probability",
ylab = "Actual Probability",
legend = FALSE,
subtitles = FALSE)
abline(0,1,col = "black",lty = 2,lwd = 2)
lines(cal1[,c("predy","calibrated.orig")],
type = "l",lwd = 2,col="red",pch =16)
lines(cal1[,c("predy","calibrated.corrected")],
type = "l",lwd = 2,col="green",pch =16)
legend(0.55,0.35, ##legend是绘制图例的函数,有兴趣的可以去深入了解这个函数
c("Apparent","Ideal","Bias-corrected"), #表示曲线名称的集合
lty = c(2,1,1), #lty表示线条类型,1代表实线,2至6都是虚线,虚的程度不一样
lwd = c(2,1,1), #lwd表示宽度,以默认值的相对大小来表示宽度
col = c("black","red","green"), #给曲线添加颜色,对应上面c("Ax","Ix","Bx")
bty = "n") # bty为o表示加边框,,注意是字母,不是数字0。bty可以取6种字符,分别为“o”、“l”、“7”、“c”、“u”、“]”第四步:彩色校准曲线方法二
这次我们用的是“riskRegression”程序包,命令如下:
library(riskRegression)
par(mgp=c(2.8,0.5,0),#xlab和ylab的位置、坐标轴数字的位置、坐标轴轴线的位置
mar=c(6,6,3,6)) #设置画布的命mar可以指定四个边的边距-下、左、上、右
xb <- Score(list("fit"=fit2),formula= 结局~1,
conf.int = TRUE,
plots = c("calibration","ROC"),
metrics = c("auc","brier"),
B=1000,M = 50,
data= Mydata)
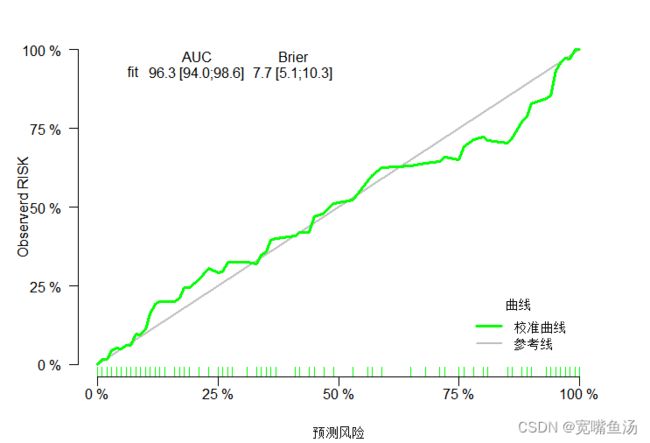
plotCalibration(xb,col="green") #注意这里的fit2就是前面步骤里的fit2 <- lrm(f_lrm, data = Mydata, x=TRUE,y=TRUE)这种绘制出来的校准曲线就是这样了
 只是我觉得这个函数我能动的比较少,我想改个参数什么的一下子还不知道怎么从哪里动手,于是找了个类似的命令。这个命令的其实是和前面的命令一样的,不过把这个函数的内容更加具体化,比如X、Y轴的命名, 我们都可以改一改了,图片却是一样的
只是我觉得这个函数我能动的比较少,我想改个参数什么的一下子还不知道怎么从哪里动手,于是找了个类似的命令。这个命令的其实是和前面的命令一样的,不过把这个函数的内容更加具体化,比如X、Y轴的命名, 我们都可以改一改了,图片却是一样的
fit2 <- glm(结局~A+B+C +D+ E
, data=Mydata
, family = binomial)
library(riskRegression)
par(mgp=c(2.8,0.5,0),#xlab和ylab的位置、坐标轴数字的位置、坐标轴轴线的位置
mar=c(6,6,3,6))
fit22 <- Score(list("fit"=fit2),
formula = 结局 ~ 1,
data = Mydata,
metrics = c("auc","brier"),
summary = c("risks","IPA","riskQuantile","ibs"),
plots = "calibration",
null.model = T,
conf.int = T,
B = 500,
M = 50)
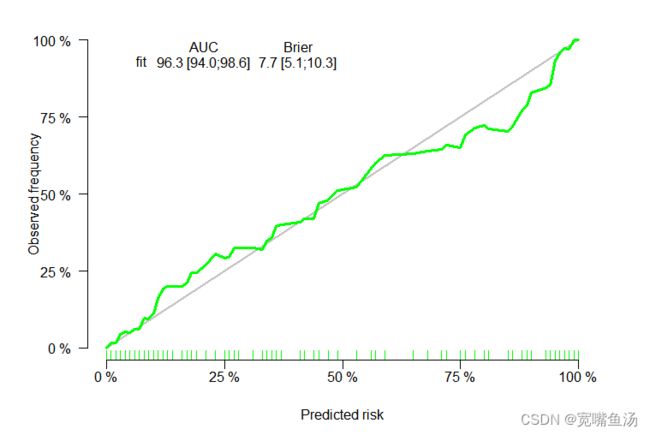
plotCalibration(fit22,col="green",
xlab = "",
ylab = "Observerd RISK",
bars = F)第四步:彩色校准曲线之图例
这种校准曲线我也觉得还不错,但是总想着给它加上图例,就是曲线说明,于是研究了下legend这个函数,给加了个图例。这种后天加上去的图例会更好,不然有些时候 ,图例会遮住线条,我就遇见过好几次了。
legend("bottomright" #图例在右下
, inset=.05, title="曲线", c("校准曲线","参考线"),
bty="n", #不添加边框
lty=c(1, 1), #增加线条的情况,1表示实线
lwd=c(3, 2),#增加线条的粗细情况,我想要绿色粗一点,灰色细一点
col=c("green", "Grey")) #图例添加完成写到最后
这期间最让我头疼的其实是图例与曲线的问题,还有就是在用regplot绘图遇到的问题了,还是要回归到函数本身。如果是想做cox回归模型,也可以用survival里面的reg函数。
regplot(reg, plots=c("density","boxes"), center=TRUE, observation=NULL, title=NULL, points=FALSE, failtime=NULL, prfail=NULL, baseS=NULL, odds=FALSE, nsamp=10000, showP=TRUE, rank=NULL, subticks=FALSE, interval=NULL, clickable=FALSE, ...)
reg:绘制列线图的回归方程,可以是来自程序包 stats、survival、rms、MASS 和 lme4,可支持的命令有glm,Glm,lm,ols,lrm,survreg,psm,coxph,cph,glm.nb,polr 或混合回归模型 lmer、glmer、glmer.nb。
plots:指定预测变量的展示图类型,需要同时指定数值型变量和因子变量的绘图类型,默认plots=c("density","boxes") 指定数值型变量为密度图和分类变量为箱线图。第一项是设置非因子变量的绘图,绘图类型有:“no plot”[不绘图]、“density”[密度图]、“boxes”[盒状图]、“spikes”[尖峰图]、“ecdf”[经验累积分布函数]、“bars”[条形图]、“boxplot”[箱线图]、 “violin”[小提琴图]或“bean”;第二项是因子变量的绘图,绘图类型有:"no plot", "boxes", "bars" or "spikes"。
~~~~~~~~~~~~11111111111111·````````````````~~~~~~~~~~~~~~~~~111111111111111111`·~~~~~~~
由于我这里的A~E这5个指标都是连续变量,我写的是默认的density,后面的boxes 起不到什么作用,没有分类变量。于是乎把plots=c("density","boxes")改为了plots=c("violin","boxes") ,具体如下
library(regplot)
预测模型<-glm(结局~A+B+C +D+ E,
family=binomial(link = logit), data=Mydata) #用glm拟合函数
regplot(预测模型, plots=c("violin","boxes") #改为violin,看下不同形状的
,observation=FALSE
,points=TRUE,dencol="green",boxcol="green")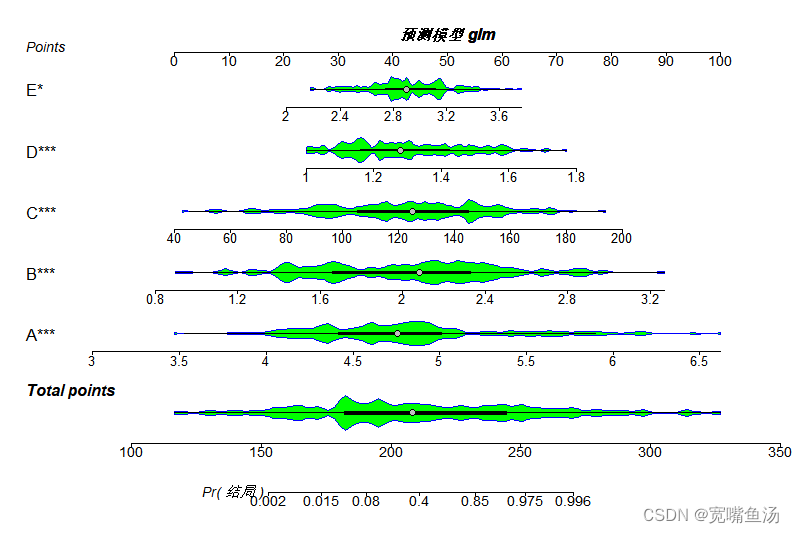 ~~~~~~~~~~~~11111111111111·````````````````~~~~~~~~~~~~~~~~~111111111111111111`·~~~~~~~
~~~~~~~~~~~~11111111111111·````````````````~~~~~~~~~~~~~~~~~111111111111111111`·~~~~~~~
有错误地方还请指出, 大家一起进步