文献翻译(BRAU-Net++: U-Shaped Hybrid CNN-Transformer Network for Medical Image Segmentation)
BRAU-Net++: U-Shaped Hybrid CNN-Transformer Network for Medical Image Segmentation
BRAU-Net:用于医学图像分割的U形混合CNN变换网络
Libin Lan, Member, IEEE, Pengzhou Cai, Lu Jiang, Xiaojuan Liu , Yongmei Li, and Yudong Zhang ,
Senior Member, IEEE
摘要
准确的医学图像分割对于临床量化、疾病诊断、治疗计划和许多其他应用至关重要。基于卷积和基于Transformer的u形结构在各种医学图像分割任务中都取得了显著的成功。前者可以有效地学习图像的局部信息,同时需要卷积运算所固有的更多特定于图像的归纳偏差。后者可以使用自注意在不同的特征尺度上有效地捕捉长程依赖性,而随着序列长度的增加,它通常会遇到二次计算和内存需求的挑战。为了解决这个问题,通过将这两种范式的优点集成到设计良好的u形架构中,我们提出了一种混合但有效的CNN-Transformer网络,称为BRAU-Net++,用于精确的医学图像分割任务。具体来说,BRAU-Net++使用双层路由注意力作为核心构建块来设计我们的u形编码器-编码器结构,其中编码器和解码器都是分层构建的,以便在降低计算复杂度的同时学习全局语义信息。此外,该网络通过引入采用卷积运算的通道空间注意力来重构跳跃连接,旨在最大限度地减少局部空间信息损失,并放大多尺度特征的全局维度交互。在三个公共基准数据集上的大量实验表明,我们提出的方法在几乎所有的评估指标下都优于其他最先进的方法,包括其基线:BRAU-Net。我们在Synapse多器官分割、ISIC-2018挑战和CVC ClinicDB上获得了82.47、90.10和92.94的平均骰子相似系数(DSC),在ISIC-2018-挑战和CVC-ClinicDB中分别获得了84.01和88.17的mIoU。这些代码将在GitHub上提供。
索引术语——BRAU-Net++,卷积神经网络,医学图像分割,稀疏注意力,Transformer。
1. 引言
精确和稳健的医学图像分割在计算机辅助诊断系统中发挥着重要作用,尤其是在图像引导的临床手术、疾病诊断、治疗计划和临床量化[1],[2],[3]中。医学图像分割通常被认为与自然图像分割基本相同[4],其相应的技术通常来源于后者[5]。这两个群体的共同点是,他们都以手动或全自动的方式提取图像的准确兴趣区域(ROI)作为研究目标。得益于深度学习技术,自然图像视觉中的分割任务取得了令人印象深刻的性能。但与自然图像分割不同,医学图像分割需要对ROI(如异常和器官)进行更准确的分割结果,以快速识别病变边界并准确评估病变程度。这是因为临床实践表明,医学图像中细微的分割错误会导致临床环境中用户体验不佳,并增加后续计算机辅助诊断的风险[6]。此外,在各种成像模式中手动描绘病变及其边界需要大量的工作,这是非常耗时甚至不切实际的,并且由此产生的分割可能会受到临床医生的偏好和专业知识的影响[7],[45]。因此,我们认为,开发智能和稳健的技术来有效准确地分割医学图像中的病变区域或器官是至关重要的。
根据深度学习的发展以及广泛而有前景的应用,已经提出了许多依赖于卷积运算的医学图像分割方法来分割医学图像中的特定目标对象。在这些方法中,像U-Net[8]和全卷积网络(FCN)[9]这样的u型编码器-解码器架构已经在医学图像分割中占据主导地位。后续的各种变体,如U-Net++[6]、U-Net3+[10]、Attention U-Net[11]、3D U-Net[12]和V-Net[13],也已被开发用于各种医学成像模式的图像和体积分割,并在心脏分割、多器官分割和息肉分割等广泛的医学应用中取得了突出成功。这些基于CNN的方法的优异性能证明了CNN具有较强的语义信息学习能力。但由于卷积运算的固有局部性,它在显式捕获长程依赖性方面往往表现出局限性。一些研究试图通过使用萎缩卷积层[14]、[15]、自注意机制[16]、[17]和图像金字塔[18]来解决这个问题。然而,这些方法并不能显著提高对长期依赖进行建模的能力。
最近,受Transformer在自然语言处理(NLP)[19]领域取得巨大成功的启发,许多研究试图将Transformer应用于视觉领域[20],[21],[22],[23]。这些工作在各种视觉任务上取得了持续的改进,这表明视觉Transformer在视觉领域具有巨大的潜力。在这些工作中,一个流行的话题是如何通过提高核心构建块(即注意力)来提高模型的性能。作为视觉Transformer的核心构建块,注意力是捕捉长期依赖关系的有力工具。然而,普通注意力是一种全注意力机制,它计算所有空间位置上的成对标记亲和力,因此它具有很高的计算复杂性,并导致大量内存占用[24]。为了缓解这个问题,一些工作试图将稀疏注意力应用于视觉Transformer,其中每个query令牌只关注部分key和value令牌,而不是整个序列[25]。为此,已经探索了几种手工制作的稀疏图案,例如在局部窗口[23]、扩张窗口[26]、[27]或轴向条纹[28]中限制注意力。在医学图像视觉领域,许多研究也将Transformer引入医学图像分割任务,如nnFormer[29]、UTNet[30]、TransUNet[1]、TransCeption[3]、HiFormer[32]、FocalUNet[33]和MISFormer[34]。然而,据我们所知,很少有作品考虑将稀疏性思想引入该领域,其中代表性的作品涉及SwinUnet[35]和Gated Axial UNet(MedT)[36]。但这些稀疏注意力机制以手工制作的方式合并或选择稀疏模式。因此,这些模式是查询不可知的。也就是说,它们由所有查询共享。将动态稀疏性和查询感知稀疏性应用于医学图像分割在很大程度上仍未被探索。
上述这些问题促使我们探索一种全自动的高级分割算法,该算法能够根据医学图像的性质产生有效的分割结果,从而有利于更多的图像引导医学应用。最近,受BiFormer[24]成功将稀疏注意力应用于视觉Transformer[37]的启发,我们提出了BRAU-Net++,以利用Transformer的力量进行医学图像分割。据我们所知,BRAU-Net++是第一个考虑将动态稀疏注意力纳入CNN-Transformer架构的混合模型。BRAU-Net++也是在BRAU-Net[38]的基础上发展而来的,它使用BiFormer块构建了一个具有跳跃连接的u形纯Transformer网络结构,用于耻骨联合胎头分割。与Swin-Unet[35]和BRAU-Net[38]类似,网络结构的主要组件包括编码器、瓶颈、解码器和跳过连接。编码器、瓶颈和解码器都是基于BiFormer[24]的核心构建块构建的:双层路由注意力,它有效地建模了长程依赖关系,并节省了计算和内存。同时,在全局注意力机制[39]的激励下,我们通过引入通道空间注意力来重新设计跳跃连接,通道空间注意力通过卷积运算来执行,旨在最大限度地减少局部空间信息损失,并放大多尺度特征的全局维度交互。此外,与[24]、[26]、[40]、[41]类似,所提出的体系结构利用深度卷积来隐式编码位置信息。在三个公开可用的医学图像数据集:Synapse多器官分割[56]、ISIC2018 Challenge[42]、[43]和CVC ClinicDB[44]上进行的大量实验表明,所提出的方法具有良好的性能和鲁棒的泛化能力。
我们的主要贡献可以总结如下:
1)我们介绍了一种u形混合CNN-Transformer网络,该网络以双层路由注意力为核心构建块来设计编码器-解码器结构,其中编码器和解码器都是分层构建的,以便在降低计算复杂度的同时有效地学习局部全局语义信息。
2) 我们利用通道空间注意机制重新设计了传统的跳跃连接,并提出了具有通道空间注意的跳跃连接(SCCSA),旨在增强通道和空间两个方面的跨维度交互,补偿下采样造成的空间信息损失。
3) 我们在三个常用的数据集上验证了BRAU-Net++的有效性:Synapse多器官分割、ISIC-2018挑战和CVC ClinicDB数据集。因此,在几乎所有的评估指标下,所提出的BRAUNet都比其他最先进的(SOTA)方法表现出更好的性能。
本文的其余部分组织如下。第二节审查先前的相关工作。第三节规定了我们的方法、主要组成部分和培训程序。第四节介绍了我们的实验设置。第五节报告了实验细节和结果。第六节对实验结果和发现进行了一些讨论和说明,最后,第七节给出了我们的结论
2. 相关工作
2.1 U型架构
-
用于医学图像分割的基于CNN的U形结构:该范式的主要技术涉及UNet[8]和FCN[9],以及随后的变体[6]、[10]、[11]、[12]、[13],其中一些变体分别被引入2D或3D医学图像分割社区。由于U形结构的简单性和优越的性能,在2D医学图像分割领域,各种类似Unet的方法,如U-Net++[6]、UNet3+[10]和DCSAU-Net[46]不断涌现。其他方法也被引入到3D医学图像分割领域,如3D Unet[12]和V-Net[13]。这种方法采用了一系列卷积池操作来设计编码器和解码器。由于其强大的表示能力,这种范式在广泛的医学应用中取得了巨大的成功。关于更多关于U-Net及其变体应用于医学图像分割的工作,读者可以参考相关综述文献[47],[48]。
-
基于Transformer的U形医学图像分割体系结构:最初的Transformer架构最初是为机器翻译任务[19]提出的,并已成为自然语言处理(NLP)问题的事实标准。后续工作对Transformer应用于计算机视觉进行了更多尝试。最近,研究人员试图开发纯Transformer或混合Transformer来执行医学图像分割。在[35]中,提出了一种用于医学图像分割的纯Transformer,即Swin-Unet,其中将来自原始图像而不是CNN特征图的标记化块输入到局部全局语义特征学习的架构中。在[1]中,CNN-Transformer混合模型TransUNet利用来自CNN特征的详细高分辨率空间信息和由Transformer编码的全局上下文来实现卓越的分割性能。与TransUNet类似,UNETR[49]和Swin UNETR[50]在编码器中使用了Transformer,并使用卷积解码器来生成分割图。这些工作使用全注意力或静态稀疏注意力来计算成对的令牌亲和性。与这些方法不同的是,我们引入了动态稀疏的注意力来选择最相关的令牌,并且网络的输入是来自原始图像的令牌化补丁。因此,信息不会由于较低的分辨率而丢失。同时,我们将卷积运算应用于跳跃连接,以增强多尺度特征的全局维度交互。
稀疏注意力机制
稀疏连接模式[37]已被引入,以解决普通注意力机制的计算和内存复杂性。稀疏注意力在视觉Transformer中获得了更多的吸引力[23],[25],[26],[27],[28]。在Swin Transformer[23]中,注意力被限制在不重叠的局部窗口上,并引入移位窗口操作以促进相邻窗口之间的窗口间通信。因此,这种关注是基于本地窗口的手工制作的。随后的研究还引入了各种手动设计的稀疏模式,如扩张窗口[26]、[27]或十字形窗口[31]。近年来,基于动态标记稀疏性的高效视觉变换器取得了巨大成功。在[51]中,通过分层修剪动态选择要传递到下一层的令牌数量来实现推理的加速。在[25]、[24]中,他们分别提出了四叉树注意力和双层路由注意力,以从粗到细的方式实现查询自适应稀疏性。不同之处在于,双层路由注意力旨在定位几个最相关的键值对,而四叉树注意力构建了一个令牌金字塔,并组装来自不同粒度级别的信息。在这项工作中,我们试图使用BiFormer块作为基本单元,构建一个带有SCCSA模块的u形编码器架构,用于医学图像分割。
通道空间注意力
计算机视觉中的注意机制研究取得了很大进展,其中通道注意和空间注意是两个重要方向。通道关注的焦点是CNN通道的信息。例如,SENet[52]自适应地重新校准信道特征响应,以增强网络的判别能力。另一方面,空间注意力集中在相关的空间区域。例如,STN[53]可以转换空间中的各种变形数据,并自动捕捉重要的区域特征。在这些个体成功的基础上,CBAM[54]以串联的方式将通道注意力和空间注意力结合起来,共同捕捉通道和空间位置之间的复杂依赖关系。受全局注意力机制[39]的启发,我们使用通道空间注意力重新设计跳跃连接,以增强通道空间维度的互动性,并补偿由于下采样而造成的空间信息损失。
3. 方法
在本节中,我们首先简要总结一下双层路由注意(BRA)。然后,我们描述了所提出的BRAU-Net++的总体架构。最后,我们介绍了BiFormer块和跳过连接信道空间注意模块(SCCSA)。
3.1 准备工作:双层路由注意
双层路由注意力(BRA)是一种动态的、可查询的稀疏注意力机制,其核心思想是在粗粒度的区域级别过滤掉语义上最不相关的键值对,只保留一小部分最相关的路由区域到细粒度的令牌对令牌的注意力。与其他手工制作的静态稀疏注意力机制[23]、[31]、[55]相比,BRA容易对长程依赖性进行建模。在这一点上,它类似于香草般的关注。但BRA的复杂性为O((HW)4/3),而普通注意力的复杂性为0((HW)2)[24]。
- 区域划分和线性投影:通过将二维输入特征图X∈RH×W×C划分为S×S个非重叠区域,
可以获得每个区域的特征维数HW / S2。随后,基于所得到的特征图Xr∈R(S2 ×HW/S2×C),可以通过线性投影导出查询、关键字、值Q、K、V∈R(S2×HW/S2×C):

其中Wq,Wk,Wv∈R(C×C)分别是查询、键、值的线性投影权重矩阵 - 区域到区域路由:该过程首先分别计算每个区域的Q和K的平均值,产生区域级查询和密钥,Qr,Kr∈R(S2×C)。接下来,通过在Qr和转置Kr之间应用矩阵乘法,导出区域到区域邻接矩阵Ar∈R(S2×S2)。最后,关键步骤是通过路由索引矩阵Ir∈N(S2×k),使用逐行top-k算子topkIndex(),仅保留每个查询区域的前k个最相关区域。区域间路由可以公式化为:
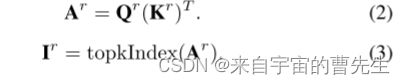
- 代币对代币的关注:由于路由区域可能在空间上分散在整个特征图上,因此需要收集路由区域中的密钥和值张量。然后在这些键值张量上应用细粒度的标记到标记注意力。该过程如图1所示,其公式如下:

其中Kg,Vg∈R(kHW×C)是集键值张量。使用深度卷积对函数LCE(·)进行参数化。
3.2 体系结构概述
BRAU-Net的总体架构如图2(a)所示。BRAU-Net++包括编码器、解码器、瓶颈和SCCSA模块。对于编码器,给定大小为H×W×3的输入医学图像,将医学图像分割为重叠的块,并通过块嵌入将每个块的特征维数投影到任意维数(定义为C)。变换后的补丁令牌通过多个BiFormer块和补丁合并层来生成层次特征表示。具体来说,补丁合并用于降低特征图的分辨率和增加维度,而BiFormer块用于学习特征表示。对于瓶颈,特征图的分辨率和维度保持不变。受U-Net[8]和Swin-Unet[35]的启发,我们设计了一种基于对称变换器的解码器,该解码器由BiFormer块和补丁扩展层组成。补片扩展层负责上采样和降维。通过SCCSA模块将提取的上下文特征与编码器的多尺度特征融合,以弥补下采样造成的空间信息损失,并放大全局维度的相互作用。最后一个补丁扩展层用于4×上采样,以恢复特征图的原始分辨率H×W,然后使用线性投影层生成像素级分割预测。我们将在下面详细说明每一块。

3.3 BiFormer块

构建块的核心是双层路由注意力(BRA)。如图3,BiFormer块由一开始的3×3深度卷积、2个LayerNorm(LN)层、一个BRA模块、3个残差连接和一个扩展比为e=3的2层MLP组成。3×3深度卷积可以隐式地对相对位置信息进行编码。BiFormer块可以公式化为:

其中,zl−1、zl和zl分别表示第l个块的深度卷积、BRA模块和MLP模块的输出。
3.4 编码器
编码器是通过使用三级金字塔结构来分层构造的。具体而言,在阶段1中,由两个3×3卷积层组成的补丁嵌入层,在阶段1-3中,使用具有3×3卷积层的补丁合并层来降低输入空间分辨率,同时增加通道数量。如图2,在阶段1中,将分辨率为H4×W4和C通道的标记化输入馈送到两个连续的BiFormer块中,以执行表示学习。阶段2-3中的标记化输入也以类似的方式执行。补丁合并层执行2×下采样以将标记数量减少一半,并将特征维数增加2×。
3.5 解码器
与编码器类似,解码器也是基于BiFormer块构建的。受Swin Unet[35]的启发,我们还采用了补丁扩展层对解码器中提取的深层特征进行上采样。补丁扩展层主要用于将特征图重塑为更高分辨率的特征图,即将分辨率提高2×,并将特征维数降低一半。最后一个补丁扩展层执行4×上采样以输出分辨率为H×W的特征图,用于预测像素级分割。
3.6 跳跃连接
与使用单一注意力机制相比,通道和空间注意力的结合可以增强模型捕捉更广泛上下文特征的能力。受[39]的启发,我们考虑将顺序通道空间注意力机制应用于跳跃连接,从而提出了一种跳跃连接通道空间注意力,简称SCCSA。SCCSA模块可以有效补偿下采样造成的空间信息损失,增强解码器每层多尺度特征的全局维度交互,从而在生成输出掩码的同时恢复细粒度细节。如图2(b)所示,SCCSA模块包括通道注意力子模块和空间注意力子模块。具体地说,我们首先通过级联编码器和解码器的输出,导出F1∈R(h×w×2n)。然后,通道注意力子模块使用多层感知器(MLP)的编码器-解码器结构放大跨维度通道空间相关性,其缩减率为e=4。我们使用两个7×7卷积层来关注来自通道注意力子模块的具有相同缩减率e的空间信息。给定输入特征图x1,x2∈Rh×w×n,中间状态F1,F2,F3,然后输出x3定义为:

其中F2和F3分别是通道和空间注意力子模块的输出;⊗和σ分别表示元素乘法和sigmoid激活函数。
3.7 损失函数
在训练过程中,对于Synapse数据集,我们使用了一种混合损失,它结合了dice损失和交叉熵损失来解决与类不平衡相关的问题。对于ISIC2018和CVC ClinicDB数据集,我们使用dice损失来优化我们的模型。骰子损失(Ldice)、交叉熵损失(Lce)和混合损失(L)定义如下:
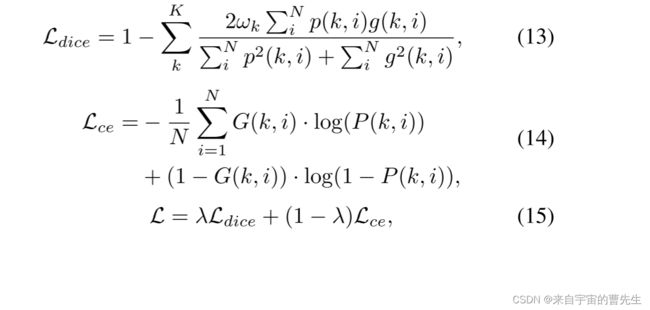
其中N是像素数,G(k,i)∈(0,1)和P(k,i)∈。K是类的个数,P KωK=1是所有类的权和。λ是一个加权因子,用于平衡Ldice和Lce的影响。在我们的研究中,ωk和λ分别根据经验设定为1K和0.6。算法1总结了我们的BRAU网络的训练过程。

4. 实验环境
4.1 数据集
我们在三个公开可用的医学图像分割数据集上训练和测试所提出的BRAU-Net++:Synapse多器官分割[56]、ISIC-2018挑战[42]、[43]和CVC ClinicDB[44]。关于数据分割的详细信息如表I所示。所有数据集都与临床诊断有关,因此它们的分割结果对患者的治疗至关重要,并且由图像及其相应的地面实况掩码组成。选择不同成像模态数据集的主要原因是评估所提出方法的性能和稳健性。
- Synapse多器官分割数据集:腹部计算机断层扫描(CT)上的自动多器官分割可以支持临床诊断、治疗计划和治疗提供工作流程。实验中使用的数据集包括MICCAI 2015多图谱腹部标记挑战赛的30次腹部CT扫描,以及3779张轴向腹部临床CT图像。每个CT体积包括85-198个512×512像素的切片,体素空间分辨率为([0.54–0.54]×[0.98–0.98]×[2.5–5.0])mm3。在[1]、[35]之后,训练集和测试集分别由18个(包含2212个轴向切片)和12个样本组成。
- ISIC-2018挑战数据集:本工作中的数据集是指用于ISIC-2018挑战中病变分割任务的训练集,其中包含2594张带有基本事实分割注释的皮肤镜图像。进行五次交叉验证以评估模型的性能,并选择最佳模型进行推理。
- CVC ClinicDB数据集:CVC ClinidDB数据集通常用于息肉分割任务。它也是MICCAI 2015关于自动息肉检测挑战赛的子挑战赛的训练数据集。该数据集包含612幅图像,随机分为490幅训练图像、61幅验证图像和61幅测试图像。

4.2 评估指标
为了评估所提出的BRAU-Net++的性能,将平均骰子相似系数(DSC)和平均豪斯多夫距离(HD)作为评估指标来评估我们的方法对8个腹部器官的评估:主动脉、胆囊、脾脏、左肾、右肾、肝脏、胰腺、脾脏和胃,并且只有DSC专门用于单个器官的评估。此外,平均并集交集(mIoU)、DSC、准确性、精密度和召回率等被视为ISIC-2018 Challenge和CVC ClinicDB数据集上模型性能的评估指标。形式上,预测可以分为真阳性(TP)、假阳性(FP)、真阴性(TN)和假阴性(FN),然后DSC、IoU、准确性、精密度和召回率计算如下:

HD可以描述为:

其中Y和Yõ分别是地面实况掩码和预测分割图。d(y,ntr y)表示点y和ntr y之间的欧几里得距离。
4.3 实现细节
我们在具有24GB内存的NVIDIA 3090图形卡上训练我们的BRAU-Net++模型及其各种消融变体。我们使用Python 3.10和PyTorch 2.0[57]来实现我们的方法。在训练过程中,我们在上述三个数据集上初始化并微调模型,使用BiFormer[24]在ImageNet-1K[58]上预训练的权重,并考虑到空间,还仅在Synapse多器官分割数据集上从头开始训练所提出的模型。在这些结果模型上,我们进行了一系列消融研究,以分析每个组件的贡献。
关于Synapse多器官分割数据集,我们将所有图像的大小调整为224×224的分辨率,并使用随机梯度下降对400个时期的模型进行训练,批量大小为24,学习率为0.05,动量为0.9,权重衰减为1e-4。关于ISIC-2018 Challenge和CVC ClinicDB数据集,我们将所有图像的大小调整为256×256的分辨率,并使用Adam[59]优化器对200个时期的所有模型进行训练,批量大小为16。我们采用CosineAnnealingLR时间表,初始学习率为5e-4。使用诸如水平翻转、垂直翻转、旋转和概率为0.25的剪切之类的数据增强来增强数据多样性。
其他超参数也是经验设置的。例如,根据224×224和256×256的分辨率,区域分割因子S分别设置为7和8。从阶段1到阶段7的top-k的数量分别设置为2、4、8、S2、8、4和2,其中S2表示使用完全注意力。
5. 实验结果
在本节中,我们详细介绍了在Synapse多器官分割、ISIC-2018 Challenge和CVC ClinicDB数据集上,所提出的BRAU-Net与其他最先进的(SOTA)方法的比较,包括基于CNN、基于Transformer和两者的混合方法。此外,我们以Synapse多器官分割数据集为例,在此基础上进行了广泛的消融研究,以分析我们方法的每个组成部分的效果。
5.1 Synapse多器官分割方法的比较
如上所述,自动多器官腹部CT分割在提高包括疾病诊断、预后分析和治疗计划在内的临床工作流程的效率方面发挥着重要作用。因此,我们选择这个数据集来评估各种方法的性能。在Synapse多器官腹部CT分割数据集上,我们的建议与以前的SOTA方法在DSC和HD方面的比较如表II所示,最佳结果以粗体显示。[32]、[60]、[33]、[34]的结果是根据公开发布的代码在我们的实验环境下复制的,而其他结果直接来自各自发表的论文。我们的BRAU-Net在这两个评估指标上都大大优于基于CNN的方法和我们的基线:BRAU-Net,这表明更深层次的混合CNN-Transformer模型可能能够对全局关系和局部表示进行建模。与两种主流的基于变压器的方法:TransUNet和Swin-Unet相比,我们的BRAU-Net在DSC上分别显著增加了4.49%和3.34%,在HD上分别显著减少了12.62mm和2.48mm。这表明使用双层路由注意力作为核心构建块来设计u形编码器-解码器结构可能有助于有效地学习全局语义信息。更具体地说,BRAU-Net在大多数器官的分割方面,特别是在左肾和肝的分割方面稳定地优于其他方法。从表II可以看出,我们的方法获得的DSC值最高,高达82.47%,这表明我们的方法预测的分割图比其他方法与地面实况掩模有更高的重叠。还可以观察到,与HiFormer和MISSFormer相比,我们在HD上获得了相对较低的值(19.07mm),这分别产生了最佳(14.7mm)和次佳(18.20mm)的结果。BRAU-Net在HD上仅比MISSFormer提高了0.87mm,但明显比HiFormer提高了4.37mm,这表明我们的方法学习目标边缘信息的能力可能不如HiFormer。总的来说,表II显示,除了HiFormer和MISSFormer之外,所提出的BRAUNet比之前的工作有显著的改进,例如,DSC的性能增益分别从0.51%到12.2%,HD的性能增益从1.59mm到20.63mm。因此,我们相信我们的方法仍然有可能获得相对更好的分割结果。

5.2 ISIC-2018挑战比较
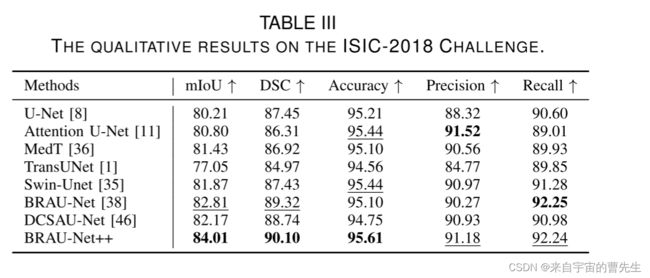
众所周知,黑色素瘤是一种常见的癌症,如果及时发现并治疗,可以挽救高达99%的生命。因此,皮肤病变的自动化诊断工具对准确检测黑色素瘤非常有帮助。我们对ISIC-2018 Challenge数据集进行了五倍交叉验证,以评估我们的方法避免过度拟合的性能。我们根据公开发布的代码复制所有方法的结果。定量和定性结果如表III和图5(左)所示。我们的方法获得了84.01的mIoU、90.10的DSC、95.61的准确度、91.18的精密度和92.24的召回率,其中我们的方法在mIoU,DSC和准确度方面获得了最好的性能,在精密度和召回率方面获得了第二好的结果。可以观察到,与最近发布的DCSAU-Net和BRAU-Net相比,所提出的BRAU-Net在mIoU上分别获得1.84%和1.2%的改进。此外,我们的方法实现了0.9224的召回率,这在临床应用中更有利。从以上分析和图5(左)可以明显看出,在ISIC-2018 Challenge数据集上,BRAU-Net比其他方法实现了更好的边界分割预测。BRAUNet的分割掩模的轮廓更接近地面实况。
5.3 CVC ClinicDB的比较
在息肉有可能转变为结直肠癌癌症之前,早期发现可以提高存活率。这对临床实践具有重要意义。因此,我们在实验中选择了这个数据集。定量结果如表IV所示。我们提出的方法在mIoU(88.17)、DSC(92.94)、Precision(93.84)和Recall(93.06)上获得了最好的结果,分别超过第二好的1.99%、1.27%、2.12%和1.03%。定性结果如图5(右)所示。可以看出,我们的方法生成的息肉掩码与地面实况的边界和形状非常匹配。
5.4 消融研究
在本节中,我们对上述三个数据集进行了广泛的消融研究,以彻底评估BRAU-Net++中涉及的每个组件的有效性。具体来说,我们消除了SCCSA模块、跳过连接的数量和top-k、输入大小和分割因子S以及模型规模和预训练权重的影响。
-
SCCSA模块的有效性:SCCSA模块是拟议的BRAU网络的重要组成部分。它使用通道空间注意力来增强通道和空间方面的跨维度交互,并有助于生成更准确的分割掩码。表II显示了Synapse上不带和带SCCSA模块的BRAU-Net的结果。与没有SCCSA的BRAU-Net相比,BRAU-Net++获得了更好的分割性能,在DSC上分别提高了0.91%,在HD评估指标上分别降低了0.39mm。这样的微小改进是有代价的:它为这个模型带来了大量的参数。一个主要原因可能是,多尺度CNN特征与通过层次变换器结构学习的全局语义特征的组合不能显著有利于分割任务。关于具体原因,我们打算将其作为未来的工作来进一步探索和分析。ISIC-2018 Challenge和CVC ClinicDB数据集的分割结果如表五所示。可以看出,在BRAU-Net模型中添加SCCSA模块几乎可以在所有评估指标下获得最佳结果。例如,短链氯化石蜡可帮助ISIC-2018挑战改善0.6%,CVC ClinicDB w.r.t.mIoU指标改善0.9%。此外,还计算了参数的数量、浮点运算(FLOP)和每秒帧数(FPS),以进一步研究该模块的有效性。我们可以观察到,短链氯化石蜡在两个数据集上对FPS没有显著损害,尤其是CVC ClinicDB。

-
跳接次数的有效性:已经证明,u形网络的跳跃连接可以通过恢复低级别的空间信息来帮助提高更精细的分割细节。本次消融主要旨在探讨不同数量的跳跃连接对BRAU网络性能提升的影响。本实验是在Synapse数据集上进行的。在1/4、1/8和1/16分辨率范围的位置添加跳过连接,通过不同位置的连接组合,可以将跳过连接的数量更改为0、1、2和3,其中“0”表示不添加跳过连接。其他添加的连接及其在平均DSC和HD度量上的相应分割性能如表VI所示。我们可以观察到,随着跳过连接数量的增加,分割性能逐渐提高,通过在1/4、1/8和1/16分辨率标度的所有位置添加跳过连接,可以获得最佳的平均DSC和高清。因此,我们在BRAU Net中采用了这种配置,以增强学习精确低级别细节的能力。这可能是BRAU-Net能够捕捉小目标特征的主要原因。

-
输入分辨率和划分因子S的有效性:进行此消融的主要目标是测试输入分辨率对模型性能的影响。我们在Synapse数据集上以128×128、224×224和256×256的分辨率进行了三组实验,并将结果报告在表VII中。根据[24],选择分割因子S作为每个阶段特征图大小的除数,以避免填充,并且具有不同输入分辨率的图像应该采用不同的分割因子S。因此,我们将上述三种分辨率的对应分割因子设置为S=4、S=7和S=8。可以看出,保持补丁大小相同(例如,32)并逐渐增加分辨率尺度,即增加令牌的序列长度,可以导致模型性能的持续提高。这符合常识,即较大分辨率的图像包含更多的语义信息,从而提高了性能。然而,这是以更大的计算成本为代价的。因此,考虑到计算成本,为了公平地与其他方法进行比较,所有实验都是基于224×224的默认分辨率作为输入进行的。

-
Top-k数量的有效性:与[24]类似,随着路由区域的大小在下一阶段逐渐减小,我们相应地增加了k,以保持合理数量的令牌。Synapse数据集上top-k数量的消融结果如表VIII所示,其中列出了网络每个阶段要参与的top-k和令牌的数量。可以看出,增加编码器近顶部阶段的令牌数量似乎可以提高分割性能。这可能是因为网络的近顶部块可以捕获低级别信息,例如边缘或纹理,这对于分割任务是必不可少的。此外,盲目增加关注标记的数量可能会损害性能,这表明显式稀疏性约束可以作为正则化来提高模型的泛化能力。这一见解与[24]类似。

-
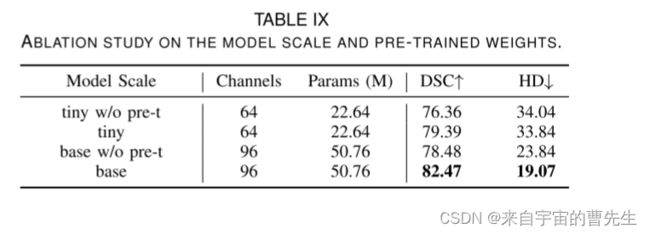
模型量表和预训练权重的有效性:与[1]、[35]类似,我们给出了网络深化的效果。此外,众所周知,基于Transformer的模型的性能受到模型预训练的严重影响。因此,我们考虑分别从从头开始训练的模型和预训练的方面,在BRAU-Net的两个不同模型尺度上提供四项消融研究。BRAU-Net++的两种不同的模型尺度分别称为微小模型和基本模型。表IX列出了他们在Synapse数据集上的配置和结果。可以看出,基本模型产生了更有利的结果。特别是在HD评估指标方面,与微小模型相比,基本模型的结果提高了14.77mm。这表明基础模型可以实现更好的边缘预测。因此,我们采用基本模型来进行医学图像分割。考虑到计算性能,我们对所有实验都采用了“基本”模型。
6. 讨论
在这项工作中,我们证明了动态和查询感知的稀疏注意力在降低计算复杂度和提高模型性能方面是有效的。为了进一步说明稀疏注意力如何在医学图像分割任务中发挥作用,在[24]之后,我们可视化路由区域和注意力响应w.r.t.查询令牌。我们采用了从编码器中第三阶段的最后一个块中提取的路由索引和注意力得分来进行可视化。也就是说,这些值是从H 16×W 16分辨率的特征图中获得的,而可视化是在原始分辨率的图像中呈现的。Synapse多器官分割、ISIC-2018 Challenge和CVC ClinicDB数据集的结果如图所示。6。可以清楚地看到,稀疏注意力类型可以有效地找到语义上最相关的区域,这表明动态稀疏注意力计算机制对于医学图像的稀疏模式的计算和选择是有效的。然而,探索其他有效的稀疏模式计算方法仍然是必要的,也是我们未来工作的重点。
我们进行了一系列消融研究,以评估BRAU-Net++的每个相关组件的贡献,其中我们提出了SCCSA模块,以增强编码器中第i阶段和解码器中第7-i阶段的这些特征在信道和空间方面的跨维交互。在几乎所有的评估指标下,实验结果都令人鼓舞。然而,从表II中可以看出,这种微小的改进是以带来大量参数为代价的。这是我们工作的一个缺点。我们认为,主要原因可能是通过层次变换器结构学习的多尺度CNN特征和全局语义特征的组合不能显著有利于分割任务。在今后的工作中,我们将重点关注如何有效解决这一问题。
有意选择三种不同的成像模式数据集:Synapse多器官分割、ISIC-2018挑战和CVCClinicDB作为基准。做出这一选择的主要原因是为了评估所提出方法的性能和稳健性。大量实验揭示了我们的方法在多模态医学图像分割任务中的通用性。
7. 总结
在本文中,我们提出了一种精心设计的u形混合CNN转换器架构BRAU-Net++,它利用动态稀疏注意力而不是全注意力或静态手工制作的稀疏注意力,可以有效地学习局部全局语义信息,同时降低计算复杂度。此外,我们提出了一个新的模块:跳跃连接通道空间注意力(SCCSA)来整合多尺度特征,以补偿空间信息的损失,增强跨维度的交互作用。实验结果表明,在Synapse多器官分割、ISIC-2018 Challenge和CVCClinicDB数据集的几乎所有评估指标下,我们的方法都能实现SOTA性能,尤其擅长捕捉小目标的特征。对于未来的工作,我们将专注于如何为多模态医学图像分割任务设计更复杂和通用的架构。
8. 引用
[1] J. Chen, Y. Lu, Q. Yu, X. Luo, E. Adeli, Y. Wang, L. Lu, A. L. Yuille, and
Y. Zhou, “TransUNet: Transformers make strong Encoders for medical
image segmentation,” arXiv:2102.04306, 2021.
[2] A. Srivastava et al., “MSRF-Net: A multi-scale residual fusion network
for biomedical image segmentation,” IEEE J. Biomed. Health. Inf. , vol.
26, no. 5, pp. 2252–2263, May 2022.
[3] R. Azad, Y. Jia, E. K. Aghdam, J. Cohen-Adad, and D. Merhof,
“Enhancing medical image segmentation with TransCeption: A multi-
scale feature fusion approach,” arXiv:2102.04306, 2023.
[4] J. Li, M. Erdt, F. Janoos, T. Chang, and Jan Egger, “Medical image
segmentation in oral-maxillofacial surgery,” in Computer-Aided Oral and
Maxillofacial Surgery, J. Egger and X. Chen, Ed. Academic Press, 2021,
pp. 1–27.
[5] A. S. Ashour, Y. Guo, and W. S. Mohamed, “Image-guided thermal
ablation therapy,” in Thermal Ablation Therapy, A. S. Ashour, Y. Guo,
and W. S. Mohamed, Ed. Academic Press, 2021, pp. 411–440.
[6] Z. Zhou et al., “UNet++: A nested U-Net architecture for medical image
segmentation,” in Deep Learn. Med. Imag. Anal. Multimodal Learn.
Clin. Decis. Support, 2018, pp. 3–11.
[7] X. Chen et al., “Learning Active Contour Models for Medical Image
Segmentation,” in Proc. IEEE Conf. Comput. Vis. Pattern Recog., Long
Beach, CA, USA, 2019, pp. 11624–11632.
[8] O. Ronneberger, P. Fischer, and T. Brox, “U-Net: Convolutional net-
works for biomedical image segmentation,” in Proc. Int. Conf. Med.
Imag. Comput. Comput.-Assist. Interv., 2015, pp. 234–241.
[9] J. Long, E. Shelhamer, and T. Darrell, “Fully convolutional networks
for semantic segmentation,” in Proc. IEEE Conf. Comput. Vis. Pattern
Recognit., Boston, MA, USA, 2015, pp. 3431–3440.
[10] H. Huang et al., “UNet 3+: A full-scale connected unet for medical
image segmentation,” in Proc. IEEE Int. Conf. Acoust. Speech Signal
Process., Barcelona, Spain, 2020, pp. 1055–1059.
[11] O. Oktay et al., “Attention U-Net: Learning where to look for the
pancreas,” arXiv:1804.03999, 2018.
[12]
O.¨
C¸ic¸ek et al., “3D U-Net: Learning dense volumetric segmentation
from sparse annotation,” in Proc. Int. Conf. Med. Imag. Comput.
Comput.-Assist. Interv., 2016, pp. 424–432.
[13] F. Milletari, N. Navab, and S. -A. Ahmadi, “V-Net: Fully convolutional
neural networks for volumetric medical image segmentation,” in Proc.
IEEE 4th Int. Conf. 3D Vis., Stanford, CA, USA, 2016, pp. 565–571.
[14] L. -C. Chen, G. Papandreou, I. Kokkinos, K. Murphy, and A. L. Yuille,
“DeepLab: Semantic image segmentation with deep convolutional nets,
atrous convolution, and fully connected CRFs,” EEE Trans. Pattern Anal.
Mach. Intell., vol. 40, no. 4, pp. 834–848, Apr. 2018.
[15] Z. Gu et al., “Context encoder network for 2D medical image segmen-
tation,” IEEE Trans. Med. Imag., vol. 38, no. 10, pp. 2281–2292, Oct.
2019.
[16] X. Wang, R. Girshick, A. Gupta, and K. He, “Non-local neural net-
works,” in Proc. IEEE Conf. Comput. Vis. Pattern Recognit., Salt Lake
City, UT, USA, 2018, pp. 7794–7803.
[17] J. Schlemper et al., “Attention gated networks: Learning to leverage
salient regions in medical images,” Med. Image Anal., vol. 53, pp. 197–
207, Apr. 2019.
[18] H. Zhao, J. Shi, X. Qi, X. Wang, and J. Jia, “Pyramid scene parsing
network,” in Proc. IEEE Conf. Comput. Vis. Pattern Recognit., Honolulu,
HI, USA, 2017, pp. 6230–6239.
[19] A. Vaswani, N. Shazeer, N. Parmar, J. Uszkoreit, L. Jones, A. N. Gomez,
L. Kaiser, and I. Polosukhin, “Attention is all you need,” in Proc. Adv.
Neural Inf. Process. Syst., 2017, pp.5998–6008.
[20] N. Carion, F. Massa, G. Synnaeve, N. Usunier, A. Kirillov, and S.
Zagoruyko, “End-to-end object detection with transformers,” in Proc.
Eur. Conf. Comput. Vis., 2020, pp. 213–229.
[21] A. Dosovitskiy et al., “An image is worth 16x16 words: Transformers for
image recognition at scale,” in Proc. Int. Conf. Learn. Representations,
2021.
[22] H. Touvron et al., “Training data-efficient image transformers & distil-
lation through attention,” Proc. Mach. Learn. Res., vol. 139, pp. 10347–
10357, Jul. 2021.
[23] Z. Liu et al., “Swin Transformer: Hierarchical vision transformer using
shifted windows,” in Proc. IEEE Int. Conf. Comput. Vis., Montreal, QC,
Canada, 2021, pp. 9992–10002.
[24] L. Zhu, X. Wang, Z. Ke, W. Zhang, and R. Lau, “Biformer: Vision trans-
former with bi-level routing attention,” in Proc. IEEE Conf. Comput. Vis.
Pattern Recognit., Vancouver, BC, Canada, 2023, pp. 10323–10333.
[25] S. Tang, J. Zhang, S, Zhu, and P. Tan, “Quadtree attention for vision
transformers,” in Proc. Int. Conf. Learn. Representations, 2023.
[26] Z. Tu, H. Talebi, H. Zhang, F. Yang, P. Milanfar, A. Bovik, and Y. Li,
“MaxViT: Multi-axis vision transformer,” in Proc. Eur. Conf. Comput.
Vis., 2022, pp. 459–479.
[27] W. Wang, L. Yao, L. Chen, B. Lin, D. Cai, X. He, and W. Liu,
“CrossFormer: A versatile vision transformer hinging on cross-scale
attention,” in Proc. Int. Conf. Learn. Representations, 2022.
[28] H. Wang, Y. Zhu, B. Green, H. Adam, A. Yuille, and L. Chen, “Axial-
DeepLab: Stand-alone axial-attention for panoptic segmentation,” in
Proc. Eur. Conf. Comput. Vis., 2020, pp. 108–126.
[29] H. -Y. Zhou, J. Guo, Y. Zhang, X. Han, L. Yu, L. Wang, and Y.
Yu, “nnFormer: Volumetric medical image segmentation via a 3D
transformer,” IEEE Trans. Image Process., vol. 32, pp. 4036–4045, 2023.
[30] Y. Gao, M. Zhou, and D. N. Metaxas, “UTNet: A hybrid transformer
architecture for medical image segmentation,” in Proc. Int. Conf. Med.
Imag. Comput. Comput.-Assist. Interv., 2021, pp. 61–71.
[31] X. Dong et al., “CSWin Transformer: A general vision transformer
backbone with cross-shaped windows,” in Proc. IEEE Conf. Comput.
Vis. Pattern Recognit., New Orleans, LA, USA, 2022, pp. 12114–12124.
[32] M. Heidari et al., “HiFormer: Hierarchical multi-scale representations
using transformers for medical image segmentation,” in Proc. IEEE
Winter Conf. Appl. Comput. Vis., Waikoloa, HI, USA, 2023, pp. 6191–
6201.
[33] M. Naderi, M. Givkashi, F. Piri, N. Karimi, and S. Samavi, “Focal-
UNet: UNet-like focal modulation for medical image segmentation,”
arXiv:2212.09263, 2022.
[34] X. Huang, Z. Deng, D. Li, and X. Yuan, “MISSFormer: An effective
medical image segmentation transformer,” arXiv:2109.07162, 2021.
[35] H. Cao, Y. Wang, J. Chen, D. Jiang, X. Zhang, Q.Tian, and M. Wang,
“Swin-Unet: Unet-like pure transformer for medical image segmenta-
tion,” in Proc. Eur. Conf. Comput. Vis., 2022, pp. 205–218.
[36] J. M. J. Valanarasu et al., “Medical Transformer: Gated axial-attention
for medical image segmentation,” in Proc. Int. Conf. Med. Imag.
Comput. Comput.-Assist. Interv., 2021, pp. 61–71.
[37] R. Child, S. Gray, A. Radford, I. Sutskever, “Generating long sequences
with sparse transformers,” arXiv:1904.10509, 2019.
[38] P. Cai, L. Jiang, Y. Li, and L. Lan, “Pubic symphysis-fetal head
segmentation using pure transformer with bi-level routing attention,”
arXiv:2310.00289, 2023.
[39] Y. Liu et al., “Global Attention Mechanism: Retain information to
enhance channel-spatial interactions,” arXiv:2112.05561, 2021.
[40] X. Chu et al., “Twins: Revisiting the design of spatial attention in vision
transformers,” in Proc. Adv. Neural Inf. Process. Syst., 2021, pp. 9355–
9366.
[41] K. Li et al., “Uniformer: Unifying convolution and self-attention for
visual recognition” IEEE Trans. Pattern Anal. Mach. Intell., vol. 45, no.
10, pp. 12581–12600, Oct. 2023.
[42] N. C. F. Codella et al., “Skin lesion analysis toward melanoma detection:
A challenge at the 2017 International symposium on biomedical imaging
(ISBI), hosted by the international skin imaging collaboration (ISIC),”
in IEEE 15th Int. Symp. Biomed. Imaging, Washington, DC, USA, 2018,
pp.168–172.
[43] P. Tschandl, C. Rosendahl, and H. Kittler1, “Data Descriptor: The
HAM10000 dataset, a large collection of multi-source dermatoscopic
images of common pigmented skin lesions,” Sci. Data, 2018.
[44] J. Bernal et al., “WM-DOVA maps for accurate polyp highlighting in
colonoscopy: Validation vs. saliency maps from physicians,” Comput.
Med. Imaging Graph., vol. 43, pp. 99–111, 2015.
[45] B. Chen, Y. Liu, Z. Zhang, G. Lu, and A. W. K. Kong, “TransAttUnet:Multi-level attention-guided u-net with transformer for medical image
segmentation,” IEEE Trans. Emerg. Topics Comput. Intell., 2023.
[46] Q. Xu, Z. Ma, N. He, and W. Duan, “DCSAU-Net: A deeper and more
compact split-attention U-Net for medical image segmentation,” Comput.
Biol. Med., vol. 154, pp. 106626, 2023.
[47] N. Siddique, S. Paheding, C. P. Elkin, and V. Devabhaktuni, “U-Net
and its variants for medical image segmentation: A review of theory
and applications,” IEEE Access, vol. 9, pp. 82031–82057, 2021.
[48] R. Azad et al., “Medical image segmentation review: The success of
U-Net,” arXiv:2211.14830, 2022.
[49] A. Hatamizadeh et al., “UNETR: Transformers for 3D medical im-
age segmentation,” in Proc. IEEE Winter Conf. Appl. Comput. Vis.,
Waikoloa, HI, USA, 2022, pp. 1748–1758.
[50] A. Hatamizadeh, V. Nath, Y. Tang, D. Yang, H. R. Roth, and D. Xu,
“Swin UNETR: Swin transformers for semantic segmentation of brain
tumors in MRI images,” in Proc. Int. Conf. Med. Imag. Comput.
Comput.-Assist. Interv. Brainlesion Workshop, 2021, pp. 272–284.
[51] Y. Rao, W. Zhao, B. Liu, J. Lu, J. Zhou, and C. Hsieh, “DynamicViT:
Efficient vision transformers with dynamic token sparsification,” in Proc.
Adv. Neural Inf. Process. Syst., 2021, pp. 13937–13949.
[52] J. Hu, L. Shen, and G. Sun, “Squeeze-and-excitation networks,” in Proc.
IEEE Conf. Comput. Vis. Pattern Recognit., Salt Lake City, UT, USA,
2018, pp. 7132–7141.
[53] M. Jaderberg, K. Simonyan, A, Zisserman, and K. Kavukcuoglu, “Spa-
tial transformer networks,” in Proc. Adv. Neural Inf. Process. Syst., 2015.
[54] S. Woo et al., “CBAM: Convolutional block attention module,” in Proc.
Eur. Conf. Comput. Vis., 2018, pp. 3–19.
[55] Z. Xia, X. Pan, S. Song, L. E. Li, and G. Huang. “Vision transformer
with deformable attention,” in Proc. IEEE Conf. Comput. Vis. Pattern
Recognit., New Orleans, LA, USA, 2022, pp. 4784–4793
[56] B. Landman et al., “Synapse Multi-Organ Abdominal CT Segmentation
Dataset,” Multi-Atlas Labeling Beyond the Cranial Vault–Workshop and
Challenge, [Online]. Available: https://www.synapse.org/#!
Synapse:syn3193805/wiki/217789.
[57] A. Paszke et al., “Automatic differentiation in PyTorch,” in Proc. Adv.
Neural Inf. Process. Syst., 2017, pp. 1–4.
[58] J. Deng, W. Dong, R. Socher, L.-J. Li; K. Li, and Li Fei-Fei, “ Imagenet:
A large-scale hierarchical image database,” in Proc. IEEE Conf. Comput.
Vis. Pattern Recognit., Miami, FL, USA, 2009, pp. 248–255.
[59] D. Kingma, and J. Ba, “Adam: A method for stochastic optimization,”
in Proc. Int. Conf. Learn. Representations, 2015.
[60] M. M. Rahman, and R. Marculescu, “Medical image segmentation via
cascaded attention decoding,” in Proc. IEEE Winter Conf. Appl. Comput.
Vis., Waikoloa, HI, USA, 2023, pp. 6211–6220.