开源分子对接软件Ledock之--CPU并行用于虚筛选
文章目录
- 前言
- 一、准备工作
- 二、使用步骤
-
- 1. LeDock的安装
- 2. LeDock虚拟筛选脚本ledock_hts.sh的调整
- 3. 运行虚拟筛选
-
- 3.1 修改输入参数
- 3.2 运行虚拟筛选任务并通过Slurm命令查看任务队列运行情况
- 3.3 运行结束后汇集虚拟筛选结果
- 3.4 用ledock获取感兴趣分子的对接pose
- 3.5 用pymol查看感兴趣分子的对接pose
- 三、使用注意事项
- 总结
- 参考文献:
前言
LeDock是一款开源分子对接软件,过往的对比研究显示LeDock在配体构象生成和打分上具有比较优势,从使用上看对接计算速度亦具有明显优势。LeDock默认使用单线程CPU计算分子对接任务,单机使用对当前多核心多线程的CPU利用不够。
本文介绍采用GNU parallel并行计算以及使用Slurm提交任务提高CPU利用率,实现高通量虚拟筛选,适合单机及HPC。
过往使用案例中,100个线程的机器每天可实现30~50万+小分子数据库的虚拟筛选,每天可以完成一个靶点在Specs数据库上的虚拟筛选工作。具有较好的性价比和实用性。
一、准备工作
操作系统及程序安装:
系统:Ubuntu 22.04
GNU Parallel的安装: Parallel的安装介绍
Slurm安装:Slurm安装介绍
LeDock安装:开源分子对接软件LeDock之–在单机上快速部署及使用
受体文件:pdb格式,pdbid 8U2E,命名为 com.pdb(其配体编号为UP9, 同上一篇博文)
虚拟筛选的数据库来源:ZINC 20 ,下载mol2结构文件,用lefrag拆分,每个小分子保存为一个.mol2文件。随机取其中999个分子,加入了UP9.mol2,共1000个分子,代表虚拟筛选的示例数据库。以上 1000个文件保存至数据库ligands_folder文件夹。
ZINC下载如下所示:
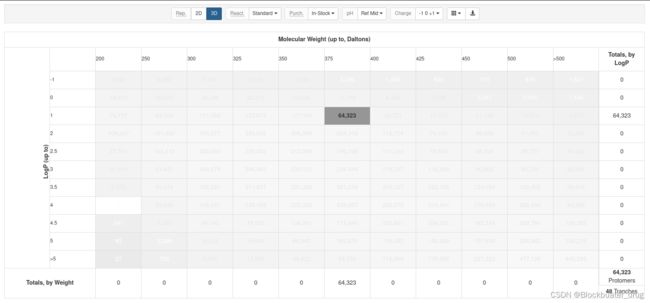
二、使用步骤
1. LeDock的安装
LeDock的安装,请参考上一篇博文:开源分子对接软件LeDock之–在单机上快速部署及使用
按照操作下载获得包含LeDock程序的 Lephar文件夹。
2. LeDock虚拟筛选脚本ledock_hts.sh的调整
ledock_hts.sh是基于LeDock,采用GNU Parallel和Slurm,在集群上用来高通量虚拟筛选的脚本,可以部署在单机或者HPC。脚本来源:https://github.com/cemilcansaylan/ledock_hts.
原脚本存在一些typo,使用的话需要做一些调整。 调整后的脚本ledock_hts_mod.sh可在此下载:ledock_hts_mod.sh
以下将展示如何使用ledock_hts_mod.sh,通过CPU并行用于高通量虚拟筛选,适合本地机器使用;如果使用HPC,在HPC Slurm安装好的前提下,调整Slurm运行脚本#SBATCH部分的内容,可以实现对多个计算节点的控制。
将以上提到的4个文件/文件夹保存于虚拟筛选的工作主目录(即 $main_folder),工作主目录内容如下所示:

3. 运行虚拟筛选
3.1 修改输入参数
用ledock_hts_mod.sh脚本的输入参数运行:
export main_folder=$(pwd) ##获取main_folder路径
# 准备虚拟筛选输入文件和sbatch文件
./ledock_hts_mod.sh --workdir $main_folder \
--task_number 2 \
--receptor $main_folder/com.pdb \
--ligands_folder $main_folder/ligands_folder \
--center_coord 14.063 -5.323 -15.805 \
--box_size 21.341 14.044 18.433 \
--account bbdrug \
--partition compute \
--cpu 12 \
--ledock_path $main_folder/Lephar \
--gnu_parallel_path /usr/bin
以上参数的解释如下,具体需要根据实际情况配置,但每一项均不可缺省:
–workdir $main_folder ##主文件夹路径
–task_number 6 ##任务数量,即虚拟筛选分多少个task
–receptor $main_folder/com.pdb ##受体文件地址,可以是受体-配体复合物,也可以是单独受体结构
–ligands_folder $main_folder/ligands_folder ##数据库文件夹路径,保存拆分为包含单个mol2格式分子的数据库
–center_coord 14.063 -5.323 -15.805 ##docking box的中心点x y z,该项必须设置
–box_size 21.341 14.044 18.433 ##box size的x y z尺寸,该项必须设置
–account bbdrug ##Slurm Sbatch的账户名,设置一个即可
–partition compute ##Slurm Sbatch的partition,需要与Slurm设置匹配
–cpu 12 ##每个task使用的CPU数量,需要少于核数或者线程数
–ledock_path $main_folder/Lephar ##LeDock程序所在文件夹路径
–gnu_parallel_path /usr/bin ##parallel所在文件夹路径
以上就完成了ledock_hts_mod.sh参数的设定。
运行之后产生3项:sbatch_submit.job,inputs文件夹,docking_output文件夹。
以上,也可以用configure file方式运行ledock_hts_mod.sh,两者输出结果相同。
configure file内容如下,保存为ledock_hts.conf (注意:$main_folder需要使用绝对路径)
--workdir $main_folder
--task_number 6
--receptor $main_folder/com.pdb
--ligands_folder $main_folder/ligands_folder
--center_coord 14.063 -5.323 -15.805
--box_size 21.341 14.044 18.433
--account bbdrug
--partition compute
--cpu 12
--ledock_path $main_folder/Lephar
--gnu_parallel_path /usr/bin
configure file作为输入的运行方式:
./ledock_hts_mod.sh --config ./ledock_hts.conf
3.2 运行虚拟筛选任务并通过Slurm命令查看任务队列运行情况
#运行虚拟筛选任务:
bash ./sbatch_submit.job
#查看任务队列运行情况:
squeue
在笔者的工作站电脑上,运行~20min完成。
每个task结束后都会有slurm的.err和.out文件输出在主文件夹,可以在#SBATCH 脚本部分设置关闭输出。
3.3 运行结束后汇集虚拟筛选结果
用ledock_anal汇集对接结果到main_folder中的hts_summary.csv:
cd $main_folder/ligands_folder
$main_folder/Lephar/ledock_anal/ledock_anal.csh
mv ./docking_summary.txt $main_folder/hts_summary.csv
cd $main_folder
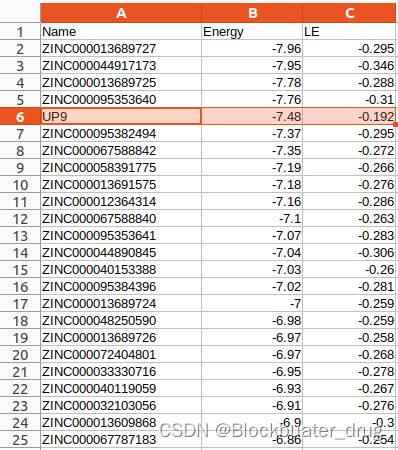
以下是部分hts_summary.csv 中的结果,展示有Energy及LE值。有4个分子打分高于固有配体UP9。
3.4 用ledock获取感兴趣分子的对接pose
在ligands_folder生成pose的pdb文件:
$main_folder/Lephar/ledock_linux_x86 -spli $main_folder/ligands_folder/UP9.dok
$main_folder/Lephar/ledock_linux_x86 -spli $main_folder/ligands_folder/ZINC000013689727.dok
$main_folder/Lephar/ledock_linux_x86 -spli $main_folder/ligands_folder/ZINC000044917173.dok
$main_folder/Lephar/ledock_linux_x86 -spli $main_folder/ligands_folder/ZINC000013689725.dok
$main_folder/Lephar/ledock_linux_x86 -spli $main_folder/ligands_folder/ZINC000095353640.dok
3.5 用pymol查看感兴趣分子的对接pose
pymol $main_folder/com.pdb \
$main_folder/inputs/pro.pdb \
$main_folder/ligands_folder/UP9_dock001.pdb \
$main_folder/ligands_folder/ZINC000013689727_dock001.pdb \
$main_folder/ligands_folder/ZINC000044917173_dock001.pdb \
$main_folder/ligands_folder/ZINC000013689725_dock001.pdb \
$main_folder/ligands_folder/ZINC000095353640_dock001.pdb
 可以看到打分较高分子在母核部分叠合较多,侧链并不完全与固有配体UP9(绿色)一致。
可以看到打分较高分子在母核部分叠合较多,侧链并不完全与固有配体UP9(绿色)一致。
三、使用注意事项
(1) center_coord, box_size分别是对接盒子长方体的中心点和长宽高尺寸,确定方式有多种,此处不再详述。需要按照对接需求设置;建议提交运行前,确认输入数据无误,保证docking site 为预定的空间。
(2)由于使用了export main_folder,整个运行及处理过程中,需要保持在同一个Terminal中输入,新打开的Terminal需要cd到main_folder,再次输入: export main_folder=$(pwd),方可继续使用。
总结
本文介绍采用GNU parallel并行计算以及使用Slurm提交任务提高CPU利用率,实现LeDock高通量虚拟筛选,适合在单机及HPC上配置。
参考文献:
- https://editor.csdn.net/md/?articleId=136072504
- https://editor.csdn.net/md/?articleId=136057641
- https://editor.csdn.net/md/?articleId=136002697