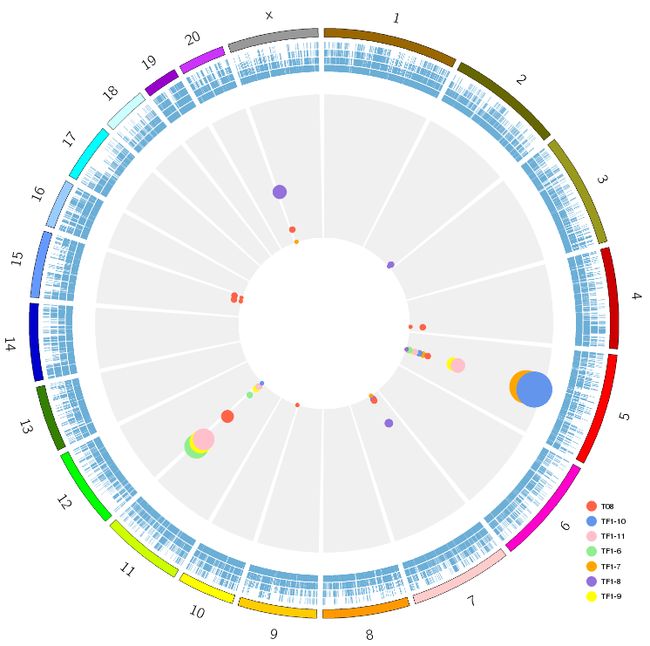
circso 对数据进行可视化
circos可以用来绘制圈图,能够对染色体上的数据进行可视化,首先需要一个染色体的文件
染色体的文件如下,每列之间空格分隔
chr - chr1 1 0 227556264 chr1 chr - chr2 2 0 192460366 chr2 chr - chr3 3 0 192294377 chr3 chr - chr4 4 0 170955103 chr4 chr - chr5 5 0 189454096 chr5 chr - chr6 6 0 181584905 chr6 chr - chr7 7 0 171882078 chr7 chr - chr8 8 0 146850525 chr8 chr - chr9 9 0 133195287 chr9
第一列 : 表名这行数据是染色体的数据
第二列: -
第三列: 染色体的名称,会显示在图上
第四列: 染色体编号
第五列:染色体的起始位置
第六列: 染色体的终止位置
第七列:染色体的颜色
在circos中有一些默认的颜色设置,用rgb设置一些默认的颜色名, 可以在circos的安装目录的etc/colors.conf 文件中查看,比如chr1 的颜色设置保存在colors.ucsc.conf 中,内容如下
chr1 = 153,102,0 chr2 = 102,102,0 chr3 = 153,153,30 chr4 = 204,0,0 chr5 = 255,0,0 chr6 = 255,0,204 chr7 = 255,204,204 chr8 = 255,153,0 chr9 = 255,204,0 chr10 = 255,255,0 chr11 = 204,255,0 chr12 = 0,255,0 chr13 = 53,128,0 chr14 = 0,0,204 chr15 = 102,153,255 chr16 = 153,204,255 chr17 = 0,255,255 chr18 = 204,255,255 chr19 = 153,0,204 chr20 = 204,51,255 chr21 = 204,153,255 chr22 = 102,102,102 chr23 = 153,153,153 chrX = 153,153,153 chr24 = 204,204,204 chrY = 204,204,204 chrM = 204,204,153 chr0 = 204,204,153 chrUn = 121,204,61 chrNA = 255,255,255
我们也可以自定义一些颜色的名称,将其添加到colors.conf 文件中保存即可,比如自定义颜色
sample1 = 255,99,71
然后就可以使用sample1 作为颜色名了,在circos中还有很多颜色的名字,都是有规律的,比如
vvlred : very very light red
vlred : very light red
lred : light red
red : red
dred : dark red
vdred : very dark red
vvdred : very very dark red
常用的颜色red, green, blue, yellow 都支持上面的颜色定义, 除此之外,还有其他很多的内置颜色, 详细信息可以查看circos安装目录下的/etc/colors.conf 文件查看, 里面的注释部分详细说明了颜色的定义方式。
接下来定义想要展示的数据,这里展示基因在染色体上的区间信息,通过 tile 的方式进行展示
chr1 8825 17551 chr1 31200 36882 chr1 44089 61948 chr1 91317 91388 chr1 91669 91774 chr1 105969 115431 chr1 115880 126341 chr1 137263 152263 chr1 185470 192339 chr1 200946 216412 chr1 246699 257364 chr1 302816 308640 chr1 326354 327127 chr1 346836 363346 chr1 351339 352292 chr1 373325 374824 chr1 469751 470980
第一列: 染色体名称
第二列:基因的起始位置
第三列:基因的终止位置
在下来准备突变位点的信息,用scatter 的方式进行展示
chr1 13400340 13400340 glyph_size=20,fill_color=red
第一列: 染色体名称
第二列: 突变的起始位置
第三列: 突变的终止位置
前三列的数据是必须的,第四列用来设置每个店的大小和颜色
最后是cir<<include colors_fonts_patt<ideogram>
<spacing> default = 10u break = 2u </spacing> radius = 0.9r thickness = 40p fill = yes fill_color = black stroke_thickness = 2 stroke_color = black show_label = yes label_font = default label_radius = dims(ideogram,radius) + 0.05r label_with_tag = yes label_size = 50 label_parallel = yes label_case = lower label_format = eval(sprintf("%s",var(label))) </ideogram>
<image> <<include etc/image.conf>> </image> karyotype = monkey_karyotype.txt chromosomes_units = 1000000<plots> <plot> type = tilefile = genes.txt r1 = 1.0r r0 = 0.88r orientation = out layers = 4 margin = 0.02u thickness = 30 padding = 5 layers_overflow = hide stroke_thickness = 1 stroke_color = blue color = blue </plot> <plot> type = scatter file = scatter.txt stroke_thickness = 0 glyph = circle max = 47000 min = 100 r1 = 0.8r r0 = 0.3r<rules> <rule> condition = var(value) < 1000 glyph_size = 20 </rule> <rule> condition = var(value) > 1000 && var(value) < 10000 glyph_size = 30 </rule> <rule> condition = var(value) > 10000 gplyph_size = 50 </rule> </rules> <backgrounds> <background> color = vvlgrey y0 = 100 </background> </backgrounds> </plot> </plots> <<include etc/housekeeping.conf>>
直接在circos.conf的目录运行circos,就会生成circos.png 和circos.svg;
因为circos中没有添加图例的方法,只能手工添加,利用R绘制图列,再用windows自带的画图工具对图片进行剪裁,
最终效果如下: