自然语言处理学习篇02——Edit Distance
第二章 Edit Distance
本篇内容将讲述Edit Distance(编辑距离的定义详见正文),具体又包含5个方面的内容:
- Defining Minimum Edit Distance
- Computing Minimum Edit Distance
- Backtrace for Computing Alignments
- Weighted Minimum Edit Distance
- Minimum Edit Distance in Computational Biololgy
1. Definition of Minimum Edit Distance
Edit Distance用于衡量两个strings之间的相似性。
两个strings之间的
Minimum edit distance是指把其中一个string通过编辑(包括插入,删除,替换操作)转换为另一个string的最小操作数。
如上图所示,d(deletion)代表删除操作,s(substitution)代表替换操作,i(insertion)代表插入操作。
(为了简单起见,后面的Edit Distance 简写为ED)
如果每种操作的cost(成本)为1,那么ED = 5.
如果s操作的cost为2(即所谓的Levenshtein Distance),ED = 8.
2. Computing Minimum Edit Distance
那么如何找到两个strings的minimun edit distance呢?要知道把一个string转换为另一个string可以有很多种方法(或者说“路径“)。我们所知道起始状态(第一个string)、终止状态(另一个string)、基本操作(插入、删除、替换),要求的是最短路径。
对于如下两个strings:
X的长度为n
Y的长度为m
我们定义D(i,j)为 X 的前i个字符 X[1...i] 与 Y 的前j个字符 Y[1...j] 之间的距离,其中0<i<n, 0<j<m,因此X与Y的距离可以用D(n,m)来表示。
假如我们想要计算最终的D(n,m),那么可以从头开始,先计算D(i, j) (i和j从1开始)的值,然后基于前面的结果计算更大的D(i, j),直到最终求得D(n,m)。
算法过程如下图所示:
上图中使用的是”Levenshtein Distance“即替换的成本为2.
请读者深入理解一下上图中的循环体部分: D(i,j)可能的取值为:
1. D(i-1, j) +1 ;
2. D(i, j-1) +1 ;
3. D(i-1, j-1) + 2 (当X新增加的字符和Y新增加的字符不同时,需要替换)或者 + 0(即两个字符串新增加的字符相同)
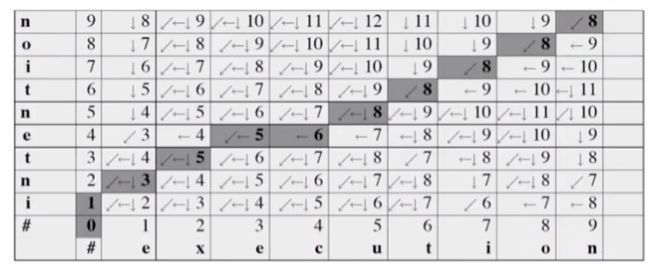
下图即对字符串 INTENTION 和 EXECUTION 一步步求ED形成的表。左上角画红圈的8就是两个字符串间的最小ED。
3. Backtrace for Computing Alignments
上一节课我们求得了Edit distance,但是仅有Edit distance也是是不够的,有时我们也需要把两个strings中的每个字符都一一对应起来(有的字母会与“空白”对应),这可以通过Backtrace(追踪)ED的计算过程得到。
通过上一节我们知道,D(i, j)的取值来源有三种,D(i-1, j)、D(i, j-1)或者D(i-1, j-1),下表通过添加箭头的方式显而易见地给出来整个表格的计算过程(下面的阴影表示的只是一种路径,你会发现得到最后结果的路径不是惟一的,因为每个单元格数字可能由左边、下边或者左下边的得到)。
从表格右上角开始,沿着追踪的剪头,就可以拎出一条路径出来(不惟一),这条路径的剪头可以轻易的展现是通过哪种方法(插入、删除、替换)完成的。
表格右上角阴影部分四个格子,路径只有一条,我们也可以很轻易地看出最后四个字母是相同的,但这种情况并不绝对,比如中间的阴影6格也只有一种路径,可是却分别对应于字母e和c。
算法实现“寻找路径”的思想很简单——就是给每个单元格定义一个指针,指针的值为LEFT/DOWN/DIAG(不明白为什么他为什么说是指针),如下图所示。
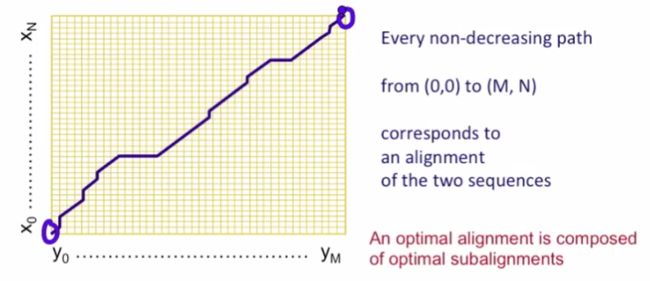
想一下普通的情况,如下图,从(0,0)到(M,N)的任何一条非下降路径都对应于两个strings间的一个排列,而最佳的排列由最佳的子排列组成。
简单思考一下算法的性能
Time: O(nm)
Space: O(nm)
Backtrace: O(n+m)
4. Weighted Minimum Edit Distance
ED也可以添加权重,因为在拼写中,某些字母更容易写错。如下图显示的混淆矩阵,数值越大就代表被误写的可能性越高。如a就很可能被误写为e,i,o,u
众所周知,键盘排布会对误写产生影响。
Weighted Min Edit Distance的算法如下图所示
这幅图将del、ins、sub三种操作都定义了不同的权重,在“莱温斯基距离“中,del和ins的cost都是1,sub是2。
5. Minimum Edit Distance in Computational Biology
本段讲述Minimum Edit Distance在计算生物学中的应用。比如比较如下图(上班部分)两个基因组序列,我们希望最后能把两个序列对齐(下半部分),进而研究不同的基因片段的功能等。
在Natural Language Processing我们讨论了最小distance和weight,而在Computational Biology中我们将要介绍最大Similarity(相似性)和scores。
在Computational Biology中有个重要算法——Needleman-Wunsch算法。
(未完待续)