林智仁LibSVM包的详细说明
1.了解libsvm工具包
LIBSVM是台湾大学林智仁(Lin Chih-Jen)教授等2001年开发设计的一个简单, 易于使用和快速有效的SVM模式识别与回归的软件包, 他不但提供了编译好的可在Windows系列系统的执行文件, 还提供了源代码, 方便改进, 修改以及在其它操作系统上应用; 该软件对SVM所涉及的参数调节相对比较少, 提供了很多的默认参数, 利用这些默认参数可以解决很多问题; 并提供了交互检验(Cross Validation)的功能. 该软件包可在http://www.csie.ntu.edu.tw/~cjlin/ 免费获得. 该软件可以解决C-SVM, ν-SVM, ε-SVR和ν-SVR等问题, 包括基于一对一算法的多类模式识别问题.
这套库可以从http://www.csie.ntu.edu.tw/~cjlin/libsvm/index.html免费获得, 目前已经发展到3.20版(2014.12.12更新).下载.tar.gz格式的版本, Windows下也可以直接解压, 主要有6个文件夹和一些源码文件,如图所示.
Java: 主要是应用于java平台;
matlab: windows下64位matlab平台;
python: 是用来参数优选的工具, 稍后介绍;
svm-toy: 一个可视化的工具, 用来展示训练数据和分类界面, 里面是源码, 其编译后的程序在windows文件夹下;
tools: 主要包含四个python文件, 用来数据集抽样(subset), 参数优选(grid), 集成测试(easy), 数据检查(checkdata);
windows: 包含libSVM四个exe程序包, 我们所用的库就是他们.
此外,文件夹里面还有个heart_scale, 是一个样本文件, 可以用记事本打开, 用来测试用的.
其他.h和.cpp文件都是程序的源码, 可以编译出相应的.exe文件. 其中, 最重要的是svm.h和svm.cpp文件, svm-predict.c, svm-scale.c和svm-train.c(还有一个svm-toy.c在svm-toy文件夹中)都是调用的这个文件中的接口函数, 编译后就是windows下相应的四个exe程序. 另外, 里面的README跟FAQ也是很好的文件, 对于初学者如果W文较好的话, 可以看一下.
对于仅仅使用libsvm库的人来说, windows下的4个exe包已经足够了, 如果是为了做深入研究, 可以按照自己的思路文件夹中的.cpp和.c文件, 然后编译验证)
2.下面开始测试libsvm工具包
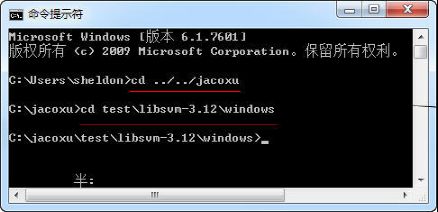
1). 把LibSVM包解压到相应的目录(因为我只需要里面windows文件夹中的exe程序包, 我们也可以只把windows文件夹拷到相应的目录), 比如C:\jacoxu\test\libsvm-3.12
2). 在电脑”开始”的”运行”中输入cmd, 进入DOS环境. 定位到C:\jacoxu\test\libsvm-3.12下, 具体命令如下:
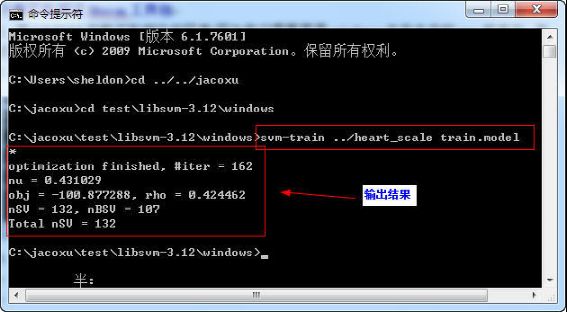
3). 进行libsvm训练, 输入命令:
svm-train ../heart_scale train.model
heart_scale: 是目录下的已经存在的样本文件, 要换成自己的文件, 只需改成自己的文件名就可以了;
train.model: 是创建的结果文件, 保存了训练后的结果:
其中:
#iter 为迭代次数;
nu 是选择的核函数类型的参数;
obj 为SVM文件转换为的二次规划求解得到的最小值;
rho 为判决函数的偏置项b;
nSV 为标准支持向量个数(0<a[i]<c);
nBSV 为边界上的支持向量个数(a[i]=c);
Total nSV为支持向量总个数(对于两类来说, 因为只有一个分类模型Total nSV = nSV,但是对于多类, 这个是各个分类模型的nSV之和).
在目录下, 还可以看到产生了一个train.model文件, 可以用记事本打开, 记录了训练后的结果.
1.svm_type c_svc //所选择的svm类型, 默认为c_svc
2.kernel_type rbf //训练采用的核函数类型, 此处为RBF核
3.gamma 0.0769231 //RBF核的参数γ
4.nr_class 2 //类别数, 此处为两分类问题
5.total_sv 132 //支持向量总个数
6.rho 0.424462 //判决函数的偏置项b
7.label 1 -1 //原始文件中的类别标识
8.nr_sv 64 68 //每个类的支持向量机的个数
9.SV //以下为各个类的权系数及相应的支持向量
10.1 1:0.166667 2:1 3:-0.333333 … 10:-0.903226 11:-1 12:-1 13:1
11.0.5104832128985164 1:0.125 2:1 3:0.333333 … 10:-0.806452 12:-0.333333 13:0.5
12. ………
13.-1 1:-0.375 2:1 3:-0.333333…. 10:-1 11:-1 12:-1 13:1
14.-1 1:0.166667 2:1 3:1 …. 10:-0.870968 12:-1 13:0.5
这里注意, 第二行出现的权系数为小数(0.5104832128985164)是因为这个点属于非边界上的支持向量, 即: (0<a[i]<c).
其他的两个(svm-predict, svm-scale)的使用过程类似.
3.libsvm使用规范
1). libSVM的数据格式
Label 1: value 2:value ….
Label: 是类别的标识, 比如上节train.model中提到的1 -1, 你可以自己随意定, 比如-10, 0, 15. 当然, 如果是回归, 这是目标值, 就要实事求是了.
Value: 就是要训练的数据, 从分类的角度来说就是特征值, 数据之间用空格隔开.比如: -15 1:0.708 2:1056 3:-0.3333
此外,需要注意的是, 如果特征值为0, 特征冒号前面的(姑且称做序号)可以不连续. 如:-15 1:0.708 3:-0.3333表明第2个特征值为0, 从编程的角度来说, 这样做可以减少内存的使用, 并提高做矩阵内积时的运算速度. 我们平时在matlab中产生的数据都是没有序号的常规矩阵, 所以为了方便最好编一个程序进行转化.
2). svmscale的用法
svmscale是用来对原始样本进行缩放的, 范围可以自己定, 一般是[0,1]或[-1,1]. 缩放的目的主要是
1) 防止某个特征过大或过小, 从而在训练中起的作用不平衡;
2) 为了计算速度. 因为在核计算中, 会用到内积运算或exp运算, 不平衡的数据可能造成计算困难.
用法: svmscale [-l lower] [-u upper]
[-y y_lower y_upper]
[-s save_filename]
[-r restore_filename] filename
其中, []中都是可选项:
-l: 设定数据下限; lower: 设定的数据下限值, 缺省为-1
-u: 设定数据上限; upper: 设定的数据上限值, 缺省为1
-y: 是否对目标值同时进行缩放; y_lower为下限值, y_upper为上限值;
-s save_filename: 表示将缩放的规则保存为文件save_filename;
-r restore_filename: 表示将按照已经存在的规则文件restore_filename进行缩放;
filename: 待缩放的数据文件, 文件格式按照libsvm格式.
默认情况下, 只需要输入要缩放的文件名就可以了: 比如(已经存在的文件为test.txt)
svmscale test.txt
这时, test.txt中的数据已经变成[-1,1]之间的数据了. 但是, 这样原来的数据就被覆盖了, 为了让规划好的数据另存为其他的文件, 我们用一dos的重定向符>来另存为(假设为out.txt):
svmscale test.txt > out.txt
运行后, 我们就可以看到目录下多了一个out.txt文件, 那就是规范后的数据. 假如, 我们想设定数据范围[0,1], 并把规则保存为test.range文件:
svmscale –l 0 –u 1 –s test.range test.txt > out.txt
这时, 目录下又多了一个test.range文件, 可以用记事本打开, 下次就可以用-r test.range来载入了.
3). svmtrain的用法
svmtrain我们在前面已经接触过, 他主要实现对训练数据集的训练, 并可以获得SVM模型.
用法: svmtrain [options] training_set_file [model_file]
其中, options为操作参数, 可用的选项即表示的涵义如下所示:
-s 设置svm类型:
0 – C-SVC
1 – v-SVC
2 – one-class-SVM
3 – ε-SVR
4 – n – SVR
-t 设置核函数类型, 默认值为2
0 — 线性核: μ‘∗ν
1 — 多项式核: (γ∗μ‘∗ν+coef0)degree
2 — RBF核: exp(–γ∗∥μ−ν∥2)
3 — sigmoid 核: tanh(γ∗μ‘∗ν+coef0)
-d degree: 核函数中的degree设置(针对多项式核函数)(默认3);
-g r(gama): 核函数中的gamma函数设置(针对多项式/rbf/sigmoid核函数)(默认1/ k);
-r coef0: 核函数中的coef0设置(针对多项式/sigmoid核函数)((默认0);
-c cost: 设置C-SVC, e -SVR和v-SVR的参数(损失函数)(默认1);
-n nu: 设置v-SVC, 一类SVM和v- SVR的参数(默认0.5);
-p p: 设置e -SVR 中损失函数p的值(默认0.1);
-m cachesize: 设置cache内存大小, 以MB为单位(默认40);
-e eps: 设置允许的终止判据(默认0.001);
-h shrinking: 是否使用启发式, 0或1(默认1);
-wi weight: 设置第几类的参数C为weight*C (C-SVC中的C) (默认1);
-v n: n-fold交互检验模式, n为fold的个数, 必须大于等于2;
-b 概率估计: 是否计算SVC或SVR的概率估计, 可选值0或1, 默认0;
model_file: 可选项, 为要保存的结果文件, 称为模型文件, 以便在预测时使用.
其中-g选项中的k是指输入数据中的属性数. option -v 随机地将数据剖分为n部分并计算交互检验准确度和均方根误差. 以上这些参数设置可以按照SVM的类型和核函数所支持的参数进行任意组合, 如果设置的参数在函数或SVM类型中没有也不会产生影响, 程序不会接受该参数; 如果应有的参数设置不正确, 参数将采用默认值.
training_set_file是要进行训练的数据集; model_file是训练结束后产生的模型文件, 文件中包括支持向量样本数, 支持向量样本以及lagrange系数等必须的参数; 该参数如果不设置将采用默认的文件名, 也可以设置成自己惯用的文件名.
默认情况下, 只需要给函数提供一个样本文件名就可以了, 但为了能保存结果, 还是要提供一个结果文件名, 比如: test.model, 则命令为:
svmtrain test.txt test.model
4). svmpredict 的用法
svmpredict 是根据训练获得的模型, 对数据集合进行预测.
用法: svmpredict [options] test_file model_file output_file
其中, options为操作参数, 可用的选项即表示的涵义如下所示:
-b probability_estimates: 是否需要进行概率估计预测, 可选值为0 或者1, 默认值为0.
model_file: 是由svmtrain 产生的模型文件;
test_file: 是要进行预测的数据文件, 格式也要符合libsvm格式, 即使不知道label的值, 也要任意填一个, svmpredict会在output_file中给出正确的label结果, 如果知道label的值, 就会输出正确率;
output_file: 是svmpredict 的输出文件, 表示预测的结果值