cython优化代码过程问题及解决总结
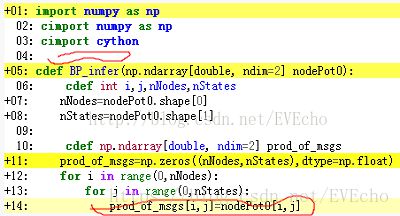
1、同样cdef的矩阵还是在Python平台(还是黄色):
源:
问题:
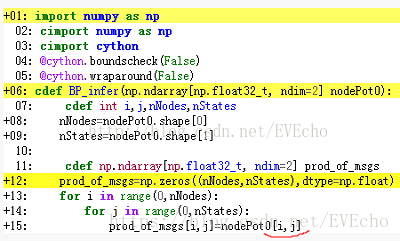
解决1),2),3)
1)加上修饰符:@cython.boundscheck(False)
@cython.wraparound(False)
2)数组索引方式
注意这里nodePot0是二维,所以nodePot0[i]是一维数组,因而也是黄色:
3)每个函数前面都要修饰符
@cython.boundscheck(False)
@cython.wraparound(False)
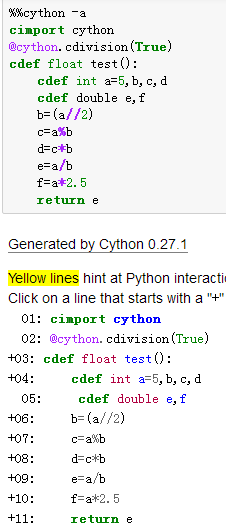
2、%和/也有黄色——@cython.cdivision(True)
解决:在函数或类前面加函数修饰@cython.cdivision(True)(每个函数和类前面都要加)
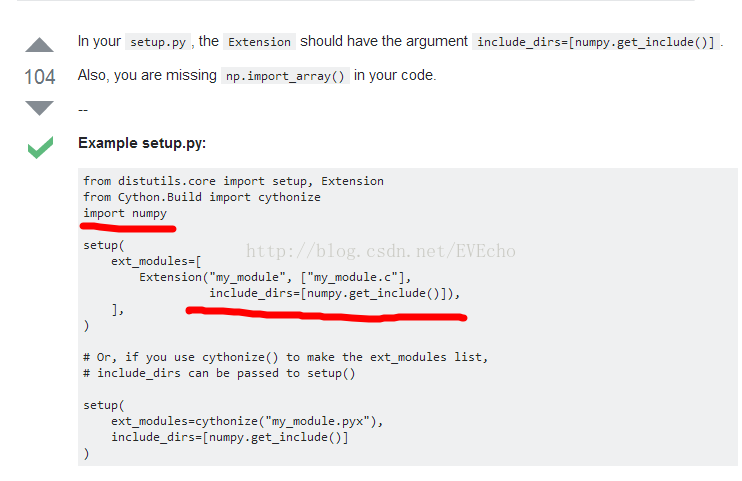
3、cmd命令编译python setup.py build_ext --inplace出现问题:
error c1083 :cannot open include file :numpy/arrayobject.h:No such file or directory
解决:在setup.py文件加include_dirs=[numpy.get_include()]
https://stackoverflow.com/questions/14657375/cython-fatal-error-numpy-arrayobject-h-no-such-file-or-directory
4、cdef的函数出现错误:module‘xxxx’ has no attribute ‘xxx’
实验例子:E:\PycharmProjects\cython_hello
将hello.pyx文件中函数的def改成cdef,再生成pyd文件,通过
import hello
hello.say_hello_to(“cython”)
调用出现问题:AttributeError: module 'hello' has no attribute 'say_hello_to'
原因:python程序中看不到cdef函数,python只能直接调用def的函数,cdef的函数只能通过Python调用def的函数来调用
解决:把和Python接口的函数用def写
5、参数类型传递问题ValueError: Buffer dtype mismatch, expected 'float' but got 'double'
传的python参数是float64的,将pyx中的float和double都改成np.float_64型
类似问题:cdef np.ndarray[int,ndim=1] a=np.zeros((4))提示expected‘int’ but got ‘double’因为np.zeros默认生成float64类型
解决:改成对应类型即可。改成cdef np.ndarray[int,ndim=1] a=np.zeros((4),dtype=int)
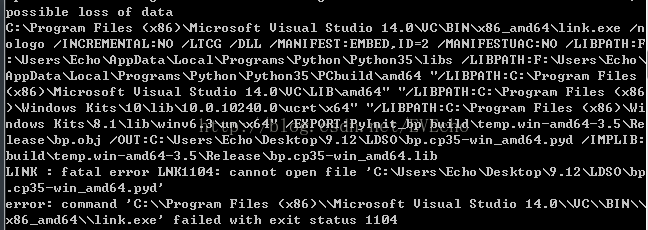
6、cmd运行语句python setup.py build_ext --inplace时错误:
原因:还处于调试状态,结束调试再运行就没问题了
Ps:Python传给cython的参数值会随其在函数中的改变而改变
===========================================================================
具体文献代码问题
7、map,rectangle=EFM.distance_map(img)求出来map和rectangle都是0:
挨个函数想办法看各个关键变量是否求对:labeled_map,inte_hist是对的
将distance_ma()函数语句放到命令行单步调试:发现saliency_at_position()函数输出错误,将此函数语句放到命令行单步调试:chi_square_distance()函数求的所有distance都是0,但是将此函数语句放到命令行单步调试结果又是对的,于是在pyx文件的chi_square_distance()函数中用print()输出关键变量,发现每个vectorA/sum_A都是0
问题所在:vectorA和sum_A都是int型变量,相除之后结果是int型
解决:将sum_A定义为np.float64_t型
8、没有错误提示后运行出现弹窗“python停止工作”,关闭弹窗后,命令窗口:python process finished with exit code -1073741819 (0xC0000005)
通过代码增、减反复操作,先找到是while循环中出现问题,进一步排查发现是语句:e=edgeNum(nbrs[j],i,starV,starE)的问题
回到用python写的BP_salient_map中调试,发现是矩阵starV, starE索引溢出
输出cython写的bp.pyx中的starV, starE在进行循环减1前后的最大最小值
发现starV的最大值多了1,对比C语言写的BP_General_C中,发现for循环中是<=:
因此改为:
由此解决了一直以来的有时候能正确运行,有时候又出现python停止运行的问题
9、显著图显示结果有错误
1)先把BP_General_C.c中DEBUG改为1,即调试模式,看看各个变量的值,修改BP_General_C.c后,需要重新用MATLAB生成BP_General_C.mexw64文件:
在MATLAB命令行输入:mex -setup ![]()
把BP_General_C.c文件放在MATLAB当前文件夹编译:![]()
每次修改了BP_General_C.c都要重新编译
2)c语言printf时,输出格式用错,输出值也会错,例如double类型用了%d格式就会错
3)BP_General_C.c中用数值索引取nodePot和edgePot的值:
一开始在bp.pyx中将nodePot reshape成一维,也用同样句式赋值,但是两边输出的prod_of_msgs和old_bel结果不一样,将bp.pyx中修改,nodePot保持二维,用以下句式赋值,得到的赋值后结果一样:
后面while中也一样,保持nodePot和edgePot原来的维度,直接用矩阵坐标索引
还有一个类似的索引错误就是得到nodeBel的地方,输出发现new_bel其实就可以作为输出,但赋值给原本输出nodeBel时就乱了,之前是:
将nodeBel改成二维,这样复制,输出结果经过reshape就对了:
当然bp.pyx可以直接输出new_bel
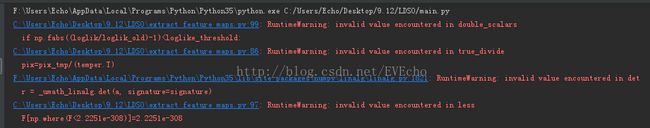
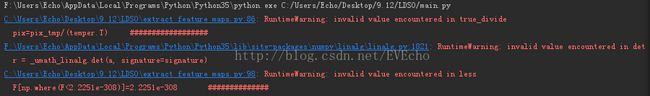
11、
RuntimeWarning:invalid value encountered in double_scalars
RuntimeWarning:invalid value encountered in true_divide
RuntimeWarning:invalid valueencountered in det
RuntimeWarning:invalid valueencountered in less
症结所在是输入gaussPDF()函数的sigma[:,:,i]参数,sigma[:,:,i]出现了奇异矩阵
解决:sigma[:,:,i]每次进入gaussPDF之前先在对角线加上一个小值: