Parzen Window and Likelihood
Kernel density estimation via the Parzen-Rosenblatt window method
Generative Adversarial Nets code
Nice explanation
对Ian提到的采用Gassian Parzen Window 去拟合G产生的样本并估计对数似然性(即给予观察样本,判断模型正确的可能性)如何实现的原理很感兴趣?
于是找到源代码parzen_ll.py来看,
但是苦于并没有学过theano及pylearn2所以看起来不是明白,本来想尝试安装py2learn来测试,结果看了官网的安装要求,请卸载python,清除所有组件。这个代价有点大
,毕竟我是要做Tensorflow的。因此只能破罐破摔来,硬着头皮看代码,有些东西只能靠google,看了一个大概。首先来说一下Parzen Window
2 Defining the Region Rn
Parzen windown 是一种应用广泛的非参数估计方法,从采样的样本 p(xn) p ( x n ) 中估计概率密度函数 p(x) p ( x ) 。
这种方法最基本的思想是给定一个特定的区域(Window)对落入其中的样本进行计数,可以得到样本落入该区域的概率大约为:
从数学的角度来求取在区域R里k个观察样本的概率,我们可以考虑一个Binomial(二项式分布):
然后在二项式分布假设的情况下, 所以我们可以求得其均值为:
在连续性假设条件下:
其中v为区域R的体积,我们可以对公式进行变形,可以得到:
上面简单的公式可以让计算给定点 x x 的概率密度,通过计算有多少点落入确定性区域。
确定区域的两种不同的方法
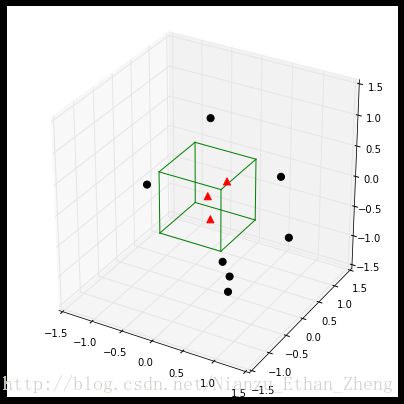
一种方法是固定体积(volume)如下图,原理是取一个固定大小的区域R,观察有多少样本落入其中。
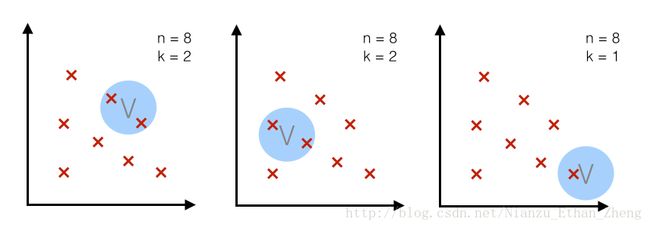
另外一种方法是样本数目K固定;k近邻算法就是这个原理,即是,对于一定大小的样本总数,我们取能框住k个样本的区域R
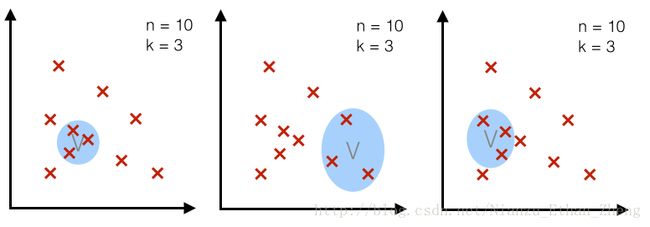
3D- hypercubes
The window function
如上面所示,我们可以可视化区域 R R ,也就很容易计算出多少点在区域内,数学表达上是:
扩展概念(立方体核,hyercube window)我们可以得到下面的概念:
通过下面的公式可以获得落入该区域的样本个数Kn
式中:
也就是:
进而我们可以的得到Rn的概率
而将将简单的立方体核函数替换掉,采用高斯核:
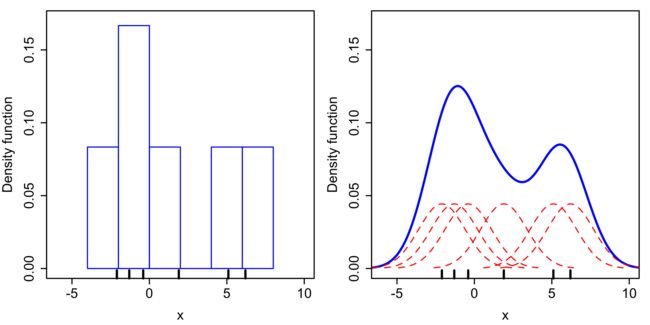
Parzen window estimation
基于上述公式,我们可以得到:
式中有:
Parzen-window技术的关键性参数
这有两个关键性参数:
- 核函数(kernel function)
- 核宽度(window width)
核密度估计需要满足如下条件:
1) 有限值非负密度函数:
2) 对于核宽度的要求
一式原因在于:
构造Parzen window估计函数
def parzen_estimation(mu, sigma, mode='gauss'):
"""
Implementation of a parzen-window estimation
Keyword arguments:
x: A "nxd"-dimentional numpy array, which each sample is
stored in a separate row (=training example)
mu: point x for density estimation, "dx1"-dimensional numpy array
sigma: window width
Return the density estimate p(x)
"""
def log_mean_exp(a):
max_ = a.max(axis=1)
return max_ + np.log(np.exp(a - np.expand_dims(max_, axis=0)).mean(1))
def gaussian_window(x, mu, sigma):
a = (np.expand_dims(x, axis=1) - np.expand_dims(mu, axis=0)) / sigma
b = np.sum(- 0.5 * (a ** 2), axis=-1)
E = log_mean_exp(b)
Z = mu.shape[1] * np.log(sigma * np.sqrt(np.pi * 2))
return np.exp(E - Z)
def hypercube_kernel(x, mu, h):
n, d = mu.shape
a = (np.expand_dims(x, axis=1) - np.expand_dims(mu, axis=0)) / h
b = np.all(np.less(np.abs(a), 1/2), axis=-1)
kn = np.sum(b.astype(int), axis=-1)
return kn / (n * h**d)
if mode is 'gauss':
return lambda x: gaussian_window(x, mu, sigma)
elif mode is 'hypercube':
return lambda x: hypercube_kernel(x, mu, h=sigma)将Parzen Window 方法应用于高斯数据集
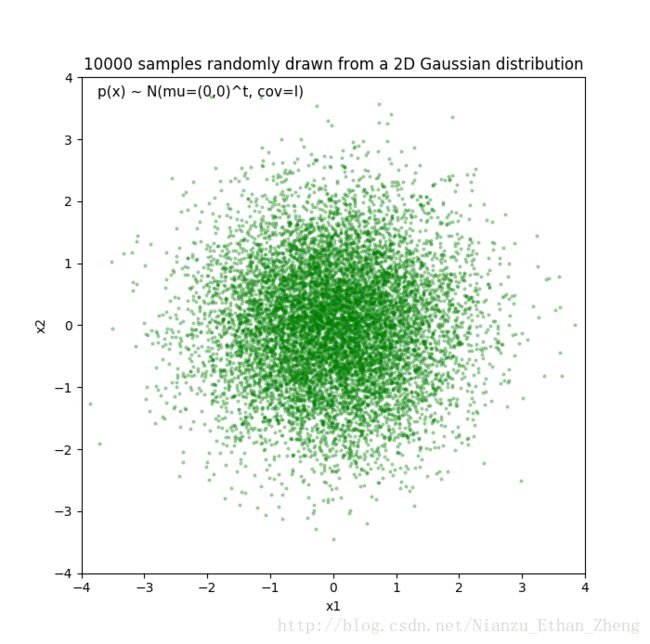
我们构造如下数据集:
设定:
import numpy as np
# Generate 10,000 random 2D-patterns
mu_vec = np.array([0,0])
cov_mat = np.array([[1,0],[0,1]])
x_2Dgauss = np.random.multivariate_normal(mu_vec, cov_mat, 10000)
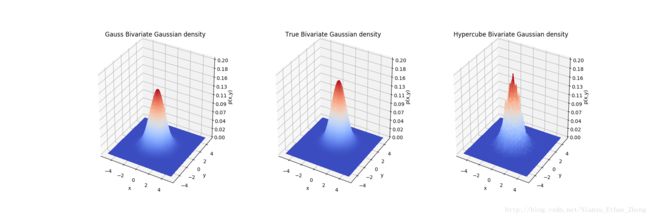
print(x_2Dgauss.shape)不同核函数对于pdf的估计效果不同
可以看到高斯核函数的概率密度估计过渡更加自然,而超维度的高斯估计则虽然相比较精度较高一些但是毛刺比较严重。
| 模型 | [0, 0] |
|---|---|
| 真实值 | 0.15915494309189535 |
| hypercube(h=0.3) | 0.15444444 |
| Gauss(h=0.3) | 0.14109129 |
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from matplotlib import cm
from matplotlib.ticker import LinearLocator, FormatStrFormatter
# Make data
mu_vec = np.array([0, 0])
cov_mat = np.array([[1, 0], [0, 1]])
x_2Dgauss = np.random.multivariate_normal(mu_vec, cov_mat, 10000)
# Pdf estimation
def pdf_multivaraible_gauss(x, mu, cov):
part1 = 1 / ((2 * np.pi) ** (len(mu)/2) * (np.linalg.det(cov)**(1/2)))
part2 = (-1/2) * (x-mu).T.dot(np.linalg.inv(cov)).dot((x-mu))
return float(part1 * np.exp(part2))
ph = parzen_estimation(x_2Dgauss, 0.3, mode='hypercube')
pg = parzen_estimation(x_2Dgauss, 0.3, mode='gauss')
print(pdf_multivaraible_gauss(np.array([[0], [0]]), np.array([[0], [0]]), cov_mat))
print(ph([[0, 0]]))
print(pg([[0, 0]]))
x = np.linspace(-5, 5, 100)
x, y = np.meshgrid(x, x)
zg = []
zt = []
zh = []
for i, j in zip(x.ravel(), y.ravel()):
zg.append(pg([[i, j]]))
zh.append(ph([[i, j]]))
zt.append(pdf_multivaraible_gauss(np.array([[i], [j]]), np.array([[0], [1]]), cov_mat))
zg = np.asarray(zg).reshape(100, 100)
zh = np.asarray(zh).reshape(100, 100)
zt = np.asarray(zt).reshape(100, 100)
# Plot the surface
fig = plt.figure(figsize=(18, 6))
ax1 = fig.add_subplot(131, projection='3d')
surf = ax1.plot_surface(x, y, zg, rstride=1, cstride=1,
cmap=cm.coolwarm, antialiased=False)
# Customize the z axis
ax1.zaxis.set_major_locator(LinearLocator(10))
ax1.zaxis.set_major_formatter(FormatStrFormatter('%.02f'))
ax1.set(zlim=[0, 0.2], xlabel='x', ylabel='y', zlabel='p(x,y)',
title="Gauss Bivariate Gaussian density")
# Re-Plot
ax2 = fig.add_subplot(132, projection='3d')
ax2.plot_surface(x, y, zt, rstride=1, cstride=1, cmap=cm.coolwarm, antialiased=False)
# Customize the z axis
ax2.zaxis.set_major_locator(LinearLocator(10))
ax2.zaxis.set_major_formatter(FormatStrFormatter('%.02f'))
ax2.set(zlim=[0, 0.2], xlabel='x', ylabel='y', zlabel='p(x,y)',
title="True Bivariate Gaussian density")
# Re-Plot
ax3 = fig.add_subplot(133, projection='3d')
ax3.plot_surface(x, y, zh, rstride=1, cstride=1, cmap=cm.coolwarm, antialiased=False)
# Customize the z axis
ax3.zaxis.set_major_locator(LinearLocator(10))
ax3.zaxis.set_major_formatter(FormatStrFormatter('%.02f'))
ax3.set(zlim=[0, 0.2], xlabel='x', ylabel='y', zlabel='p(x,y)',
title="Hypercube Bivariate Gaussian density")
# Add a color bar which maps values to colors.
fig.savefig("./Gauss_kernel_{}.png".format('Bivariate_Gaussian'))
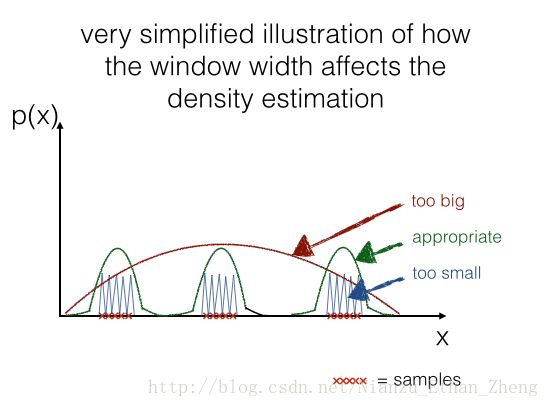
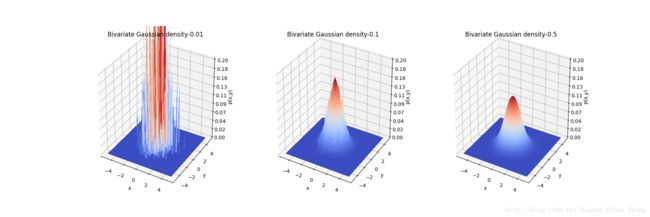
plt.show()不同核宽度对pdf估计的影响
可以看到当核宽度非常小(0.01)时, pdf已经不可控, 而当h=0.5, 估计效果又有所下降。因此如何能根据数据样本来确定核宽度大小,是一个非常重要的问题。
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from matplotlib import cm
from matplotlib.ticker import LinearLocator, FormatStrFormatter
# Make data
mu_vec = np.array([0, 0])
cov_mat = np.array([[1, 0], [0, 1]])
x_2Dgauss = np.random.multivariate_normal(mu_vec, cov_mat, 10000)
# Pdf estimation
def pdf_multivaraible_gauss(x, mu, cov):
part1 = 1 / ((2 * np.pi) ** (len(mu)/2) * (np.linalg.det(cov)**(1/2)))
part2 = (-1/2) * (x-mu).T.dot(np.linalg.inv(cov)).dot((x-mu))
return float(part1 * np.exp(part2))
pg1 = parzen_estimation(x_2Dgauss, 0.01, mode='gauss')
pg2 = parzen_estimation(x_2Dgauss, 0.1, mode='gauss')
pg3 = parzen_estimation(x_2Dgauss, 0.5, mode='gauss')
x = np.linspace(-5, 5, 100)
x, y = np.meshgrid(x, x)
zg = []
zh = []
zt = []
for i, j in zip(x.ravel(), y.ravel()):
zg.append(pg1([[i, j]]))
zh.append(pg2([[i, j]]))
zt.append(pg3([[i, j]]))
zg = np.asarray(zg).reshape(100, 100)
zh = np.asarray(zh).reshape(100, 100)
zt = np.asarray(zt).reshape(100, 100)
# Plot the surface
fig = plt.figure(figsize=(18, 6))
ax1 = fig.add_subplot(131, projection='3d')
surf = ax1.plot_surface(x, y, zg, rstride=1, cstride=1,
cmap=cm.coolwarm, antialiased=False)
# Customize the z axis
ax1.zaxis.set_major_locator(LinearLocator(10))
ax1.zaxis.set_major_formatter(FormatStrFormatter('%.02f'))
ax1.set(zlim=[0, 0.2], xlabel='x', ylabel='y', zlabel='p(x,y)',
title="Bivariate Gaussian density-0.01")
# Re-Plot
ax2 = fig.add_subplot(132, projection='3d')
ax2.plot_surface(x, y, zh, rstride=1, cstride=1, cmap=cm.coolwarm, antialiased=False)
# Customize the z axis
ax2.zaxis.set_major_locator(LinearLocator(10))
ax2.zaxis.set_major_formatter(FormatStrFormatter('%.02f'))
ax2.set(zlim=[0, 0.2], xlabel='x', ylabel='y', zlabel='p(x,y)',
title="Bivariate Gaussian density-0.1")
# Re-Plot
ax3 = fig.add_subplot(133, projection='3d')
ax3.plot_surface(x, y, zt, rstride=1, cstride=1, cmap=cm.coolwarm, antialiased=False)
# Customize the z axis
ax3.zaxis.set_major_locator(LinearLocator(10))
ax3.zaxis.set_major_formatter(FormatStrFormatter('%.02f'))
ax3.set(zlim=[0, 0.2], xlabel='x', ylabel='y', zlabel='p(x,y)',
title="Bivariate Gaussian density-0.5")
# Add a color bar which maps values to colors.
fig.savefig("./Gauss_kernel_{}.png".format('Gauss_width'))
plt.show()已知真实分布情况下
可以看到 h = 0.21529369369369367
当然大多数情况下并不知道真实的概率分布,那可以通过最大似然法来选择核宽度
首先介绍似然性
Likelihood
可能性其实就是所有采的样本在你所建立的模型中概率的乘积,让这个乘积最大就是最大似然法,而为了计算方便,采用对数的方法,将
乘积运算转换为加法运算。
对parzen_ll的理解
终于到了主题:
def theano_parzen(mu, sigma):
"""
Credit: Yann N. Dauphin
"""
x = T.matrix()
mu = theano.shared(mu)
a = ( x.dimshuffle(0, 'x', 1) - mu.dimshuffle('x', 0, 1) ) / sigma
E = log_mean_exp(-0.5*(a**2).sum(2))
Z = mu.shape[1] * T.log(sigma * numpy.sqrt(numpy.pi * 2))
return theano.function([x], E - Z)这个函数用于创造一个parzen的计算器,其中x的大小为2D向量,通过dimshuffule来变为Ax1xB 这样的向量,mu为图像的所有像素的值,sigma对于所有维都是固定的均为sigma。通过创造一个log_mean_exp函数计算平均值
def log_mean_exp(a):
"""
Credit: Yann N. Dauphin
"""
max_ = a.max(1)
return max_ + T.log(T.exp(a - max_.dimshuffle(0, 'x')).mean(1))定义好Parzen函数之后,通过get_nll函数获得似然值,其中的操作是每次输入一个batch_size,最后对各个batch_size求取平均值
def get_nll(x, parzen, batch_size=10):
"""
Credit: Yann N. Dauphin
"""
inds = range(x.shape[0])
n_batches = int(numpy.ceil(float(len(inds)) / batch_size))
times = []
nlls = []
for i in range(n_batches):
begin = time.time()
nll = parzen(x[inds[i::n_batches]])
end = time.time()
times.append(end-begin)
nlls.extend(nll)
if i % 10 == 0:
print i, numpy.mean(times), numpy.mean(nlls)
return numpy.array(nlls)其中可以追查mu的来源,mu ==samples ,于是在下面我们可以发现出mu的蛛丝马迹,reshape函数将三个通道的图片扁平为一维向量,而整个空间定格为整个图片的像素点数。
samples = model.generator.sample(args.num_samples).eval()
output_space = model.generator.mlp.get_output_space()
if 'Conv2D' in str(output_space):
samples = output_space.convert(samples, output_space.axes, ('b', 0, 1, 'c'))
samples = samples.reshape((samples.shape[0], numpy.prod(samples.shape[1:])))
del model
gc.collect()值得一提的是为了获得较好的估计值,他采用验证集来确定最大的似然值对应的sigma,验证集采用的是50000-60000的MNIST字体。
def cross_validate_sigma(samples, data, sigmas, batch_size):
lls = []
for sigma in sigmas:
print sigma
parzen = theano_parzen(samples, sigma)
tmp = get_nll(data, parzen, batch_size = batch_size)
lls.append(numpy.asarray(tmp).mean())
del parzen
gc.collect()
ind = numpy.argmax(lls)
return sigmas[ind]Parzen Window的公式大致对的上,明确了输入(对于MNIST)是60000x1x784的一个批次,大约10个,主要可能考虑到对于高达784的多变量高斯分布计算量必然很大。
然后考虑到likelihood的计算公式
将Parzen Window估计概率并应用于求解最大似然法,目前来说,对求解似然性于不是最好的办法,但是也没有更好的办法。其基本思路大致是,产生的样本通过高斯核Parzen窗口法计算出一个概率模型Pg(784维的高斯分布),然后估计测试样本的概率,从而计算出在该分布下的对数似然性,方差参数采用交叉验证来获得。
最大似然法
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from matplotlib import cm
from matplotlib.ticker import LinearLocator, FormatStrFormatter
# Make data
np.random.seed(2017)
mu_vec = np.array([0, 0])
cov_mat = np.array([[1, 0], [0, 1]])
x_2Dgauss = np.random.multivariate_normal(mu_vec, cov_mat, 10000)
x_test = np.random.multivariate_normal(mu_vec, cov_mat, 1000)
pg = parzen_estimation(x_2Dgauss, 0.01, mode='gauss')
sigma, nll = cross_validate_sigma(x_2Dgauss, x_test, np.linspace(1e-4, 1, 20), batch_size=100)
fig = plt.figure(figsize=(6, 6))
ax = fig.gca()
ax.plot(np.linspace(1e-4, 1, 20), nll, '-*r')
ax.set(xlabel='log likelihood', ylabel='\sigma value', title='NLL method ')
print(sigma)
fig.savefig('./{}.png'.format('NLL_method'))
plt.show()
# Pdf estimation
def pdf_multivaraible_gauss(x, mu, cov):
part1 = 1 / ((2 * np.pi) ** (len(mu)/2) * (np.linalg.det(cov)**(1/2)))
part2 = (-1/2) * (x-mu).T.dot(np.linalg.inv(cov)).dot((x-mu))
return float(part1 * np.exp(part2))
tr = pdf_multivaraible_gauss(np.array([[0], [0]]), np.array([[0], [0]]), cov_mat)
errs = []
sigmas = np.linspace(1e-4, 1, 1000)
for sigma in sigmas:
pg = parzen_estimation(x_2Dgauss, sigma, mode='gauss')
err = np.abs(pg(np.array([[0, 0]]))-tr)
errs.append(err)
fig = plt.figure(figsize=(6, 6))
ax = fig.gca()
ax.plot(sigmas, errs, '-*b')
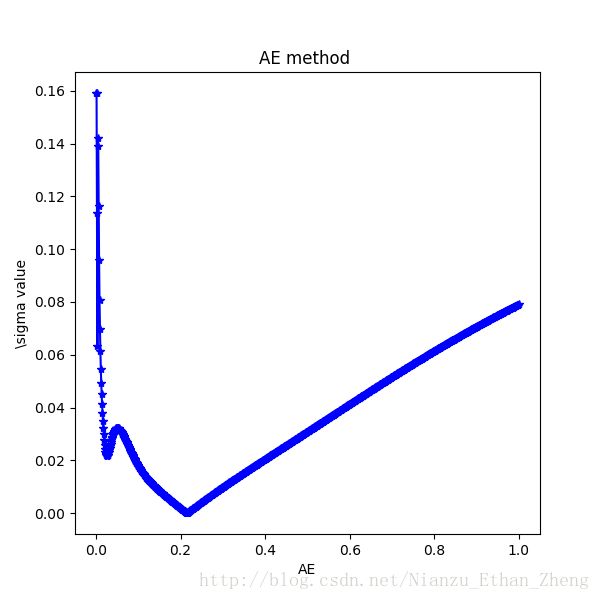
ax.set(xlabel='AE', ylabel='\sigma value', title='AE method ')
ind = np.argmin(errs)
print(sigmas[ind])
fig.savefig('./{}.png'.format('AE_method'))
plt.show()可以看到在h = 0.21060526315789474, 附近似然性取得最大值
| 方法 | 最优值 |
|---|---|
| 最大似然法 | 0.21060526315789474 |
| 误差法 | 0.21529369369369367 |
可以看到极大似然法可以有效估计核宽度。
Parzen window 技术的优缺点:
Parzen window技术作为一种非参数方法,有着和其他方法一样的缺点,就是每次估计需要整个样本参与运算,而并非是参数估计,只是带入参数计算。
几个挑战:
1) 数据样本的大小, Parzen window的计算复杂度为 n2d n 2 d , 其中 n n 为训练样本数目, 而 d d 为样本维度。
2) 合适的核宽度的选择, 一般来说:
3) 核函数的选择
4) 应用于两个方面:Bayes估计与互信息的求取