使用R语言进行决策树建模
关于决策树的理解及自定义代码实现请参考我的另一个博客:
数据挖掘常用算法理解与R语言实现(系列待完成)
本次技能点:
训练集和测试集的选取
决策树构建与减值
决策树的print和plot
预测值与实际值关系描述
用到的函数或包:ctree(party包),rpart(rpart包),set.seed、sample、predict、cptable、cp属性、prune、xerror属性。
代码:
#一、使用part包构建决策树
#我们将在iris数据集上演示使用party包构建决策树的方法,在iris数据集中,茎叶的长宽都能来预测花的种类
#我们将使用party包中的ctree()用来建立决策树,用predict()来预测新的数据
#---设置学习集和测试集
str(iris)
set.seed(1234)
ind<-sample(c(1:2),nrow(iris),replace = T,prob = c(0.7,0.3))
#这里解释set.seed的含义,在产生一个随机样本之前设置set.seed(n) n代表标号,下次再调用set.seed(n)的时候,随机样本
#产生函数会生成一样的结果,以方便结果的重现,比如我们执行以下代码,会发现ind2==ind
#set.seed(1234)
#ind2<-sample(2,nrow(iris),replace = TRUE,prob = c(0.7,0.3))
#sample函数 即产生随机样本,用法为sample(x,size,replace,prob) x代表从哪些集合中抽取随机数,size代表抽取几个随机数
trainData<-iris[ind==1,]
testData<-iris[ind==2,]
#---使用party包中的ctree()函数来产生决策树
#先定义公式,Species作为因变量,其余作为自变量
myFormula<-Species~Sepal.Length+Sepal.Width+Petal.Length+Petal.Width
install.packages("party")
library(party)
irisTree<-ctree(myFormula,data=trainData)
table(predict(irisTree),trainData$Species)
plot(irisTree,main = "prediction tree")
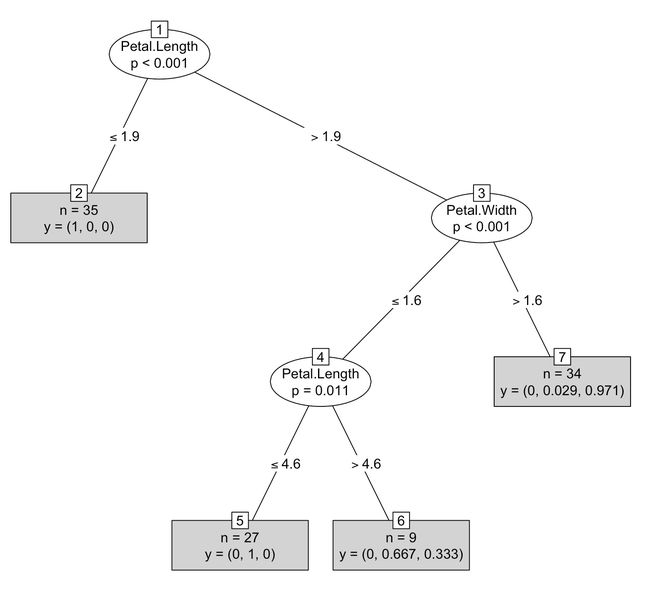
plot(irisTree,type="simple")
#在简化版的决策树图中,n代表在该节点中的实例个数,y代表各个种类分布情况,比如第二个节点中有35个实例,且都属于第一类
#使用构建好的决策树进行预测
testPre<-predict(irisTree,testData)
#检验预测结果,发现只有两个是False,即50个测试集预测错了2个
testData$Species==testPre
#二、使用rpart包构建决策树
#我们将用rpart包中的rpart函数进行构建决策树,然后同样用predict函数进行预测
install.packages("mboost")
data("bodyfat",package = "TH.data")
str(bodyfat)
attributes(bodyfat)
dim(bodyfat)
library(rpart)
set.seed(12345)
ind2<-sample(2,nrow(bodyfat),replace=T,prob = c(0.7,0.3))#sample里的2即代表c(1:2)的意思
trainData2<-bodyfat[ind2==1,]
testData2<-bodyfat[ind2==2,]
myFormula2<-DEXfat~age+waistcirc+hipcirc+elbowbreadth+kneebreadth
bodyfatTree<-rpart(myFormula2,data=trainData2,control = rpart.control(minsplit = 10))
print(bodyfatTree)
attributes(bodyfatTree)
par(mar = c(4,4,4,4)) #设置画图区域的离四周边界的距离
plot(bodyfatTree)
text(bodyfatTree,use.n = T) #添加节点文字信息
print(bodyfatTree$cptable)
#选择具有最小误差的决策树,通过决策树对象的cptable中的xerror属性最小来选择。
opt<-which.min(bodyfatTree$cptable[,"xerror"])
#取出所选对象的cp的值
cp<-bodyfatTree$cptable[opt,"CP"]
#决策树的剪枝
bodyfat_prune<-prune(bodyfatTree,cp=cp) #通过cp值选择决策树,最终的决策树对象赋值给bodyfat_prune
print(bodyfat_prune)
plot(bodyfat_prune)
text(bodyfat_prune,use.n = T)
bodyfat.pre<-predict(bodyfat_prune,newdata = testData2)
xlim<-range(bodyfat$DEXfat)#设置x轴方位
plot(bodyfat.pre~DEXfat,data=testData2,xlab="Oberserved",ylab="pre")
abline(a=0,b=1)
#补充知识:
# 1、CP参数定义
#
# cp: complexity parameter复杂性参数,用来修剪树的.
#
# 当决策树复杂度超过一定程度后,随着复杂度的提高,测试集的分类精确度反而会降低。因此,建立的决策树不宜太复杂,
# 需进行剪枝。该剪枝算法依赖于复杂性参数cp,cp随树复杂度的增加而减小,当增加一个节点引起的分类精确度变化量小于树
# 复杂度变化的cp倍时,则须剪去该节点。故建立一棵既能精确分类,又不过度适合的决策树的关键是求解一个合适的cp值
# 。一般选择错判率最小值对应的cp值来修树.
# 2、CP选择问题。
#
# 建立树模型要权衡两方面问题,一个是要拟合得使分组后的变异较小,另一个是要防止过度拟合,而使模型的误差过大,
# 前者的参数是CP,后者的参数是Xerror。所以要在Xerror最小的情况下,也使CP尽量小。
# 3、关于abilne()
# 函数lines()其作用是在已有图上加线,命令为lines(x,y),其功能相当于plot(x,y,type="1")
# 函数abline()可以在图上加直线,其使用方法有四种格式。
# (1)abline(a,b)
# 表示画一条y=a+bx的直线
# (2)abline(h=y)
# 表示画出一条过所有点得水平直线
# (3)abline(v=x)
# 表示画出一条过所有点的竖直直线
# (4)abline(lm.obj)
# 表示绘出线性模型得到的线性方程相关结果: