天池大数据竞赛——天池精准医疗大赛人工智能辅助糖尿病遗传风险预测赛后总结
天池大数据竞赛官方网址(链接)
天池精准医疗大赛是我第一次正式参加与学习的数据竞赛,在这十几天的过程中,学习到很多参与这些数据竞赛的技巧和知识,虽然结果并不理想,但是总归是学习到不少。这篇文章也是梳理总结一下这段时间以来的经验和技术。在此需要感谢天池技术圈里的大佬共享的知识资源和Kaggle网站中共享的一些学习思路和资源。具体参考链接请见Reference。
一、赛题背景介绍
1.赛制与赛题背景
天池精准医疗大赛的赛题主页(链接)
总结起来的就是,通过给出一定数量的个人的体检数据,以血糖值为目标来建立相关的模型实现血糖预测功能。用人工智能的方法和思想处理、分析、解读和应用糖尿病相关大数据,设计高精度,高效,解释性强的算法来挑战糖尿病精准预测。
生命科学特别是基因科技已经广泛而且深刻影响到每个人的健康生活,于此同时,基因遗传史无前例的用一种全新的视角解读生命和探究疾病本质。数据挖掘能够处理分析海量医疗健康数据,通过认知分析获取信息,服务于政府、健康医疗机构、制药企业及患者。糖尿病作为一种常见慢性疾病,目前无法根治,但却能通过科学有效的干预、预防和治疗,来降低发病率和提高患者的生活质量。借助于数据挖掘对糖尿病遗传数据的研究,希望用数据挖掘的方法和思想处理、分析、解读和应用糖尿病相关大数据,通过设计高精度,高效,且解释性强的算法来挑战糖尿病精准预测这一难题,为精准医疗提供有力的技术支撑,为糖尿病的深入研究提供新思路。
整个比赛下来,涉及的内容可能会比较多,所以我计划分为两篇或者三篇来写。而这一部分就主要介绍数据的基本情况及其基本统计信息,结合删除离群值、平滑处理、多项式分解、特征工程等方式完成数据处理。然后在下一部分介绍三种应用于本课题的数据挖掘算法及一种独特的单模型融合方法,结合相应的评价指标完成本文提出的模型的评估工作。
2.赛题数据
比赛的初级阶段分为A榜和B榜,两者都划分为训练集和测试集,训练集中包含年龄、性别、各项体检指标以及预测目标血糖值。而测试集相对于训练集则缺少了对应的血糖值,也就是我们所期望预测到的值。
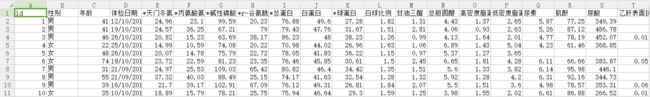
数据集中包含两个部分:训练集文件和测试集文件,部分字段名已经做脱敏处理,训练集中包含37个医学指标作为基本数据特征,每个文件的第一行是字段名,之后的每一行代表的是一个个体。文件共包含42个字段,包含数值型、字符型、日期型等众多数据类型,详细的字段名下数据类型请见表1,部分字段内容在部分人群中有缺失,其中第一列为个体ID号。训练文件的最后一列为标签列,即需要预测的目标血糖值。
数据的大致格式如下图所示,选取其中十个人的数据的一部分作为展示。
二、数据统计信息
1.数据缺失比例统计
为了实现对数据集的预处理与特征工程,我们需要了解数据集的统计信息。本文对数据集做出了以下的统计:
数据的缺失比例,以数据的每个基本特征为单位,统计各列数据的缺失值的数目,并与该列数据总数据量相比,统计每个基本特征的缺失比例,便于下一步删除缺失比例较高的基本特征。数据集的各个基本特征缺失比例下图所示。
由上图我们可以得出,乙肝表面抗原、乙肝表面抗体、乙肝核心抗体、乙肝e抗原、乙肝e抗体这五个基本特征缺失比例超过70%,相较于其他基本特征,缺失的比例非常大。在进一步探究这五个基本特征的影响权重后,可以考虑进行删除。
1.数据特征影响权重
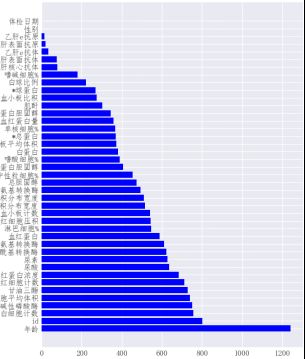
数据特征影响权重,以LightGBM为核心算法,通过相关函数可以实现每个输入特征与预测结果之间的关联性,也就是数据特征对于预测结果的影响权重。通过这一比较结果,我们可以获知对于预测模型无效的数据特征,同时根据该结果,调整各个单模型的算法的基本参数,为模型调优提供参考依据。数据集的基本特征影响权重如下图所示。
由上图得知,体检日期与性别对于模型的预测并无实际的关联,结合医学常识也可证实这一观点,样本的血糖值与体检日期和性别并无关联。因此在模型的搭建的过程中,为了算法的轻量化与运算速度的提升,体检日期与性别这两类基本特征需要进行剔除。
3.基本特征与血糖值分布
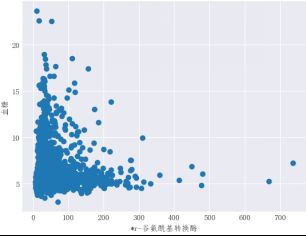
基本特征与血糖值的分布,在数据处理前,了解各个基本特征与血糖分布的整体情况,为特征工程提供直观的参考依据。对于训练集,将血糖值为Y轴坐标,各个基本特征为X轴坐标,做出各个基本特征与血糖值的分布情况,下图为R-谷氨酰基转换酶和血糖分布情况。
由上图可知,r-谷氨酰基转换酶和血糖分布并非服从正态分布或者满足某种分布规律,同时也可以发现其中包含离群值,但是由于数据样本较少,对于离群值的处理还需要进一步的讨论。
4.特征先验高斯分布
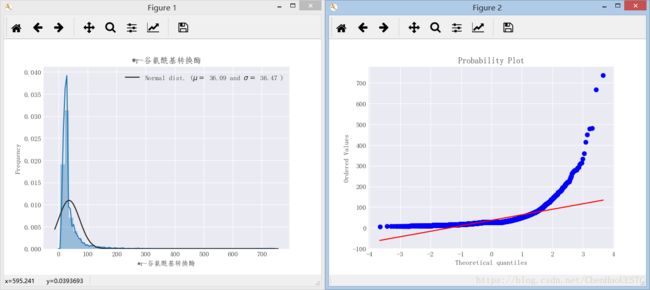
基本特征的先验高斯分布,是以先验的高斯分布去绘制血糖值与输入的基本特征之间的分布关系,在理想状态下,分布曲线应与相应的推理条件吻合,绘制基本特征的先验高斯分布,有助于后期数据的平滑处理。r-谷氨酰基转换酶和血糖的先验高斯分布图如图4所示,由图观察可以轻易得出,其概率分布与理想情况存在较大的偏离,因此需要对数据集的基本特征做平滑处理。
5.特征关联热力图
特征关联热力图,采用相应的数据处理库函数可以采用热力图的形式展示数据的基本特征关联情况。数据集的基本特征关联热力图如下图所示,图中的每个像素的颜色深浅代表横纵轴上的特征的关联程度,数据集的基本特征关联热力图有利于特征工程的选取。
特征关联热力图采用的是Python中seaborn库进行操作,相关介绍请见seaborn绘制热力图坐标标签。
以上部分处理操作,参考自天池比赛技术圈麻婆豆腐同学的分享,以及Kaggle中房价预测这一比赛的分享内容。网址请见Reference[2]。
三、数据预处理
结合Section 2中的数据集的统计信息,本文中对于数据集的预处理操作包含以下四种:去除缺失比例非常高的基本特征、去除对模型预测无影响的基本特征和离群值、处理缺失值和对数据集的基本特征做平滑处理。
1.去除高缺失比例数据
去除缺失比例非常高的基本特征,是指删除缺失比例超过70%的数据,由Section 2中可知乙肝表面抗原、乙肝表面抗体、乙肝核心抗体、乙肝e抗原、乙肝e抗体这五个基本特征缺失比例超过70%,且这五种基本特征对于预测模型的影响权重较小,没有合理的办法对这些缺失的数据进行填充,因此选择将这些缺失比例很高的基本特征剔除。
2.去除离群值与无意义值
3.缺失值处理
处理缺失值,是指对于数据集中所有缺失的数据采取恰当的方式进行填充的过程。在各种实用的数据库中,数据缺失的情况经常发全甚至是不可避免的。因此,在大多数情况下,数据集是不完备的,或者说存在某种程度的不完备。数据缺失在数据挖掘的过程中,会造成了以下影响:首先,系统丢失了大量的有用信息;第二,系统中所表现出的不确定性更加显著,系统中蕴涵的确定性成分更难把握;第三,包含空值的数据会使挖掘过程陷入混乱,导致不可靠的输出。对于数据缺失这一问题,数据挖掘领域有以下几种处理方法:特殊值填充、平均值填充、中位数填充、K最近距离邻法(K-means clustering)、回归方程法(Regression)、期望值最大化法(Expectation maximization)等,结合数据集的实际分布情况与实验论证,文章选取中位数填充的方法实现数据集的缺失值处理。
4.特征平滑处理
对数据集的基本特征做平滑处理,是由于数据集的基础特征的分布与推理条件中先验高斯分布之间存在较大的差异,如果数据集中的特征是不平滑的,这对于模型的拟合来说会有负面影响,同时有的噪声数据会影响拟合的函数的准确性,因此在特征工程工作和数据挖掘之前,先要对数据集进行平滑处理操作。值得一提的是,并非所有的基本特征都需要进行平滑处理,如淋巴细胞这一基本特征,其基本分布与推理条件中的先验高斯分布基本吻合,如下图所示。
四、数据特征工程
特征是数据中抽取出来的对结果预测有用的信息,可以是文本或者数据。特征工程是使用专业背景知识和技巧处理数据,使得特征能在机器学习算法上发挥更好的作用的过程。过程包含了特征提取、特征构建、特征选择等模块。特征工程的目的是筛选出更好的特征,获取更好的训练数据。因为好的特征具有更强的灵活性,可以用简单的模型做训练,更可以得到优秀的结果。优越的特征可以缩短模型参数调优的过程,降低模型的复杂度使模型趋于简单。
本比赛中,由于源数据集的基本特征为医学特征,难以构建合理的新特征,本文选用多项式特征的方法完成特征工程部分工作。采用sklearn中专门产生多项式的函数PolynomialFeature产生相互影响的特征集。多项式特征的方法产生相互影响的数据集的过程可以用图8来作为演示,由本身的输入矩阵产生一个伪特征矩阵,由此可以用线性回归的方式来做非线性回归的预测,通过产生高维度的特征空间。
五、代码部分
赛题数据请见天池数据竞赛官网,根据实际情况适当调整代码。
'''
Author:chenhao
Date: Jan 19 ,2017
Description: Data Visualization and Data Characteristics
'''
import time
import datetime
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import lightgbm as lgb
from dateutil.parser import parse
from sklearn.cross_validation import KFold
from sklearn.metrics import mean_squared_error
from pylab import mpl
from scipy import stats
from scipy.stats import norm, skew
import warnings
def ignore_warn(*args , **kwargs):
pass
warnings.warn = ignore_warn
pd.set_option('display.float_format',lambda x:'{:.3f}'.format(x))
color = sns.color_palette()
sns.set_style('darkgrid')
data_path = 'data/'
train = pd.read_csv(data_path + 'd_train_20180102.csv', encoding='gb2312')
test = pd.read_csv(data_path + 'd_test_A_20180102.csv', encoding='gb2312')
train['体检日期'] = (pd.to_datetime(train['体检日期']) - parse('2017-09-10')).dt.days
test['体检日期'] = (pd.to_datetime(test['体检日期']) - parse('2017-09-10')).dt.days
train['性别'] = train['性别'].map({'男': 1, '女': 0, '??':0})
test['性别'] = test['性别'].map({'男': 1, '女': 0})
train_fill = train.drop(['id','性别','血糖'],axis=1)
train_fill.fillna(train_fill.median(axis=0), inplace=True)
data = pd.concat([train,test],axis=0)
null_percentage = data.isnull().sum()/len(data)
print ('The null data percentage is:',null_percentage)
mpl.rcParams['font.sans-serif'] = ['FangSong']
null_percentage = null_percentage.reset_index()
null_percentage.columns = ['column_name','column_value']
ind = np.arange(null_percentage.shape[0])
fig , ax = plt.subplots(figsize = (6, 8))
rects = ax.barh(ind,null_percentage.column_value.values,color='red')
ax.set_yticks(ind)
ax.set_yticklabels(null_percentage.column_name.values,rotation='horizontal')
ax.set_xlabel("各基本特征缺失数据值")
plt.show()
def make_feat(train, test):
train_id = train.id.values.copy()
test_id = test.id.values.copy()
data = pd.concat([train, test])
data['性别'] = data['性别'].map({'男': 1, '女': 0})
data['体检日期'] = (pd.to_datetime(data['体检日期']) - parse('2017-9-10')).dt.days
data.fillna(data.median(axis=0), inplace=True)
train_feat = data[data.id.isin(train_id)]
test_feat = data[data.id.isin(test_id)]
return train_feat, test_feat
importance = train.drop(['血糖'],axis=1)
importance_name = importance.columns
train_feat, test_feat = make_feat(train, test)
predictors = [f for f in test_feat.columns if f not in ['血糖']]
def evalerror(pred, df):
label = df.get_label().values.copy()
score = mean_squared_error(label, pred) * 0.5
return ('0.5mse', score, False)
print('开始训练...')
params = {
'learning_rate': 0.01,
'boosting_type': 'gbdt',
'objective': 'regression',
'metric': 'mse',
'sub_feature': 0.7,
'num_leaves': 60,
'colsample_bytree': 0.7,
'feature_fraction': 0.7,
'min_data': 100,
'min_hessian': 1,
'verbose': -1,
}
print('开始CV 5折训练...')
t0 = time.time()
train_preds = np.zeros(train_feat.shape[0])
test_preds = np.zeros((test_feat.shape[0], 5))
kf = KFold(len(train_feat), n_folds=5, shuffle=True, random_state=520)
for i, (train_index, test_index) in enumerate(kf):
print('第{}次训练...'.format(i))
train_feat1 = train_feat.iloc[train_index]
train_feat2 = train_feat.iloc[test_index]
lgb_train1 = lgb.Dataset(train_feat1[predictors], train_feat1['血糖'], categorical_feature=['性别'])
lgb_train2 = lgb.Dataset(train_feat2[predictors], train_feat2['血糖'])
gbm = lgb.train(params,
lgb_train1,
num_boost_round=3000,
valid_sets=lgb_train2,
verbose_eval=100,
feval=evalerror,
early_stopping_rounds=100)
feat_imp = pd.Series(gbm.feature_importance(), index=predictors).sort_values(ascending=False)
train_preds[test_index] += gbm.predict(train_feat2[predictors])
test_preds[:, i] = gbm.predict(test_feat[predictors])
print('线下得分: {}'.format(mean_squared_error(train_feat['血糖'], train_preds) * 0.5))
print('CV训练用时{}秒'.format(time.time() - t0))
print(feat_imp)
mpl.rcParams['font.sans-serif'] = ['FangSong']
feat_imp = feat_imp.reset_index()
feat_imp.columns = ['column_name','column_value']
ind = np.arange(feat_imp.shape[0])
fig , ax = plt.subplots(figsize = (6,8))
rects = ax.barh(ind,feat_imp.column_value.values,color='blue')
ax.set_yticks(ind)
ax.set_yticklabels(feat_imp.column_name.values,rotation='horizontal')
ax.set_xlabel("各个基本特征影响权重")
plt.show()
mpl.rcParams['font.sans-serif'] = ['FangSong']
mpl.rcParams['axes.unicode_minus'] = False
fig , ax = plt.subplots()
ax.scatter(x=train_fill['*天门冬氨酸氨基转换酶'],y=train['血糖'])
plt.ylabel('血糖')
plt.xlabel('*天门冬氨酸氨基转换酶')
soft , b = stats.boxcox(train_fill['*天门冬氨酸氨基转换酶'])
fig , ax = plt.subplots()
ax.scatter(x= soft,y=train['血糖'])
plt.ylabel('血糖')
plt.xlabel('*天门冬氨酸氨基转换酶')
print(soft)
print(b)
plt.show(1)
plt.show(2)
mpl.rcParams['font.sans-serif'] = ['FangSong']
mpl.rcParams['axes.unicode_minus'] = False
dist = sns.distplot(train_fill['嗜碱细胞%'],fit=norm)
(mu,sigma) = norm.fit(train_fill['嗜碱细胞%'])
print('\n mu = {:.2f} and sigma = {:.2f}\n'.format(mu, sigma))
plt.legend(['Normal dist. ($\mu=$ {:.2f} and $\sigma=$ {:.2f} )'.format(mu, sigma)], loc='best')
plt.ylabel('Frequency')
plt.title('嗜碱细胞%')
fig = plt.figure()
res = stats.probplot(train_fill['嗜碱细胞%'], plot=plt)
plt.show()
soft , b = stats.boxcox(train_fill['甘油三酯'])
mpl.rcParams['font.sans-serif'] = ['FangSong']
mpl.rcParams['axes.unicode_minus'] = False
sns.distplot(soft,fit=norm)
(mu,sigma) = norm.fit(soft)
print('\n mu = {:.2f} and sigma = {:.2f}\n'.format(mu, sigma))
plt.legend(['Normal dist. ($\mu=$ {:.2f} and $\sigma=$ {:.2f} )'.format(mu, sigma)], loc='best')
plt.ylabel('Frequency')
plt.title('血糖分布')
fig = plt.figure()
res = stats.probplot(soft, plot=plt)
plt.show()
mpl.rcParams['font.sans-serif'] = ['FangSong']
mpl.rcParams['axes.unicode_minus'] = False
corrmat = train.corr()
f,ax = plt.subplots(figsize=(15,12))
ax.set_xticklabels(corrmat,rotation='horizontal')
sns.heatmap(corrmat, vmax =0.9,square=True)
label_y = ax.get_yticklabels()
plt.setp(label_y , rotation = 360)
label_x = ax.get_xticklabels()
plt.setp(label_x , rotation = 90)
plt.show()
mpl.rcParams['font.sans-serif'] = ['FangSong']
mpl.rcParams['axes.unicode_minus'] = False
sns.set()
cols = ['年龄','白细胞计数','甘油三酯','红细胞平均血红蛋白浓度','尿素','尿酸']
sns.pairplot(train_fill[cols],size=2.5)
plt.show()
a = train.loc[train[train['红细胞平均血红蛋白浓度'] > 425].index]
a = a['血糖']
print(a)
mpl.rcParams['font.sans-serif'] = ['FangSong']
mpl.rcParams['axes.unicode_minus'] = False
fig , ax = plt.subplots()
ax.scatter(x=train_fill['*天门冬氨酸氨基转换酶'],y=train['血糖'])
plt.ylabel('血糖')
plt.xlabel('*天门冬氨酸氨基转换酶')
soft , b = stats.boxcox(train_fill['*天门冬氨酸氨基转换酶'])
fig , ax = plt.subplots()
ax.scatter(x= soft,y=train['血糖'])
plt.ylabel('血糖')
plt.xlabel('*天门冬氨酸氨基转换酶')
print(soft)
print(b)
plt.show(1)
plt.show(2)
这一部分大致介绍了数据的基本情况及其基本统计信息,结合删除离群值、平滑处理、多项式分解、特征工程等方式完成数据处理。然后在下一部分介绍三种应用于本课题的数据挖掘算法及一种独特的单模型融合方法,结合相应的评价指标完成本文提出的模型的评估工作。吃完饭后继续写。。= =
Reference
[1] 人工智能辅助糖尿病遗传风险预测所有的参考文献,网址链接。
[2] 寒武纪之糖尿病血糖值预测的数据初探(肆),网址链接。