基于K-Means模型的数据聚类(复习12)
本文是个人学习笔记,内容主要基于K-means聚类模型KMeans对手写体数据集进行聚类分组。
无监督学习(Unsupervised Learning)着重于发现数据本身的分布特点,发现数据的“群落”,寻找“离群”的样本。
K-means算法:预先设定聚类的个数,然后不断更新聚类中心,目标是让所有数据点到其所属聚类中心距离的平方和趋于稳定。
K-means算法执行的4个阶段:(1)随机布设K个特征空间内的点作为初始的聚类中心(2)对于每个待归类的数据,从K个聚类中心中寻找距离最近的一个,并把该数据标记为从属于这个聚类中心(3)在所有的数据都被标记聚类中心之后,根据这些数据新分配的类簇,重新计算出K个聚类中心(4)一轮结束后,所有数据点从属的聚类中心与上一次分配的类簇一致,则迭代停止,否则回到步骤(2)继续循环。
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
digits_train=pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/optdigits/optdigits.tra',header=None)
digits_test=pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/optdigits/optdigits.tes',header=None)X_train=digits_train[np.arange(64)] #从训练集和测试机上均分理处64惟的像素特征和1维的数字目标
y_train=digits_train[64]
X_test=digits_test[np.arange(64)]
y_test=digits_test[64]from sklearn.cluster import KMeans
kmeans=KMeans(n_clusters=10) #初始化KMeans模型,设置聚类中心数量为10
kmeans.fit(X_train)
y_pred=kmeans.predict(X_test)from sklearn import metrics
print(metrics.adjusted_rand_score(y_test,y_pred))
#Output:0.666071810158评估聚类性能的方法:(1)如果被用来评估的数据本身带有正确的类别信息,则利用Adjusted Rand Index(ARI),ARI与分类问题中计算准确性的方法类似,兼顾了类簇无法和分类标记一一对应的问题。(2)如果被用来评估的数据没有所属类别,则使用轮廓系数(Silhouette Coefficient)来度量聚类结果的质量,评估聚类的效果。轮廓系数同时兼顾了聚类的凝聚都和分离度,取值范围是[-1,1],轮廓系数越大,表示聚类效果越好。
轮廓系数的具体计算步骤:
[a.]对于已聚类数据中第i个样本 xi ,计算 xi 与其同一类簇内的所有其他样本距离的平均值,记作 ai ,用于量化簇内的凝聚度
[b.]选取 xi 外的一个簇 b ,计算 xi 与簇 b 中所有样本的平均距离,遍历所有其他簇,找到最近的这个平均距离,记作 bi ,用于量化簇之间分离度
[c.]对于样本 xi ,轮廓系数为 sci=bi−aimax(bi,ai)
[d.]最后,对所以样本 X 求出平均值,即为当前聚类结果的整体轮廓系数。
import numpy as np
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_score
import matplotlib.pyplot as plt
plt.rcParams['figure.figsize']=(10,10)
plt.subplot(3,2,1)
x1=np.array([1,2,3,1,5,6,5,5,6,7,8,9,7,9]) #初始化原始数据
x2=np.array([1,3,2,2,8,6,7,6,7,1,2,1,1,3])
X=np.array(list(zip(x1,x2))).reshape(len(x1),2)
plt.xlim([0,10])
plt.ylim([0,10])
plt.title('Instances')
plt.scatter(x1,x2)
colors=['b','g','r','c','m','y','k','b']
markers=['o','s','D','v','^','p','*','+']
clusters=[2,3,4,5,8]
subplot_counter=1
sc_scores=[]
for t in clusters:
subplot_counter +=1
plt.subplot(3,2,subplot_counter)
kmeans_model=KMeans(n_clusters=t).fit(X) #KMeans建模
for i,l in enumerate(kmeans_model.labels_):
plt.plot(x1[i],x2[i],color=colors[l],marker=markers[l],ls='None')
plt.xlim([0,10])
plt.ylim([0,10])
sc_score=silhouette_score(X,kmeans_model.labels_,metric='euclidean') #计算轮廓系数
sc_scores.append(sc_score)
plt.title('k=%s,silhouette coefficient=%0.03f'%(t,sc_score))
plt.figure()
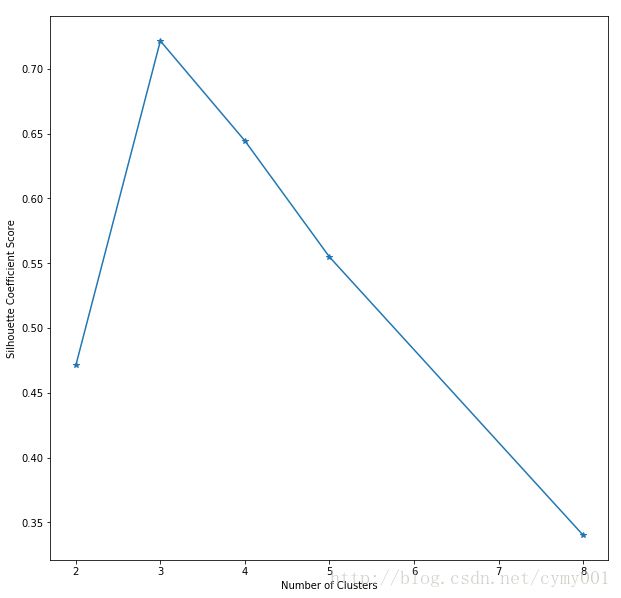
plt.plot(clusters,sc_scores,'*-') #绘制类簇数量与对应轮廓系数关系
plt.xlabel('Number of Clusters')
plt.ylabel('Silhouette Coefficient Score')
plt.show() K-means算法的两大缺点:(1)容易收敛到局部最优解(2)需要预先设定簇的数量。
利用“肘部观察法”可以粗略地估计相对合理的聚类个数。K-means模型最终期望*所有数据点到其所属的类簇距离的平方和趋于稳定,所以可以通过观察这个值随着K的走势来找出最佳的类簇数量。理想条件下,这个折线在不断下降并且趋于平缓的过程中会有斜率的拐点,这表示从这个拐点对应的K值开始,类簇中心的增加不会过于破坏数据聚类的结构*。
import numpy as np
from sklearn.cluster import KMeans
from scipy.spatial.distance import cdist
import matplotlib.pyplot as plt
cluster1=np.random.uniform(0.5,1.5,(2,10))
cluster2=np.random.uniform(5.5,6.5,(2,10))
cluster3=np.random.uniform(3,4,(2,10))
X=np.hstack((cluster1,cluster2,cluster3)).T
plt.scatter(X[:,0],X[:,1])
plt.xlabel('x1')
plt.ylabel('x2')
plt.show()K=range(1,10)
meandistortions=[]
for k in K:
kmeans=KMeans(n_clusters=k)
kmeans.fit(X)
meandistortions.append(sum(np.min(cdist(X,kmeans.cluster_centers_,'euclidean'),axis=1))/X.shape[0])
plt.plot(K,meandistortions,'bx-')
plt.xlabel('k')
plt.ylabel('Average Dispersion')
plt.title('Selecting k with the Elbow Method')
plt.show()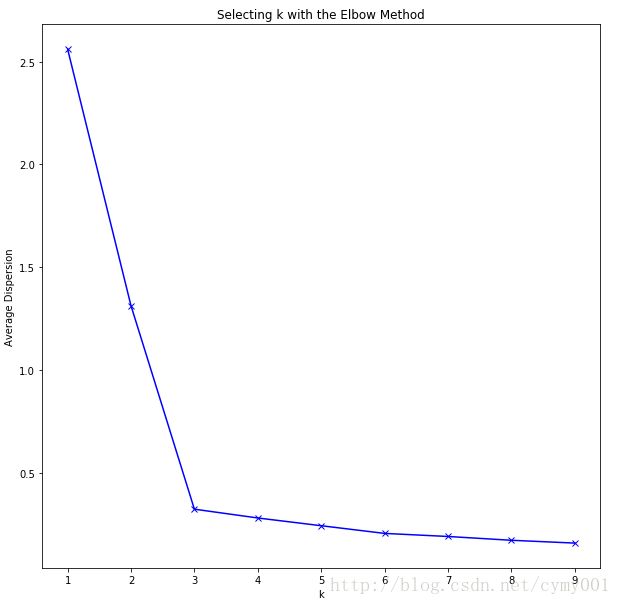
从上图可见,类簇数量从1降到2再降到3的过程,更改K值让整体聚类结构有很大改变,这意味着新的聚类数量让算法有更大的收敛空间,这样的K值不能反映真实的类簇数量。而当K=3以后再增大K,平均距离的下降速度显著变缓慢,这意味着进一步增加K值不再会有利于算法的收敛,同时也暗示着K=3是相对最佳的类簇数量。