今天任务,学习 http://www.bio-info-trainee.com/3533.html 我这个教程,自己写推文,我是如何拿到 KEGG数据库的 hsa04650 Natural killer cell mediated cytotoxicity这个通路的所有基因名字的 。
这是今天的任务,在谷歌搜索到了相关的内容
KEGG数据库的rest API(附带R语言小技巧) https://cloud.tencent.com/developer/article/1078458,很容易解决,但是毕竟是菜鸟土豆,所以要一切从头开始。首先,根据Jimmy老师提示KEGGREST包含KEGG通路里的所有基因,查找到KEGGREST包的说明书(https://bioconductor.org/packages/release/bioc/vignettes/KEGGREST/inst/doc/KEGGREST-vignette.html),了解这个包的使用。
1.安装和加载KEGGREST包
if(F){
source("http://bioconductor.org/biocLite.R")
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
options(BioC_mirror="http://mirrors.ustc.edu.cn/bioc/")#修改镜像,安装会加速
BiocManager::install("KEGGREST")
}
library(KEGGREST)
#若是上面安装不了,可以采用下面方法安装
library(devtools)
source("https://bioconductor.org/biocLite.R")
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
BiocInstaller::biocLite('KEGGREST')
2. 熟悉这个包的功能
listDatabases()#显示KEGGREST所包含的数据内容, 可以在进一步查询中使用这些数据。
listDatabases()
## [1] "pathway" "brite" "module" "ko" "genome" "vg"
## [7] "ag" "compound" "glycan" "reaction" "rclass" "enzyme"
## [13] "disease" "drug" "dgroup" "environ" "genes" "ligand"
## [19] "kegg"
可以使用以下keggList()功能获取KEGG中可用的生物列表:
org <- keggList("organism")
head(org)
## T.number organism species
## [1,] "T01001" "hsa" "Homo sapiens (human)"
## [2,] "T01005" "ptr" "Pan troglodytes (chimpanzee)"
## [3,] "T02283" "pps" "Pan paniscus (bonobo)"
## [4,] "T02442" "ggo" "Gorilla gorilla gorilla (western lowland gorilla)"
## [5,] "T01416" "pon" "Pongo abelii (Sumatran orangutan)"
## [6,] "T03265" "nle" "Nomascus leucogenys (northern white-cheeked gibbon)"
## phylogeny
## [1,] "Eukaryotes;Animals;Vertebrates;Mammals"
## [2,] "Eukaryotes;Animals;Vertebrates;Mammals"
## [3,] "Eukaryotes;Animals;Vertebrates;Mammals"
## [4,] "Eukaryotes;Animals;Vertebrates;Mammals"
## [5,] "Eukaryotes;Animals;Vertebrates;Mammals"
## [6,] "Eukaryotes;Animals;Vertebrates;Mammals"
还有一些内容可以查看这个包的说明书,在此不一一列举。
3.查找hsa04650所有基因
获得特定KEGG标识符列表后,可以使用 keggGet()它们获取有关它们的更多信息。在这里,我们查找人类基因信号通路hsa04650所有基因。
gs<-keggGet('hsa04650')
names(gs[[1]]) # 说明书里发现的哈
## [1] "ENTRY" "NAME" "DESCRIPTION" "CLASS" "PATHWAY_MAP" "DISEASE" "DRUG" "DBLINKS" "ORGANISM" "GENE" "COMPOUND" "KO_PATHWAY"
## [13] "REFERENCE"
gs[[1]]$GENE # #提取基因
## [1] "3105"
## [2] "HLA-A; major histocompatibility complex, class I, A [KO:K06751]"
## [3] "3106"
## [4] "HLA-B; major histocompatibility complex, class I, B [KO:K06751]"
## [5] "3107"
## [6] "HLA-C; major histocompatibility complex, class I, C [KO:K06751]"
...
4. 提取全部的基因(也是最关键的一步)
class(gs[[1]]$GENE)#先看下数据类型,再做相应的处理
## [1] "character"
数据类型为字符串的类型,考虑使用 strsplit进行拆分,
用法
strsplit(x, split, fixed = FALSE, perl = FALSE, useBytes = FALSE)
strsplit(gs[[1]]$GENE,';')
class(strsplit(gs[[1]]$GENE,';'))
# [1] "list
发现分割后的数据成为了列表,所以我们需要lapply()函数处理列表。使用lapply()函数处理数据后,再使用unlist()函数,将列表转换为非列表形式。
genes <- unlist(lapply(gs[[1]]$GENE,function(x) strsplit(x,';')[[1]][1]))
genes
# [1] "3105" "HLA-A" "3106" "HLA-B" "3107" "HLA-C" "3135" "HLA-G" "3133" "HLA-E" "3812"
# [12] "KIR3DL2" "3811" "KIR3DL1" "3803" "KIR2DL2" "3802"
...
可能上面代码对于初学者理解起来有些困难,但是结合list用法,[[1]]代表获取前面列表中第一个元素,[1]代表获取元素的第一个子数据,再看下面代码就会发现为什么这么写了。
lapply(gs[[1]]$GENE,function(x) strsplit(x,';'))
## [1]]
## [[1]][[1]]
## [1] "3105"
## [[2]]
## [[2]][[1]]
##[1] "HLA-A" " major histocompatibility
## complex, class I, A [KO:K06751]"
##[[3]]
##[[3]][[1]]
## [1] "3106"
...
发现genes数据偶数位为我们所需要的基因名,所以采用整除方法得到。
genes[1:length(genes)%%2 ==0] #length(genes)为基因所在位置
#[1] "HLA-A" "HLA-B" "HLA-C" "HLA-G" "HLA-E" "KIR3DL2" "KIR3DL1" "KIR2DL2" "KIR2DL1" "KIR2DL3" "KIR2DL4"
...
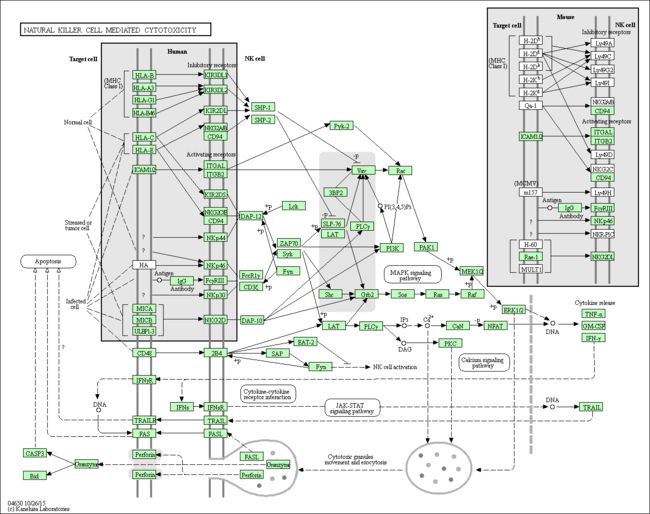
5.查看信号通路图
png <- keggGet("hsa04650", "image")
t <- tempfile()
library(png)
writePNG(png, t)
if (interactive()) browseURL(t)
参考文档:
【1】查看几个KEGG的通路的overlap情况 | 生信菜鸟团 http://www.bio-info-trainee.com/3533.html
【2】KEGG数据库的rest API(附带R语言小技巧) https://cloud.tencent.com/developer/article/1078458
【3】KEGGREST包的说明书(KEGGREST包的说明书https://bioconductor.org/packages/release/bioc/vignettes/KEGGREST/inst/doc/KEGGREST-vignette.html
生信技能树公益视频合辑:学习顺序是linux,r,软件安装,geo,小技巧,ngs组学!
B站链接:https://m.bilibili.com/space/338686099
YouTube链接:https://m.youtube.com/channel/UC67sImqK7V8tSWHMG8azIVA/playlists
生信工程师入门最佳指南:https://mp.weixin.qq.com/s/vaX4ttaLIa19MefD86WfUA
学徒培养:https://mp.weixin.qq.com/s/3jw3_PgZXYd7FomxEMxFmw
生信技能树 - https://www.jianshu.com/u/d645f768d2d5