上期我们给大家介绍过一些基因组测序技术,随着测序技术的发展及其方法不断改进,这的确大大降低基因组测序的成本。有了新颖的测序技术,各式各样的生信分析工具就接踵而至。大家常用哪些生信工具挖掘数据呢?是否觉得用了生信工具做数据,会比做传统实验的效率更高呢?今天小编为你介绍常用的生信分析工具,这对你撰写研究方案和SCI文章写作很有帮助哦!
1. solQTL (了解分析茄科、菊科的表型和基因型)
http://solgenomics.net/qtl/
solQTL是番茄基因组测序计划的基因组信息资源门户网站数据库SGN (http://solgenomics.net) 的子模块。
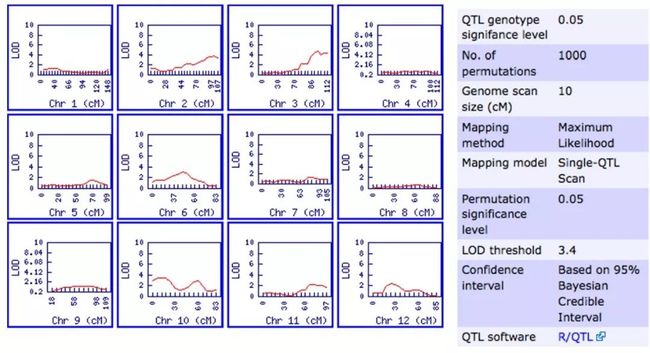
在solQTL上可看到全基因组QTL图谱的很多参数资料:
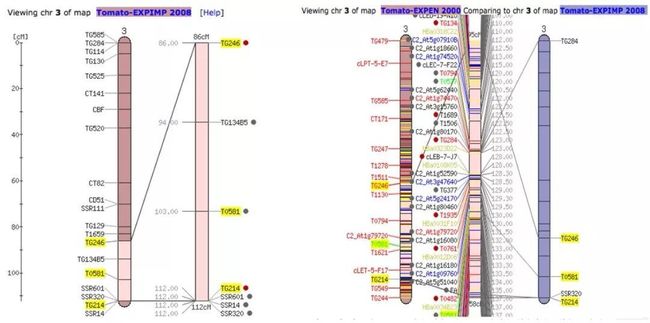
同时,还能清楚看到染色体每个位点:
目前的植物数据库Gramene (http://www.gramene.org)和MaizeGDB (http://www.maizegdb.org),以及动物数据库Animal QTLdb (http://www.animalgenome.org)的唯一最大的缺陷的是不会记录文献上的原始数据,所以用这些数据库是不能很好地分析QTL。
所以,solQTL数据库的网站界面容易操作,允许研究人员将茄科的表型和基因型原始数据上传到SGN数据库,进行QTL(数量性状位点)分析并动态交联到相关的基因组注释。同时,研究人员还能利用solQTL选择QTL图谱的实时统计参数,也可以选择把自己上传的原始和分析的数据公开给其他用户看。
SolQTL数据库的操作说明书可以从该网站上下载哦!
http://solgenomics.net/qtl/guide.pl
2. SeqBuster (分析人类胚胎干细胞小RNA)
http://estivill_lab.crg.es/seqbuster
允许对任何自定义数据库进行注释。在一个独立的平台上SeqBuster综合了多个分析模块,构成了第一个提供miRNA变异体(isomiRs)特性的重要生信工具,SequBuster常用于贮存人胚胎干细胞小RNA的数据集
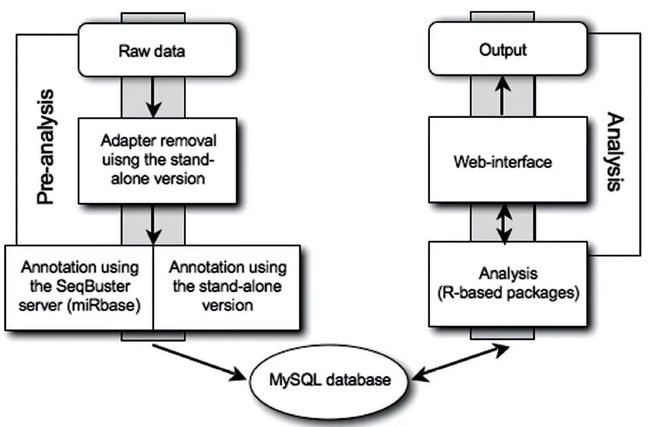
预分析
预分析模块上,原始数据被识别和注释,注释是经由网络服务器生成,也可用自定义的数据库经由独立运行的平台上生成。经处理和注释的数据就会被贮存在MySQL数据库中,这种网络界面可允许用数种R语言包分析数据。输出的数据可以在HTML上看到并保存在服务器、下载到本地硬盘。
3. Phamerator (用于比较噬菌体基因组)
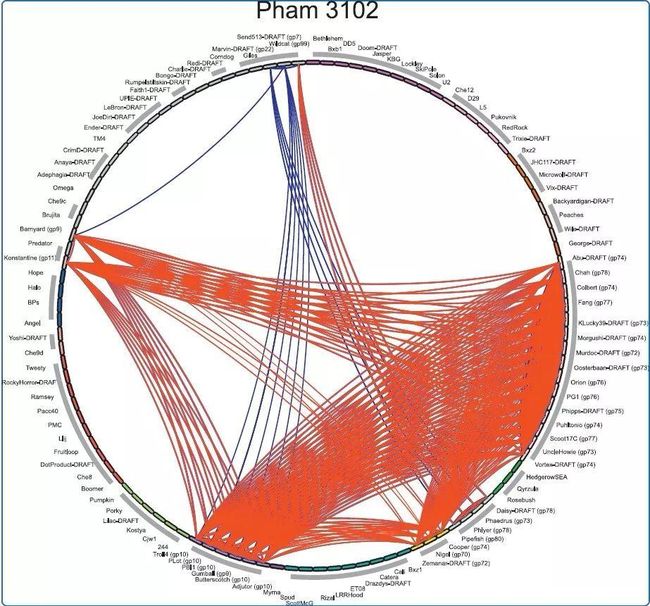
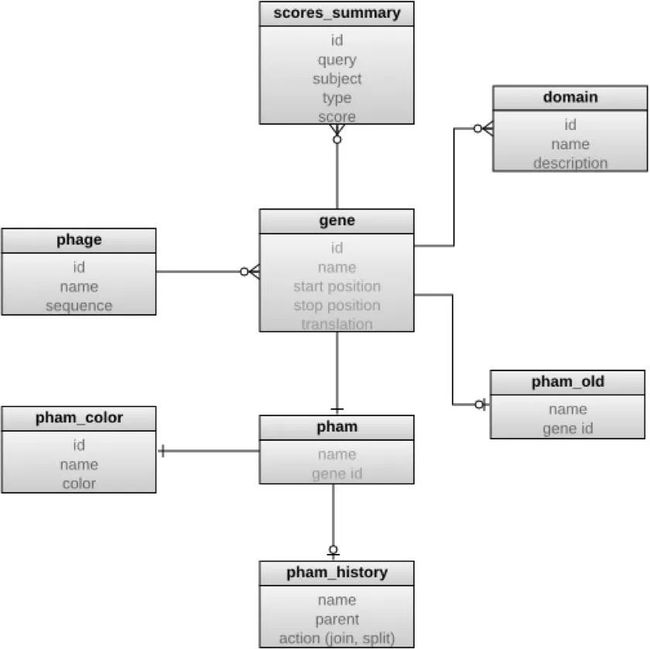
通过成对比较将编码蛋白的基因分配到相关序列的“Phamily基因家族”中,以生成具有共同基因关系的数据库。Phamerator用于生成多种含核苷酸和氨基酸序列的噬菌体以及含有保守结构域的基因的圆形基因组图谱,便于分析通过水平上的基因交换的噬菌体种群的迁移,从而了解到单个基因的进化史。
Phamerator使用MySQL数据库软件,相关基因数据可从GenBank和NCBI保守结构域数据库中找到。
Phamerator数据库的架构:
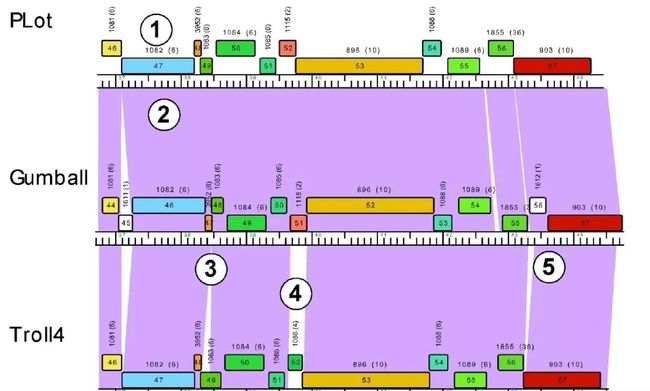
在Phamerator上生成的基因组图谱:
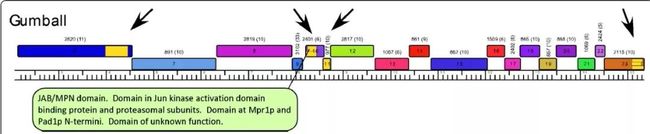
还可以把Gumball基因组其中一段放大,可以了解到基因的功能呢。比如说,在这段6~23区域的基因有四个是保守结构域的基因。
4. ARG-ANNOT [Antibiotic Resistance Gene-ANNOTation](用于检测细菌基因组的抗生素抗性(AR)基因)
在此之前,虽然已有数个AR基因数据库如,2005年的ARGO数据库、2007年的MvirDB的微生物数据库、2009年的ARDB数据库、2012年的Resfinder数据库,但是除了ResFinder之外的这些数据库已再也没更新过。ResFinder只能查到已被人发现的AR基因,而且也不能检测与AR有关的染色体靶基因的点突变。
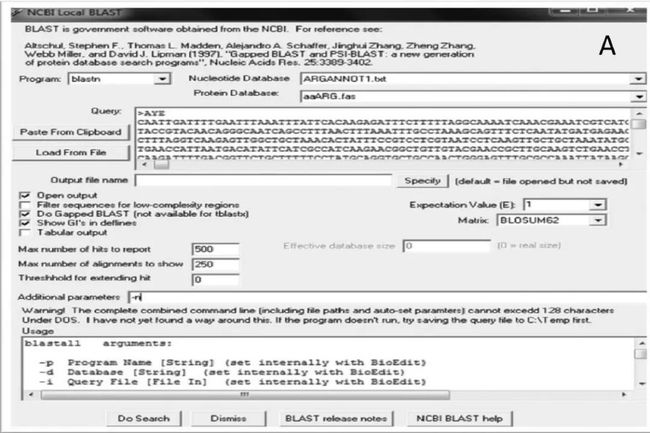
于是,在2014年,Rolain课题组创建了ARG-ANNOT数据库,ARG-ANNOT利用Bio-EditH软件通过BLAST程序来操作,允许用户不需要在网络界面上能分析已知和推定假设的AR基因的序列。具有抗生素抗性的决定簇来自于发表的文章和网络资源,核苷酸和蛋白序列则从NCBI GenBank数据库中提取。通过构建了包含1689个AR基因的数据库后, 随机抽取100条序列测试这个Bio-Edit软件,以证明ARG-ANNOT具有100%特异性和敏感性。
http://www.mbio.ncsu.edu/bioedit/bioedit.html
ARG-ANNOT database browser on Bio-Edit
粘贴要查询的序列、选择数据库类型,再选择不同参数,进行搜索。
参考文献(图片均来自文献 ):
solQTL: a tool for QTL analysis, visualization and linking to genomes at SGN database. BMC Bioinformatics. 2010 Oct 21;11:525.
SeqBuster, a bioinformatic tool for the processing and analysis of small RNAs datasets, reveals ubiquitous miRNA modifications in human embryonic cells. Nucleic Acids Res. 2010 Mar;38(5):e34.
ARGANNOT, a new bioinformatic tool to discover antibiotic resistance genes in bacterial genomes. Antimicrob Agents Chemother. 2014;58(1):212-20.
Phamerator: a bioinformatic tool for comparative bacteriophage genomics. BMC Bioinformatics. 2011 Oct 12;12:395.
• END •