利用LSTM对脑电波信号进行分类
最近我们在做利用LSTM网络对脑电波信号(纺锤体)进行分类的相关工作。我们的数据集是来自于美国开源的睡眠数据集(national sleep research resource)https://sleepdata.org
我们获得数据包含 纺锤波的持续时间,振幅,时间,等几个特征。我们采集的样本主要来自于正常人和病人。我们初步的思想是将纺锤波视为一个事件,生成一个序列。出现纺锤波信号的为1,反之为0.然后利用神经网络,来学习这个序列的内部特征,通过序列来判断病人有没有患病。
众所周知LSTM神经网络在处理时序信息时有一个很好的效果,特别是几年来nlp的飞速发展。
我们首先需要的是对序列进行二进制编码
def bit_coding(data, step): #对一个数据进行编码
code = []
pre_data = 0
count = 0
length = len(data)
while count < length:
n = (data[count]-pre_data) / step
if n > 0:
if n % 1 > 0:
n = int(n)
code += [0] * n + [1]
else:
n = int(n)
code += [0] * (n - 1) + [1]
pre_data = data[count]
count += 1
return code其中的step就是设置的精度,多少步长进行统计,这个值将决定你获得一个脑电波的序列的维度大小。
我们再生成一个脑电波的类的相关信息
class SpindleData:
path = ""
paths = []
labels = []
data = []
step = 0.0001
max_length = 0#设置默认的编码间隔
coding_q = []
def __init__(self, path="datasets", step=0.0001 ):
self.path = path
self.step =step
self.paths, self.labels = self.get_data_labels() #获得路径以及标签
self.coding()
def get_data_labels(self): # 返回获取的数据以及标签[0,1,0,1,...] "./datasets/"
path = self.path
cate = [(os.path.join(path, x)) for x in os.listdir(path)]
paths = []
labels = []
for i, p in enumerate(cate):
path_tmps = glob.glob(os.path.join(p, "*.csv"))
for p in path_tmps:
paths.append(p)
labels.append(i)
np.asarray(labels) #将标签转化为np的格式
return paths, labels
def coding(self):#所有的数据读取以及存储(这里保存了数据的原始数据占用内存可能比较大)
codeing_q = []
for p in self.paths:
data = pd.read_csv(p, skiprows=(0, 1), sep=",")
print("正在读取第%d个csv文件..." % (self.paths.index(p)+1))
data =data['Time_of_night']
self.data.append(data)
for i, d in enumerate(self.data):
code = bit_coding(d, step=self.step)
print("正在对第%d个序列进行编码..."%(i+1))
codeing_q.append(code)#将二位的编码加入到序列中
self.max_length = max([len(x) for x in codeing_q])
codeing_q = preprocessing.sequence.pad_sequences(codeing_q, maxlen=self.max_length) #将所有的串都弄成相同的维度
self.coding_q = np.asarray(codeing_q)这个类主要包含编码后的0/1序列,1序列表示的是纺锤波出现,0表示的是纺锤波没有出现。同时我们利用了keras 的对齐。超过固定长度的部分会被截断,不足的部分会补零。同时我们再生成一个labels数组,这个对应的是病人患病与健康。其实就是一个二分类问题。
我们再来搭建一个LSTM神经网络
def learning_lstm(): #lstm暂时还是比较适合于文本中,对于有序序暂不合适
x_train, y_labels, length = data_test()
x_train = np.expand_dims(x_train, axis=2)
model = Sequential()
# model.add(Embedding(max_feature, 32))
model.add(LSTM(32, input_shape=(length, 1)))
model.add(Dense(1, activation='sigmoid'))
model.compile(optimizer='rmsprop', loss='binary_crossentropy', metrics=['acc'])
model.summary()
history = model.fit(x_train, y_labels, epochs=10, batch_size=16, validation_split=0.2)
draw(history)
def data_test():
length = 0 #每一个系列的长度
spindle = SpindleData()
x_train = spindle.coding_q
y_train = spindle.labels
length = spindle.max_length
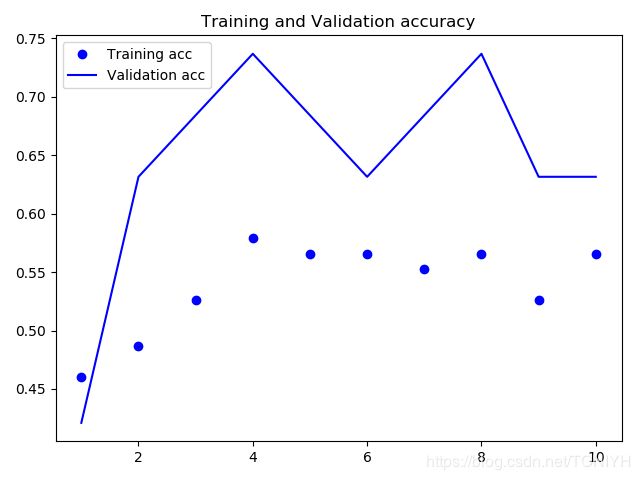
return x_train, y_train, length我实验中的step设置的最小间距0.0001,因此每个序列都有120,000维.暂时实验的效果不是很明显。
由于我们暂时获取的数据还比较小,导致训练不充分,同时可能会导致过拟合等问题。接下来我们会进行改进测试。
我先把github源码发上来,相关的原发发布到了我的github上:https://github.com/danzhewuju/Spindle 欢迎指正!