Luna16数据集
最近在弄跟肺结节相关的东西,所以用了Luna数据集。写一点自己的理解,里面可能会有不准确的地方,希望大家指正。
一.Luna数据集简介(摘自官网)
1.Luna16数据集
- 简介:来自于公开的LIDC/IDRI数据集。该数据集剔除了LIDC/IDRI数据集中切片厚度大于2.5mm的扫描数据,共产生了888套CT。Luna16数据集中结节的判定标准为四名放射科专家中至少有三名认定该结节半径大于3mm。因此在数据集的注释中,非结节、半径小于3mm的结节和被1名或两名放射科专家认为是半径大于3mm的结节被认定为无关的发现。
2.Luna16数据集的组成
- subset0-subset10:10个zip文件中包含所有的CT图像。
- annotation.csv:csv文件中包含用于肺结节检测比赛作为参考标准使用的注释。注释中一共包含1186个结节。
3.mhd和.raw文件
- subset0-10中每一套CT扫描都是由.mhd和.raw共同给出的。.mhd会给出CT图像的一些基本信息。.raw图像用来存储CT的具体数据。
4.annotation.csv文件
- 该文件给出了不同CT中,结节的世界质心坐标和半径。
二.Luna数据集图片解析
1.mhd文件解析
- 使用notepad打开.mhd文件,会看到如下键值对。我们对其中重要的几个键值对进行解释。
ObjectType = Image
NDims = 3
BinaryData = True
BinaryDataByteOrderMSB = False
CompressedData = False
TransformMatrix = 1 0 0 0 1 0 0 0 1
Offset = -161.699997 -169.5 -316.46499599999999
CenterOfRotation = 0 0 0
AnatomicalOrientation = RAI
ElementSpacing = 0.6640620231628418 0.6640620231628418 0.625
DimSize = 512 512 471
ElementType = MET_SHORT
ElementDataFile = 1.3.6.1.4.1.14519.5.2.1.6279.6001.100530488926682752765845212286.raw
- ObjectType是文件类型
- NDims是raw数据的维度
- offset是质心坐标。(第一次看到这个坐标的时候觉得很奇怪,还记得annotation中结节对应的坐标吗。两种坐标都是这种形式的。)其实这种表示也同样应用与dicom文件。结节在矩阵中的位置的计算公式如下
x= (x_ano-x_offset)/x_ElementSpacing
y= (y_ano-y_offset)/y_ElementSpacing
z= (z_ano-z_offset)/z_ElementSpacing
其中,x是实际对应三维矩阵中的坐标。
x_ano是肺结节在annotation.csv中的坐标.
x_offset是质心坐标.
x_ElementSpacing是在x轴方向上的步长。相当于每一个像素对应现实世界中的长度。
- TransformMatric:图像矩阵是否翻转的标志。(在现实中CT扫描的时候有的人是正卧,有的人是仰卧的,所以会导致图像会出现翻转的情况。)
- DimSize:三个数字分别表示x,y,z方向上的维度。
- ElementSpacing:x,y,z方向上的步长
- ElementDataFile:该mhd对应的raw文件,raw中存放了CT值矩阵,也就是所有CT扫描断层。
2.读取CT值矩阵。CT是按层扫描的,每一层CT图像罗列形成三维矩阵。
读取数据的流程是先读取mhd,通过MatricTransform判别三维CT值矩阵在x,y方向上是否需要翻转。然后返回三维CT矩阵。
def load_itk_image(filename):
with open(filename) as f:
contents = f.readlines()
line = [k for k in contents if k.startswith('TransformMatrix')][0]
transform = np.array(line.split(' = ')[1].split(' ')).astype('float')
transform = np.round(transform)
if np.any(transform != np.array([1, 0, 0, 0, 1, 0, 0, 0, 1])):
isflip = True
else:
isflip = False
itkimage = sitk.ReadImage(filename)
numpyimage = sitk.GetArrayFromImage(itkimage)
if(isflip == True):
numpyimage = numpyimage[:,::-1,::-1]
return numpyimage
3.CT值矩阵的预处理与归一化
- CT值与我们平时看到的像素值是不同的。CT值是测定人体某器官密度的计量单位单位是HU。人体组织的范围大约为-1000~+1000.因此如果我们想可视化一层CT,那么我们就要对这一层的二维CT矩阵进行预处理与归一化。
具体成像的原理可以找详细资料,比如医学中的肺窗和纵膈窗。不同窗位和算法形成的图像也是不同的。这里我们只是从可视化的角度找了一种最简单的处理方法。
- 预处理:首先就是将CT值过大和过小的数据置为0。
def truncate_hu(image_array):
image_array[image_array > 400] = 0
image_array[image_array <-1000] = 0
- 归一化
def normalazation(image_array):
max = image_array.max()
min = image_array.min()
image_array = (image_array-min)/(max-min)
avg = image_array.mean()
image_array = image_array-avg
return image_array
- 这里得到的image_array就是归一化后的图像矩阵了。我们可以用matplotlib可视化一层。
4.可视化一层
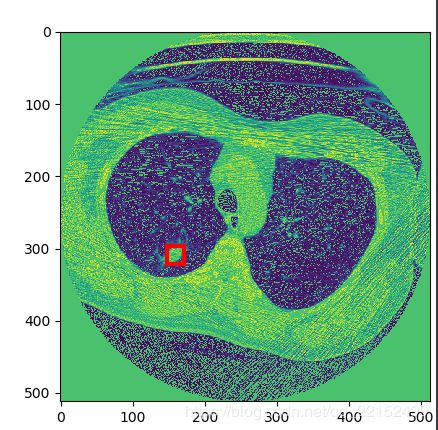
- 下面的程序挑取一层进行可视化,并标注结节位置。
a = image_array.transpose(1,2,0)[:,:,0] #transpose是将(z,x,y)的三维矩阵转为(x,y,z)的矩阵
plt.gca().add_patch( plt.Rectangle((147,297), 24,24, fill=False,edgecolor='r', linewidth=3))
plt.imshow(a[:,:,1]*255)#在图中画框
plt.show()