sklearn机器学习:PCA在人脸识别,降噪,手写数字案例中的使用
使用sklearn进行PCA降维之前还是需要知道PCA与SVD的知识的,移步主成分分析与奇异值分解。接下来称为一个调包侠加调参侠,对于原理基本不会说什么。依次学习以下内容。
- PCA中的重要参数与使用PCA进行降维
- PCA中的SVD以及一些接口的使用
- 人脸识别案例
- 降噪案例
- PCA对手写数据集的降维
一、PCA中的重要参数与使用PCA进行降维
在数据预处理与特征工程部分,提到过一种重要的特征选择方法:方差过滤。如果一个特征的方差很小,则意味着这个特征上很可能有大量取值都相同(比如90%都是1,只有10%是0,甚至100%是1),那这一个特征的取值对样本而言就没有区分度,这种特征就不带有有效信息。从方差的这种应用就可以推断出,如果一个特征的方差很大,则说明这个特征上带有大量的信息。因此,在降维中,PCA使用的信息量衡量指标,就是样本方差,又称可解释性方差,方差越大,特征所带的信息量越多的表现可能会因此受影响。
下面使用鸢(yuan 第一声)尾花数据集来使用sklearn中的PCA
#高维数据的可视化
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn.decomposition import PCA
import pandas as pd
iris = load_iris()
#iris.data.shape
x = iris.data #(150,4) 特征矩阵
y = iris.target #标签,3种鸢尾花类别
pca = PCA(n_components=2) #保留两个特征
x_dec = pca.fit_transform(x) #拟合并导出新的特征矩阵
x_dec.shape #输出(150, 2)
实例化对象那步pca = PCA(n_compinents=2) 其中参数是指定降到多少维,这里是二维,鸢尾花数据集是4维的,继续上面的,降到2维那么就可以进行可视化了
plt.scatter(x_dec[y==0,0],x_dec[y==0,1],label=iris.target_names[0])
plt.scatter(x_dec[y==1,0],x_dec[y==1,1],label=iris.target_names[1])
plt.scatter(x_dec[y==2,0],x_dec[y==2,1],label=iris.target_names[2])
plt.title("PAC OF IRIS")
plt.legend()
plt.show()
可视化的样子如下

显然,簇的分布是很明显的,这时候不管是使用KNN或者随机森林等都不容易分错,准确率基本都能达到95%以上,所以说即使降到2维了也不会影响模型效果。
来看一下PCA的属性.explained_variance_,.explained_variance_ratio_

对于参数n_components有多种类型取值,可以是整数,表示降维后特征的数量;可以是[0,1]的小数,表示希望保留的信息量百分比;可以是字符类型"mle",此时会自选时候的整数值;可以直接使用PCA(k)的形式也行;甚至可以不填,不填的话默认是min(x.shape),由于一般是特征数量小于样本数量,所以结果通常是特征数量。
#n_components不填的话默认是min(x.shape),由于一般是特征数量小于样本数量,所以结果通常是特征数量
pca = PCA()
x_def = pca.fit_transform(x)
pca.explained_variance_ratio_
输出:array([0.92461872, 0.05306648, 0.01710261, 0.00521218])
补充:容易看出来第一个特征(特征向量)几乎包含了所有的原始信息,如果维度巨多比如成百上千,也可以发现是前几个特征几乎包含了所有的元素信息,所以通常取前K个特征就行。我觉得自己表达的不清楚,具体的,PCA推导中最后得到的对角化的矩阵对角线元素均为各个特征值,特征值从上到下是递减排列的,而且降的特别快,如果觉得稀里糊涂,一定要看主成分分析与奇异值分解
#极大似然估计自选超参数 选择的新特征数量是3
pca = PCA(n_components="mle")
x_mle = pca.fit_transform(x)
x_mle.shape,pca.explained_variance_ratio_
输出:((150, 3), array([0.92461872, 0.05306648, 0.01710261]))
#按照信息量占比选择超参数
pca = PCA(n_components=0.95)
x_ratio = pca.fit_transform(x)
x_ratio.shape,pca.explained_variance_ratio_
输出:((150, 2), array([0.92461872, 0.05306648]))
二、PCA中的SVD
使用PCA进行降维的时候是需要计算协方差矩阵 X X T XX^T XXT的,其中 X = ( x i j ) m × n X=(x_{ij})_{m×n} X=(xij)m×n表示特征矩阵,这个矩阵的计算量是非常大的,sklearn中会使用SVD了来帮助计算,在主成分分析与奇异值分解中说过了其实可以根据PCA的中的一些计算得到SVD中的三个矩阵的。这里就不继续说了,实际上sklearn中是使用了算法得到了 k × n k×n k×n维度的矩阵 V k × n V_{k×n} Vk×n的,然后 X ∗ V T X*V^T X∗VT得到 m × k m×k m×k的新特征矩阵,特征数即 k k k。关于这个 V V V是可以直接通过属性获得的,大致流程如下。
#PCA中的SVD在transform过程之后,fit中奇异值分解
#的结果除了V(k,n)以外,就会被舍弃,而V(k,n)会被保存在属性components_ 当中,可以调用查看。
pca = PCA(2).fit(x)
pca.components_ ,pca.components_.shape #k×n -> 2×4 x*V^T -> 4×2 2表示新特征数量
输出
(array([[ 0.36138659, -0.08452251, 0.85667061, 0.3582892 ],
[ 0.65658877, 0.73016143, -0.17337266, -0.07548102]]), (2, 4))
三、案例:人脸识别来看降维后的V的信息保存
#人脸识别案例
from sklearn.datasets import fetch_lfw_people
from sklearn.decomposition import PCA
import numpy as np
import matplotlib.pyplot as plt
faces = fetch_lfw_people()
faces.data.shape,faces.images.shape #62*47=2924,像素 前者是2维数组后者是3维数组
输出:((13233, 2914), (13233, 62, 47))
figs,objs = plt.subplots(3,5
,figsize=(8,5)
,subplot_kw={"xticks":[],"yticks":[]} #不显示坐标轴
)
for i,obj in enumerate(objs.flat):#或者直接使用faces.images[i]
obj.imshow(faces.data[i].reshape(62,47),cmap="gray")
来看原始数据的输出
现在我们进行降维,使用fit后得到的矩阵 V V V来画图
#提取新特征空间 降维
x = faces.data
pca = PCA(200).fit(x)
V = pca.components_
#V.shape #输出(200, 2914) k=200,n=2914
figs,objs = plt.subplots(2,5,figsize=(8,5),subplot_kw={"xticks":[],"yticks":[]})
for i,obj in enumerate(objs.flat):
obj.imshow(V[i,:].reshape(62,47),cmap="gray")
再来看画出的图像
比起降维前的数据,新特征空间可视化后的人脸非常模糊,这是因为原始数据还没有被映射到特征空间中。但是可以看出,整体比较亮的图片,获取的信息较多,整体比较暗的图片,却只能看见黑漆漆的一块。在比较亮的图片中,眼睛,鼻子,嘴巴,都相对清晰,脸的轮廓,头发之类的比较模糊。
这说明,新特征空间里的特征向量们,大部分是"五官"和"亮度"相关的向量,所以新特征向量上的信息肯定大部分是由原数据中和"五官"和"亮度"相关的特征中提取出来的。到这里,我们通过可视化新特征空间V,解释了一部分降维后的特征:虽然显示出来的数字看着不知所云,但画出来的图表示,这些特征是和”五官“以及”亮度“有关的。这也再次证明了,PCA能够将原始数据集中重要的数据进行聚集
我们学到了神奇的接口inverse_transform,可以将我们归一化,标准化,甚至做过哑变量的特征矩阵还原回原始数据中的特征矩阵,这几乎在向我们暗示,任何有inverse_transform这个接口的过程都是可逆的。PCA应该也是如此。在sklearn中,我们通过让原特征矩阵X右乘新特征空间矩阵V(k,n)来生成新特征矩阵X_dr,那理论上来说,让新特征矩阵X_dr右乘V(k,n)的逆矩阵 ,就可以将新特征矩阵X_dr还原为X。那sklearn是否这样做了呢?下面通过人脸识别来看看
#人脸识别案例
from sklearn.decomposition import PCA
from sklearn.datasets import fetch_lfw_people
import numpy as np
import matplotlib.pyplot as plt
faces = fetch_lfw_people()
x = faces.data
pca = PCA(200)
x_face = pca.fit_transform(x)
print(x_face.shape)
x_inverse = pca.inverse_transform(x_face)
print(x_inverse.shape)
输出
(13233, 200)
(13233, 2914)
从输出来看,确实是还原成了2914维,那么就画出来看
figs, objs = plt.subplots(2,10,figsize=(10,2.5),subplot_kw={"xticks":[],"yticks":[]})
for i in range(10):
objs[0,i].imshow(x[i].reshape(62,47),cmap="gray")
objs[1,i].imshow(x_inverse[i].reshape(62,47),cmap="gray")

对比一下,一个是原始数据得到的人脸,一个是降维后使用接口inverse_transform还原回去的数据,得到的人脸是非常相似的,虽然后者有点模糊。
这说明,inverse_transform并没有实现数据的完全逆转。这是因为,在降维的时候,部分信息已经被舍弃了,X_face中往往不会包含原数据100%的信息,所以在逆转的时候,即便维度升高,原数据中已经被舍弃的信息也不可能再回来了。所以,降维不是完全可逆的。但是同时也说明了,使用200维度,的确保留了原数据的大部分信息,所以图像看起来,才会和原数据高度相似,只是稍稍模糊罢了。
四、使用PCA进行降噪
降维的目的之一就是希望抛弃掉对模型带来负面影响的特征,而我们相信,带有效信息的特征的方差应该是远大于噪音的,所以相比噪音,有效的特征所带的信息应该不会在PCA过程中被大量抛弃。inverse_transform能够在不恢复原始数据的情况下,将降维后的数据返回到原本的高维空间,即是说能够实现”保证维度,但去掉方差很小特征所带的信息“。利用inverse_transform的这个性质,我们能够实现噪音过滤。
#案例 用PCA做噪音过滤
from sklearn.datasets import load_digits
dig = load_digits()
dig.data.shape,dig.images.shape
输出 : ((1797, 64), (1797, 8, 8))
#定义画图方法
def draw_img(data):
figs, objs = plt.subplots(2,10,figsize=(10,2.5),subplot_kw={"xticks":[],"yticks":[]})
for i,obj in enumerate(objs.flat):
obj.imshow(data[i].reshape(8,8),cmap="binary")
现在来为数据加上噪音
#为数据加上噪音 在指定的数据集中,随机抽取服从正态分布的数据 两个参数,分别是指定的数据集,
#和抽取出来的正太分布的方差
noisy = np.random.normal(dig.data,2)
draw_img(noisy)
现在来对具有噪音的数据进行降维,然后还原。
#降维 之后还原 清楚
pca = PCA(0.8)
x_dr = pca.fit_transform(noisy)
x_inverse = pca.inverse_transform(x_dr)
draw_img(x_inverse)
五、PCA对手写数据集的降维
#PCA对手写数字数据集的降维
import pandas as pd
data = pd.read_csv("D:/digit recognizor.csv")
data.shape
输出:(42000, 785)
x = data.iloc[:,1:]
y = data.iloc[:,0]
pca = PCA().fit(x)
plt.plot(np.cumsum(pca.explained_variance_ratio_))
plt.xlabel("dimention")
plt.ylabel("explained variance ratio")
plt.show()

从图中可以发现,当维度在100的时候可解释方差贡献率就很大了,那么我们从1-100中选一个最合适的。使用学习曲线来求超参数k,还是先确定大致范围最后再确定具体指
# 确定大致范围
from sklearn.model_selection import cross_val_score
from sklearn.ensemble import RandomForestClassifier as RFC
x = data.iloc[:,1:]
y = data.iloc[:,0]
scores = []
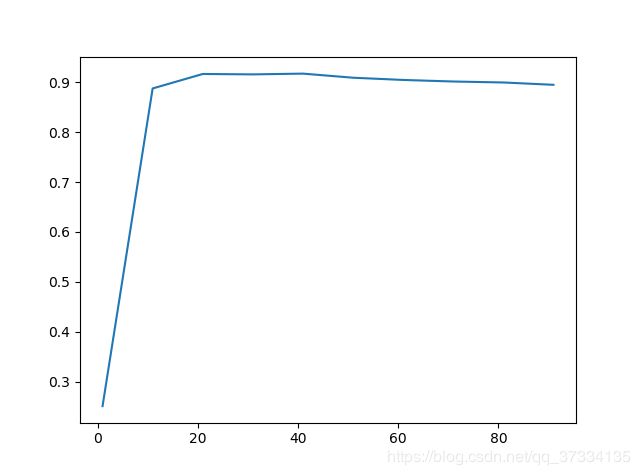
for i in range(1,101,10):
x_tr = PCA(i).fit_transform(x)
score = cross_val_score(RFC(n_estimators=10,random_state=20),x_tr,y,cv=5).mean()
scores.append(score)
plt.plot(range(1,101,10),scores)
plt.show()
#确定范围
from sklearn.model_selection import cross_val_score
from sklearn.ensemble import RandomForestClassifier as RFC
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import pandas as pd
data = pd.read_csv("D:/digit recognizor.csv")
x = data.iloc[:,1:]
y = data.iloc[:,0]
scores = []
for i in range(15,30):
x_tr = PCA(i).fit_transform(x)
score = cross_val_score(RFC(n_estimators=10,random_state=20),x_tr,y,cv=5).mean()
scores.append(score)
plt.plot(range(15,30),scores)
plt.show()
从图中可以看到k=25的时候,模型效果最好
x_tr = PCA(25).fit_transform(x)
cross_val_score(RFC(n_estimators=10,random_state=20),x_tr,y,cv=5).mean()
输出:0.9195949043467323
跟之前使用的特征工程的时候的效果0.96xxx相比,效果不是很好,调下参数n_estimators
x_tr = PCA(25).fit_transform(x)
cross_val_score(RFC(n_estimators=100,random_state=20),x_tr,y,cv=5).mean()
输出:0.9460957505533008
效果变好了,但是还是比0.96差不少,也许调参能使得模型效果上去,但是考虑到这里特征只有25个,那么使用KNN就不会那么慢了,使用换个KNN的模型来跑
from sklearn.neighbors import KNeighborsClassifier as KNN
x_tr = PCA(25).fit_transform(x)
cross_val_score(KNN(),x_tr,y,cv=5).mean()
输出:0.9703568517177686
精确度一下就上去了,那么就使用KNN而不使用随机森林了。