- 1 数据准备
- 2 Logit回归
- 3 决策树
- 3.1 经典决策树
- 3.2 条件推断树
- 3.3 随机森林
- 3.4 支持向量机(SVM)
knitr::opts_chunk$set(echo = TRUE)
| 包 |
函数 |
分类方法 |
| 基本包 |
glm() |
逻辑回归 |
| rpart |
rpart() |
经典决策树 |
| party |
ctree() |
条件推断树 |
| randomForest |
randomForest() |
经典决策树的随机森林 |
| party |
cforest() |
条件推断树的随机森林 |
| e1071 |
svm() |
支持向量机 |
数据准备
#读取数据
loc <- "http://archive.ics.uci.edu/ml/machine-learning-databases/"
ds <- "breast-cancer-wisconsin/breast-cancer-wisconsin.data"
url <- paste(loc, ds, sep="")
breast <- read.table(url, sep=",", header=FALSE, na.strings = "?")
names(breast) <- c("ID", "clumpThickness", "sizeUniformity", "shapeUniformity", "maginalAdhesion","singleEpithelialCellSize", "bareNuclei", "blandChromatin", "normalNucleoli", "mitosis", "class")
df <- breast[-1] # 删除ID
#将class转为因子
df$class <- factor(df$class, levels=c(2,4), labels=c("benign", "malignant"))
#将库分为训练集和验证集
set.seed(1234)
train <- sample(nrow(df), 0.7*nrow(df))
df.train <- df[train,] #70%样本进入训练集
df.validate <- df[-train,] #30%样本进入验证集
#查看训练集和验证集的因变量分布
table(df.train$class)
table(df.validate$class)
Logit回归
#逻辑回归
fit.logit <- glm(class~., data=df.train, family=binomial())
summary(fit.logit)
#基于训练集的回归模型,对验证集的样本进行预测。
#predict()默认输出对数概率,指定参数type="response"即可得到概率。
prob <- predict(fit.logit, df.validate, type = "response")
logit.pred <- factor(prob>.5 , levels=c(FALSE, TRUE), labels=c("benign","malignant"))
#评估预测的准确性:预测结果与实际情况交叉对比。
logit.perf <- table(df.validate$class, logit.pred, dnn=c("Actual","Predicted"))
logit.perf
决策树
经典决策树
- rpart包中的rpart()函数构造决策树;
- prune()函数对决策树进行剪枝。
library(rpart)
set.seed(1234)
#生成树
dtree <- rpart(class~., data=df.train, method="class", parms = list(split="information"))
#rpart()返回的cptable值包括不同大小的树对应的预测误差。
#CP复杂度参数,nsplit树的分支数; rel errror误差; xerror10折交叉验证误差;xstd交叉验证误差的标准差。
dtree$cptable
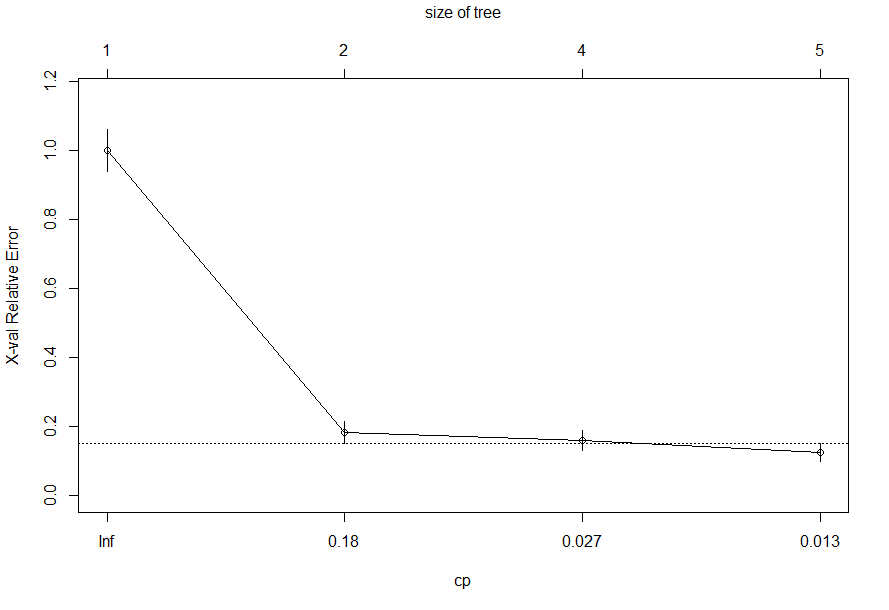
#画图。虚线代表基于一个标准差准则得到的上限。选择虚线下最左侧CP值对应的树。
plotcp(dtree)
#剪枝
dtree.pruned <- prune(dtree, cp=0.0176)
# package ‘rpart.plot’ is not available (for R version 3.6.0) 没法安装画图包。
#library(rpart.plot)
#prp(dtree.pruned, type=2, extra=104, fallen.leaves=TRUE, main="Decision Tree")
# 基于训练集的回归模型,对验证集的样本进行预测,并验证。
dtree.pred <- predict(dtree.pruned, df.validate, type="class")
dtree.perf <- table(df.validate$class, dtree.pred, dnn = c("Actual", "Predicted"))
dtree.perf
条件推断树
- 变量和分割的选择基于显著性检验,而不是纯净度或同质性一类的度量。
- 条件推断树由party包中的ctree()函数获得。
- 条件推断树不需要剪枝。
library(party)
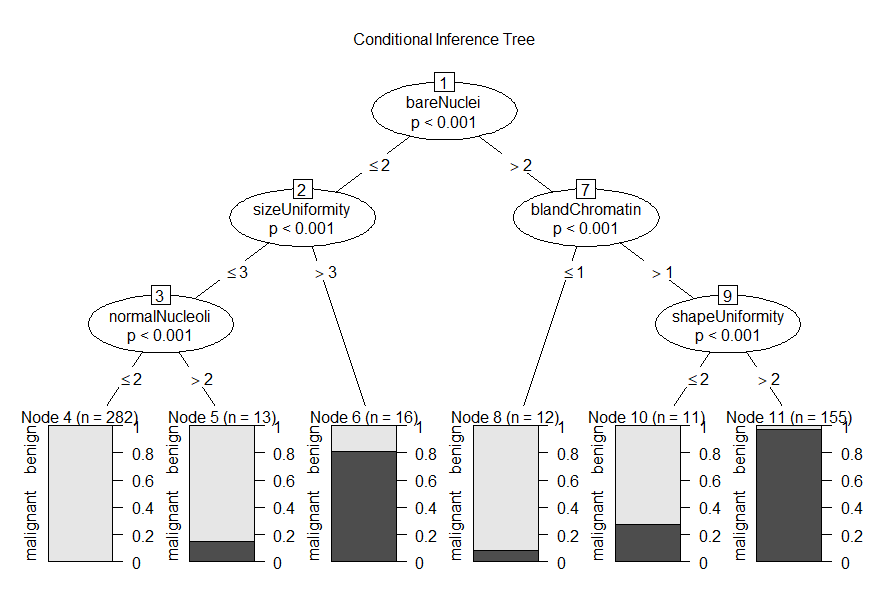
fit.ctree <- ctree(class~., data=df.train)
plot(fit.ctree, main="Conditional Inference Tree")
ctree.pred <- predict(fit.ctree, df.validate, type="response")
ctree.perd <- table(df.validate$class, ctree.pred, dnn = c("Actual", "Predicted"))
ctree.perd
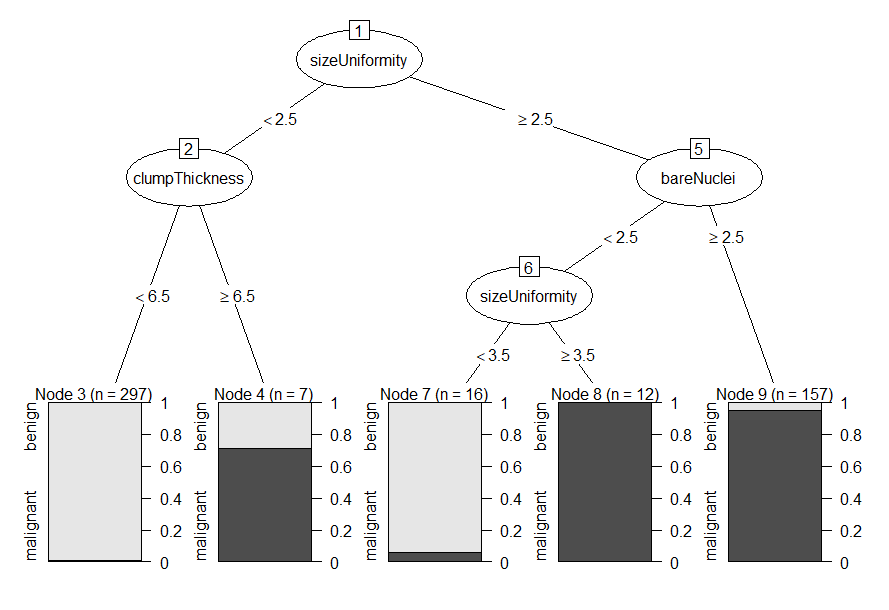
- partykit包,通过plot(as.party(an.rpart.tree))可以画经典决策树。
library(partykit)
plot(as.party(dtree.pruned))
随机森林
- 随机森林中,同时生成多个预测模型,并将模型的结果汇总以提升分类准确率。
- randomForest包中的randomForest()函数可用于生成(传统决策树)随机森林。
- party包中的cforest()函数可用于生成(条件推断树)随机森林。
- 当预测变量间高度相关时,基于条件推断树的随机森林可能效果更好。
- 随机森林可处理大规模问题(多样本多变量)和大量缺失值的数据。
- 随机森林可计袋外预测误差(OOB error)和度量变量重要性。
library(randomForest)
set.seed(1234)
fit.forest <- randomForest(class~., data=df.train, na.action=na.roughfix, importance=TRUE)
fit.forest
# 度量变量重要性
importance(fit.forest, type=2)
# 预测和评估
forest.pred <- predict(fit.forest, df.validate)
forest.perf <- table(df.validate$class, forest.pred, dnn = c("Actual","Predicted"))
forest.perf
支持向量机(SVM)
- SVM旨在在多维空间中找到一个能将全部样本单元分成两类的最优平面。
- SVM通过kernlab包的ksvm()函数和e1071包中的svm()函数实现。前者功能更强大,后者更易用。
- SVM在预测新样本单元时,不允许有缺失值出现。
library(e1071)
set.seed(1234)
fit.svm<- svm( class~., data=df.train)
fit.svm
svm.pred <- predict(fit.svm, na.omit(df.validate))
svm.perf <- table(na.omit(df.validate)$class, svm.pred, dnn=c("Actual", "Predicted"))
svm.perf
- SVM拟合样本时,两个参数影响最终结果。样本范围参数gamma,成本参数cost。
- gamma默认为预测变量个数的倒数; cost默认为1。
- tune.svm()函数可以设置gamma和cost参数的范围,根据各组合的表现,输出一个最佳组合。
- tune.svm()比较耗时间。
set.seed(1234)
tuned <- tune.svm(class~., data = df.train, gamma = 10^(-6:1), cost=10^(-10:10))
tuned
fit.svm<- svm( class~., data=df.train, gamma=0.01, cost=1)
svm.pred <- predict(fit.svm, na.omit(df.validate))
svm.perf <- table(na.omit(df.validate)$class, svm.pred, dnn=c("Actual", "Predicted"))
svm.perf