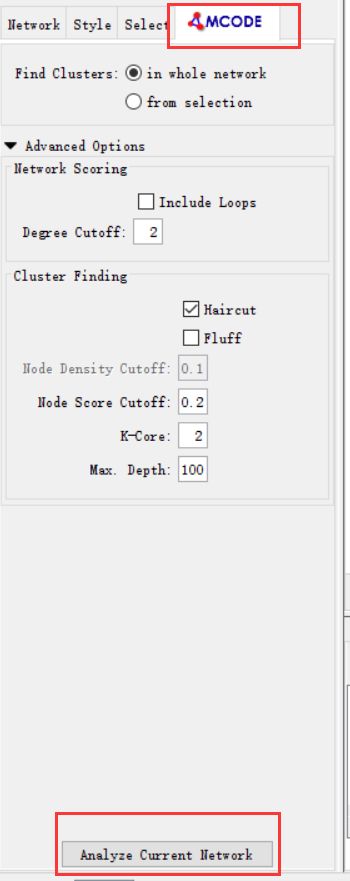
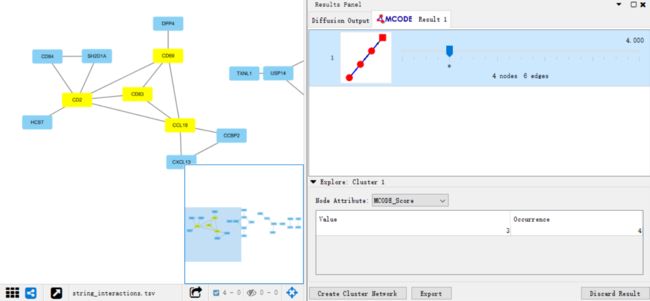
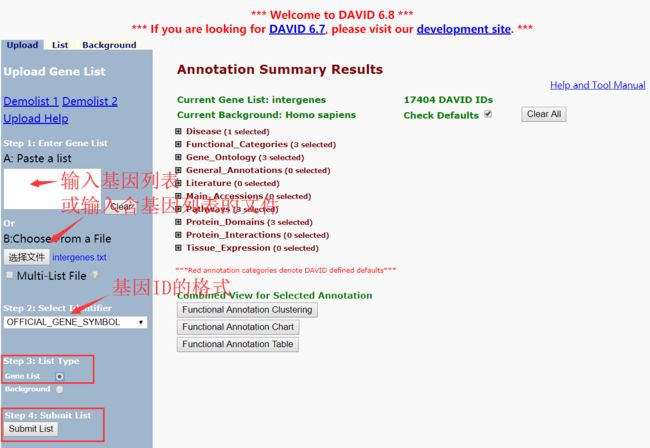
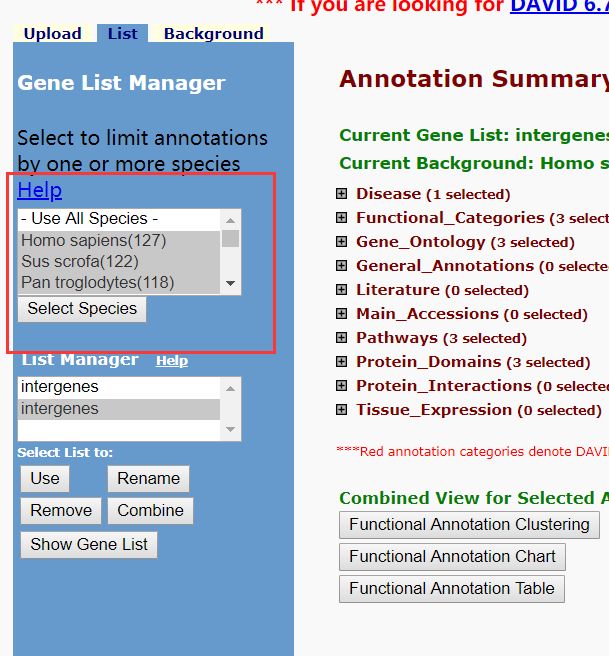
- 小鹏P7自动泊车技术方案浅析
yuyuelongfly
自动驾驶小鹏P7APA自动泊车自动驾驶
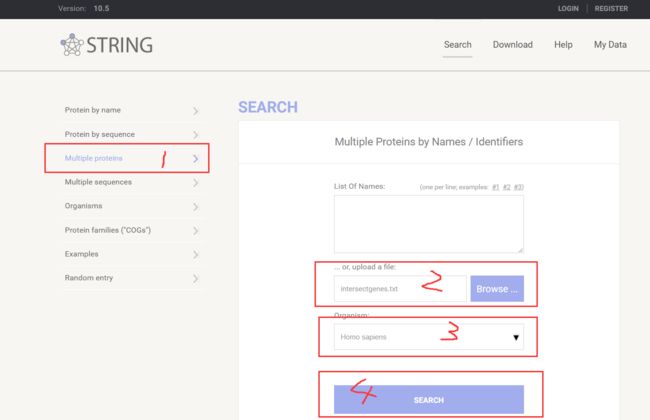
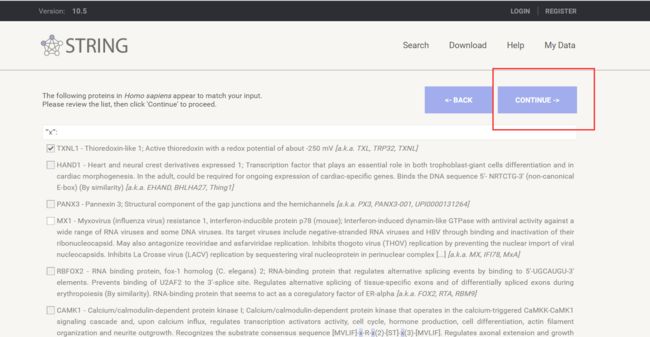
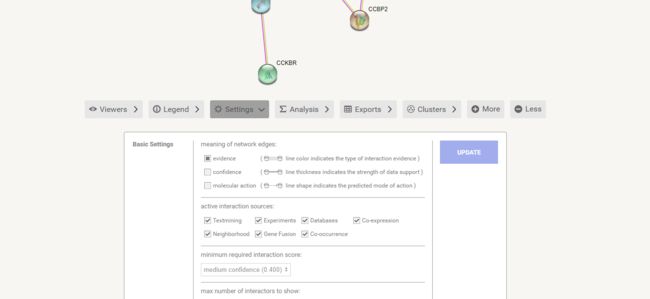
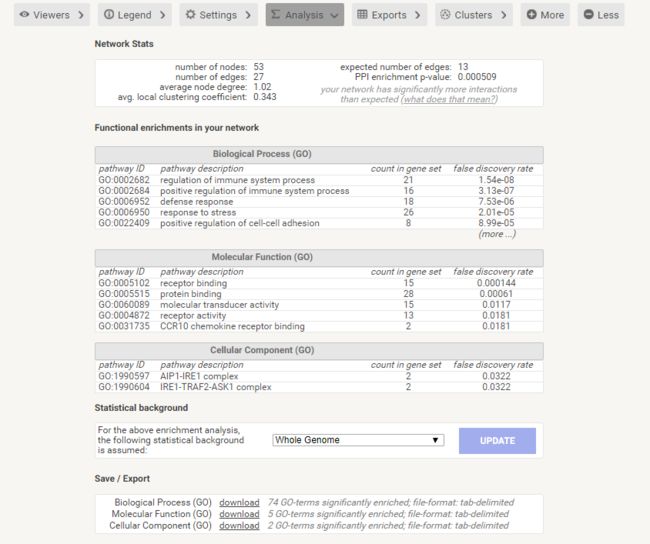
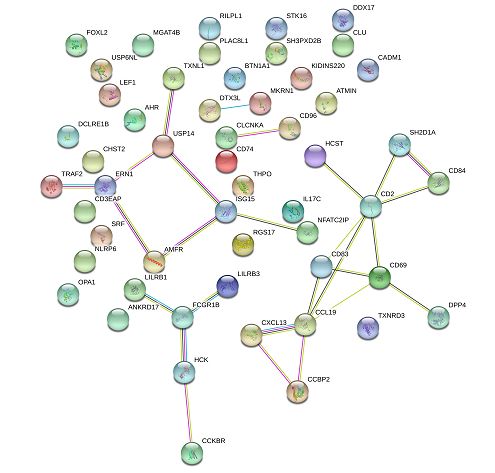
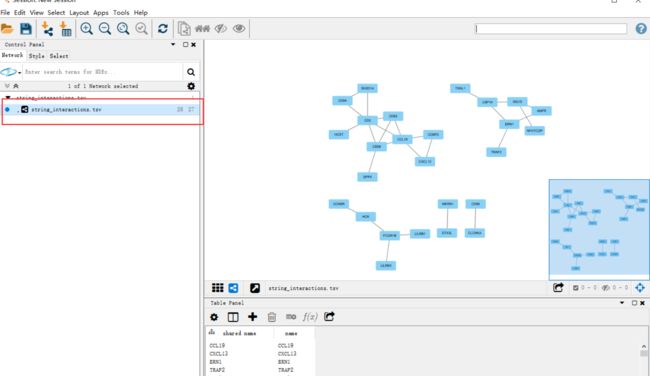
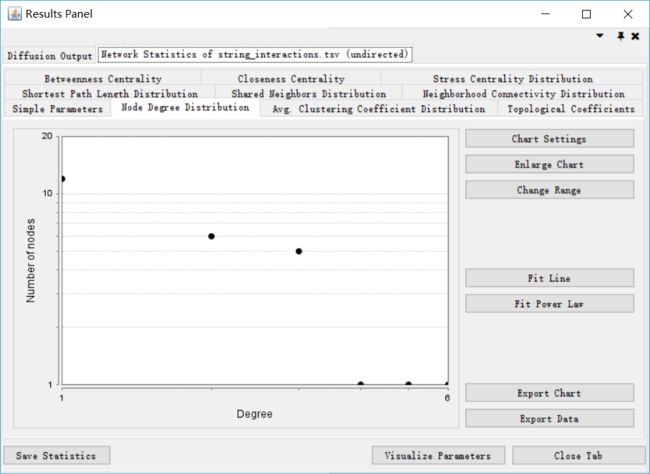
目录一、概述二、感知算法1.视觉库位检测1.1.视觉系统1.2.库位检测算法1.3.同步建图与定位技术1.4.其他要素检测2.超声波库位检测3.视觉库位检测与超声波库位检测融合三、路径规划与控制四、HMI一、概述泊车算法离不开感知&融合、规划&控制,从目前行业技术发展的角度来看,泊车涉及的每一个算法都不算完美,甚至可以说仍不成熟。然而,小鹏P7采用优秀的系统方案设计,特别是通过引入同步建图与定位技
- Java-servlet(四)详细讲解Servlet类层次结构与生命周期
珹洺
Javaservletjavaservlet开发语言运维服务器后端
Java-servlet(四)详细讲解Servlet和生命周期前言一、Servlet类层次结构1.Servlet包2.Servlet与ServletConfig与Serializable的关系1.三者分别是什么2.三者之间的关系3.如何让类具有Servlet功能二、Servlet生命周期1.初始化阶段2.服务阶段3.销毁阶段总结前言Servlet是JavaWeb开发的核心组件,负责处理客户端请求并
- 【北上广深杭大厂AI算法面试题】深度学习篇...MobileNet 系列网络发展与高效性分析(附代码)(二)
努力毕业的小土博^_^
AI算法题库人工智能算法深度学习卷积神经网络
【北上广深杭大厂AI算法面试题】深度学习篇…MobileNet系列网络发展与高效性分析(附代码)(二)【北上广深杭大厂AI算法面试题】深度学习篇…MobileNet系列网络发展与高效性分析(附代码)(二)文章目录【北上广深杭大厂AI算法面试题】深度学习篇...MobileNet系列网络发展与高效性分析(附代码)(二)MobileNet系列网络发展与高效性分析(附代码)4.MobileNetV2(2
- ”天下第一神数“——紫微斗数的JAVA实现!紫微玄机速run~
钮钴禄·爱因斯晨
赛博算命JAVA实现javapython开发语言
各位佬儿们好呀~~互三必回哦~更多精彩:个人主页赛博算命精彩文章:梅花易数的java实现赛博算命系列文章不作溢美之词,不作浮夸文章,此文与功名进取毫不相关也!与各位共勉!!文章目录#前言:一、紫微斗数简介二、紫微斗数的数学原理1.**命盘构建规则**2.**星曜分布算法**3.**运势推导逻辑**三、Java实现步骤1.代码分布实现1.1**数据结构设计**1.2**命盘构建算法实现**1.3**
- 华为OD-不限经验,急招,机考资料,面试攻略,不过改推,捞人
2301_79125642
java
超星(学习通)-Java后端一面网易互娱40min(感觉是G了)一篇不太像面经的面经2023总结,前端大二上进小红书秋招面经第一波海康红外图像算法实习(微影)面经测试工程师社招-测试面试题大厂在职傻屌。TPlink图像算法工程师一二三面经深圳海康红外图像算法实习(微影)面经TPLink提前批面经(已OC)传统车辆转规控算法岗秋招记录腾讯TEG测试与质量管理全记录瑞幸Java开发校招一面腾讯金融科技
- 小鹏汽车申请注册“P7 Ultra”商标 或为P7车型升级版铺路
大湾区经济门户网
汽车媒体人工智能大数据
大湾区经济网品牌工程频道报道,据企查查APP显示,广东小鹏汽车科技有限公司近日提交“P7Ultra”商标注册申请,国际分类为运输工具,当前状态为“注册申请中”。业内推测,此举或为小鹏P7车型高端版本量产上市做准备。作为小鹏汽车主力车型,P7系列自2020年上市以来累计交付量已突破14万辆,2024年前4个月仍保持月均超2000辆销量。此次申请“Ultra”后缀商标,或延续其产品线升级策略——此前小
- 大模型最新面试题系列:训练篇之模型监控与调试
人肉推土机
大模型最新面试题集锦大全面试人工智能pytorchAI编程语言模型
1.训练过程中需要监控哪些关键指标?如何设置报警阈值?关键指标损失函数值:包括训练损失和验证损失,反映模型在训练和验证数据上的拟合程度。准确率:分类任务中的预测正确样本占总样本的比例,评估模型的预测能力。召回率和F1值:在二分类或多分类任务中,用于更全面地评估模型性能,特别是在正负样本不均衡的情况下。学习率:监控学习率的变化,确保其处于合适的范围,避免学习率过大导致模型不稳定或过小导致训练收敛过慢
- 【华为OD机试真题E卷】54、统一限载货物数最小值 | 机试真题+思路参考+代码解析(C++、Java、Py)
KFickle
JavaPy)华为odc++java华为OD机试真题统一限载货物数最小值
文章目录一、题目题目描述输入输出样例1样例2二、代码与思路参考C++代码Java代码Python代码订阅本专栏后即可解锁在线OJ刷题权限个人博客首页:KFickle专栏介绍:最新的华为OD机试真题D、E卷,每题都使用C++,Java,Python语言进行解答,每个题目的思路分析都非常详细,持续更新,支持在线OJ刷题,订阅后评论获取权限,有代码问题随时解答,代码仅供学习参考一、题目题目描述火车站附近
- 【MATLAB源码-第128期】基于matlab的雷达系统回波信号仿真,输出脉压,MTI,MTD等图像。
Matlab_猿助手
调制解调通信原理MATLABmatlab开发语言信息与通信
操作环境:MATLAB2022a1、算法描述雷达(RadioDetectionandRanging)是一种使用无线电波来探测和定位物体的系统。它的基本原理是发射无线电波,然后接收这些波从目标物体上反射回来的信号。通过分析这些反射波,雷达能够确定物体的位置、速度、方向和其他特性。历史背景雷达技术起源于20世纪初。最初的发展动机主要是军事上的需求,特别是在第二次世界大战期间,雷达在侦测敌机和舰船上发挥
- 地球科学数据学习笔记---流向与风向、浪向
fried-ghost
地球科学数据学习笔记学习笔记数据分析
一、流向(current)流向一般指流体前进的方向、去向,一般以正北方向为正,例如流体从南流向北,则流向为0°,其示意图如下二、风向与浪向风向与浪向一般都指来向,与流向相反,例如风从南吹向北,则为南风,风向为180°。气象数据中一般会将风速数据存成u、v两个分量(雷达数据除外),u分量表示纬向风,v分量表示经向风。u为正,表示西风,风向为270°;v为正,表示南风,风向为180°。示意图如下所示,
- 用Python画一只溜达小狗——turtle库基础入门
编程大本营
pythonpython
一只脑门有点方的小狗,其实还可以把脑门和后脑勺完善一下,更圆润一些。但这样也挺可爱,就保有这样不完美但独一无二的它吧。绘制过程主要就是拼接和调整圆弧,尽量做到过度自然。小狗的绘制主要使用了turtle库的circle()函数,初接触时可能会略有不适应,但用起来之后会发现它很强大!对circle()函数用法还不熟悉的同学可参考这篇博客:《如何用Python画一只兔子——turtle库circle()
- 【yolov8】模型导出----pytorch导出为onnx模型
栗子风暴
YOLOpytorch人工智能深度学习
【yolov8】模型导出一、为什么要使用yolo的导出模式二、确保安装必要的库:三、yolov8模型导出3.1不同格式配置参数3.2导出格式四、导出模型性能优化4.1使用TensorRT导出模型有什么好处?4.2导出YOLOv8模型时,如何启用INT8量化?4.3为什么输出模型时动态输入尺寸很重要?4.4优化模型性能需要考虑哪些关键的导出参数?五、问题六、疑问训练模型的最终目标是将其部署到实际应用
- PCL 最小二乘拟合空间曲线
点云侠
点云进阶算法c++计算机视觉3d开发语言
目录一、曲线拟合1、算法原理2、参考文献二、代码实现三、结果展示四、测试数据本文由CSDN点云侠原创,原文链接。如果你不是在点云侠的博客中看到该文章,那么此处便是不要脸的爬虫与GPT。博客长期更新,最近一次更新时间为:2024年7月14日。①代码在PCL1.14.1中运行;②完善代码;③新增标准测试数据一、曲线拟合1、算法原理 电力线三维重建指将提取得到的单根电力线进行精确矢量化。在理想情况下,
- 【AGI】DeepSeek开源周:The whale is making waves!
LeeZhao@
AIGC重塑生活神器agi开源人工智能AIGC生活语言模型
DeepSeek开源周:Thewhaleismakingwaves!思维火花引言一、DeepSeek模型体系的技术演进1.通用语言模型:DeepSeek-V3系列2.推理优化模型:DeepSeek-R1系列3.多模态模型:Janus系列二、开源周三大工具库的技术解析1.FlashMLA:解码效率的极限突破(2025.02.24)2.DeepEP:MoE通信范式的重构(2025.02.25)3.De
- 【AGI】中国大模型扛把子:通义家族
LeeZhao@
AIGC重塑生活神器agi人工智能AIGC面试自然语言处理语言模型
中国大模型扛把子:通义家族引言一、通义千问的技术架构与模型谱系二、技术突破与性能优势三、开源生态与行业影响四、未来展望:从“千问时代”到通用智能五、通义家族大模型列表(1)多模态大模型(2)大语言模型结语引言在人工智能大模型领域,中国科技企业正以惊人的速度突破技术边界。阿里云推出的**通义千问(Qwen)**系列大模型,凭借其多层次的技术架构、多样化的模型生态及开源战略,已成为全球AI领域的重要标
- AdaBoost算法
Mr终游
机器学习算法决策树
目录一、核心原理:二、算法步骤三、关键优势:四.局限与解决五、代码示例(鸢尾花数据集)AdaBoost(AdaptiveBoosting)是一种经典的集成学习算法,通过组合多个弱分类器(如决策树)来构建强分类器。其核心思想是通过迭代优化残差(错误)和动态调整样本权重,逐步提升模型性能。以下是对AdaBoost的简明总结和关键要点:一、核心原理:提升法:通过顺序训练多个弱分类器,每轮专注修正前一个模
- 模块15.常用API
.又是新的一天.
Java基础算法java
文章目录模块15.常用API第一章.Math类1.Math类介绍2.Math类方法第二章.BigInteger1.BigInteger介绍2.BigInteger使用第三章.BigDecimal类1.BigDecimal介绍2.BigDecimal使用3.BigDecimal除法过时方法解决第四章.Date日期类1.Date类的介绍2.Date类的使用3.Date类的常用方法第五章.Calenda
- 【华为OD技术面试手撕真题】113、组合总和 | 手撕真题+思路参考+代码解析(C & C++ & Java & Python & JS)
KJ.JK
华为OD技术面试手撕真题华为od面试c语言华为od机试E卷华为od机试真题组合总和
文章目录一、题目题目描述样例1二、代码参考C语言思路C语言代码C++语言思路C++代码Java语言思路Java代码Python语言思路Python代码JS语言思路JS代码作者:KJ.JK个人博客首页:KJ.JK专栏介绍:本专栏更新每年华为OD机试的高频手撕代码题,每个题目都会使用五种语言进行解答(C&C++&Java&Python&JS),思路分析都非常详细,争取实现最低的时间复杂度和高通过率,每
- llama_deploy
伊织code
#文档翻译llamallama_deployllamaindex
本文于240924翻译整理自:https://docs.llamaindex.ai/en/stable/module_guides/workflow/deployment/文章目录一、关于`llama_deploy`为什么使用`llama_deploy`?等等,`llama-agents`在哪里?二、入门1、安装2、高级部署3、部署核心系统4、部署工作流5、与部署交互6、部署嵌套工作流三、一个`l
- python: DDD using postgeSQL and SQL Server
geovindu
Pythonpythonjava前端数据库postgresqlsqlservermssql
postgreSQL注意:#psycopg2驱动的连接字符串#engine=create_engine('postgresql://post:geovindu@localhost:5433/TechnologyGame')#Session=sessionmaker(bind=engine)#使用psycopg3驱动的连接字符串#engine=create_engine('postgresql+ps
- Bert学习笔记
缓释多巴胺。
大模型相关知识语言模型bert
一、Bert架构BERT使用了双向的TransformerGPT使用从左到右的单向信息ELMo把单独训练的从左到右及从右到左的LSTM模型进行合并二、Bert预训练任务2.1遮蔽语言模型MLM任务:随机屏蔽(masking)部分输入token,然后只预测那些被屏蔽的token。问题:预训练任务与微调任务不一致原因:在finetuning期间从未看到[MASK]token,预训练和finetunin
- 分布式事务 CAP三进二和Base定理
柿子加油努力
DistributedTransactions分布式
关系型数据库遵循ACID原则事务在英文中是transaction,和现实世界中的交易很类似,它有如下四个特性:1、A(Atomicity)原子性原子性很容易理解,也就是说事务里的所有操作要么全部做完,要么都不做,事务成功的条件是事务里的所有操作都成功,只要有一个操作失败,整个事务就失败,需要回滚。比如银行转账,从A账户转100元至B账户,分为两个步骤:1)从A账户取100元;2)存入100元至B账
- 深入探究LLamaFactory推理DeepSeek蒸馏模型时无法展示<think>思考过程的问题
羊城迷鹿
DeepSeekLLama-Factory思维链
文章目录问题背景初始测试与问题发现LLaMAFactory测试结果对照实验:Ollama测试系统性排查与解决方案探索1.尝试更换模板2.深入研究官方文档3.自定义模板实现优化界面展示:实现思考过程的可视化实现方法参数调整影响分析实验一实验二进入大模型应用与实战专栏|查看更多专栏内容问题背景最近在本地环境中部署了DeepSeek-R1-Distill-Qwen-1.5B,即由Qwen2.5-Math
- DeepSeek安装部署笔记(二)
山哥ol
笔记运维python
Bat批处理文件的编写第五步启动openWebUI的批处理编写1、下面的代码,复制到文本文件,再改扩展名2、这样,在桌面直接双击此文件运行第五步启动openWebUI的批处理编写1、下面的代码,复制到文本文件,再改扩展名@echooffREM关闭回显使界面更简洁setCONDA_ROOT=D:\condasetENV_NAME=openwebuiREM使用’/K’参数保持CMD窗口不关闭,并执行后
- C++ 泛型编程
四代目 水门
C++学习笔记c++开发语言
C++泛型编程一、泛型编程基础1.核心概念实现算法与数据结构的分离基于模板技术(函数模板/类模板)本质:类型参数化,减少重复代码典型应用:STL容器、迭代器、算法2.类型本质内存布局的抽象不同类型对应不同的内存分配策略二、函数模板1.基本语法cpptemplate//或template返回类型函数名(参数列表){//函数体}2.关键特性支持隐式推导和显式指定类型可重载(包括与普通函数重载)可声明为
- springboot整合rabbitMQ
twx95
java-rabbitmqspringbootrabbitmq
安装rabbitMQ虚拟机或者服务器上安装我这里使用的是vm虚拟机做演示第一步:安装docker参考linux安装docker-CSDN博客第二步:拉取rabbitMQ镜像3-management(镜像版本)dockerpullrabbitmq:3-management查看镜像是否拉取成功dockerimages第三步:运行rabbitMQdockerrun\-eRABBITMQ_DEFAULT_
- LeetCode-Hot100-006三数之和
YQ_ZJH
LeetCode100题leetcode数据结构排序算法算法c++蓝桥杯java
思路先排序解决重复的问题。再三重循环遍历,但是第二重和第三重使用双指针的做法,复杂度降低为O(n2)O(n^2)O(n2)。代码本次代码来自于力扣官方题解评论区,非本人原创,请注意classSolution{publicList>threeSum(int[]nums){Arrays.sort(nums);//先排序List>res=newArrayList0&&nums[i]==nums[i-1]
- [数据结构] [C++ STL] vector使用详解
高亚奇
数据结构数据结构c++开发语言
一、概述vector(向量):是一种序列式容器,事实上和数组差不多,但它比数组更优越。一般来说数组不能动态拓展,因此在程序运行的时候不是浪费内存,就是造成越界。而vector正好弥补了这个缺陷,它的特征是相当于可分配拓展的数组(动态数组),它的随机访问快,在中间插入和删除慢,但在末端插入和删除快。二、定义及初始化使用之前必须加相应容器的头文件:#include//vector属于std命名域的,因
- React 基础教程
阿贾克斯的黎明
前端react.js前端前端框架
目录React基础教程一、React简介二、安装和设置三、创建第一个React组件(一)函数式组件(二)类组件四、渲染组件五、组件的属性和状态(一)属性(Props)(二)状态(State)六、组件的生命周期方法七、事件处理八、总结React是一个用于构建用户界面的JavaScript库。它以高效、灵活和可维护性而受到广泛的欢迎。本教程将介绍React的基础知识,帮助你快速上手React开发。一、
- 【python】软件更新:用conda或Poetry
无水先生
AI原理和python实现python指南和应用人工智能综合pythonconda开发语言
一、说明在实现anancoda的软件更新问题,需要明确几个问题:1)是python包吗?2)是C++包吗?更新的方法有别。python包可以pip访问。C++包必须是conda访问。二、更新C++包的循环依赖问题如果在Windows10上的Ananconda3.7上安装和更新软件包。运行代码时:condaupdate--all或者condainstallpandas收到以下错误:RemoveErr
- Spring4.1新特性——Spring MVC增强
jinnianshilongnian
spring 4.1
目录
Spring4.1新特性——综述
Spring4.1新特性——Spring核心部分及其他
Spring4.1新特性——Spring缓存框架增强
Spring4.1新特性——异步调用和事件机制的异常处理
Spring4.1新特性——数据库集成测试脚本初始化
Spring4.1新特性——Spring MVC增强
Spring4.1新特性——页面自动化测试框架Spring MVC T
- mysql 性能查询优化
annan211
javasql优化mysql应用服务器
1 时间到底花在哪了?
mysql在执行查询的时候需要执行一系列的子任务,这些子任务包含了整个查询周期最重要的阶段,这其中包含了大量为了
检索数据列到存储引擎的调用以及调用后的数据处理,包括排序、分组等。在完成这些任务的时候,查询需要在不同的地方
花费时间,包括网络、cpu计算、生成统计信息和执行计划、锁等待等。尤其是向底层存储引擎检索数据的调用操作。这些调用需要在内存操
- windows系统配置
cherishLC
windows
删除Hiberfil.sys :使用命令powercfg -h off 关闭休眠功能即可:
http://jingyan.baidu.com/article/f3ad7d0fc0992e09c2345b51.html
类似的还有pagefile.sys
msconfig 配置启动项
shutdown 定时关机
ipconfig 查看网络配置
ipconfig /flushdns
- 人体的排毒时间
Array_06
工作
========================
|| 人体的排毒时间是什么时候?||
========================
转载于:
http://zhidao.baidu.com/link?url=ibaGlicVslAQhVdWWVevU4TMjhiKaNBWCpZ1NS6igCQ78EkNJZFsEjCjl3T5EdXU9SaPg04bh8MbY1bR
- ZooKeeper
cugfy
zookeeper
Zookeeper是一个高性能,分布式的,开源分布式应用协调服务。它提供了简单原始的功能,分布式应用可以基于它实现更高级的服务,比如同步, 配置管理,集群管理,名空间。它被设计为易于编程,使用文件系统目录树作为数据模型。服务端跑在java上,提供java和C的客户端API。 Zookeeper是Google的Chubby一个开源的实现,是高有效和可靠的协同工作系统,Zookeeper能够用来lea
- 网络爬虫的乱码处理
随意而生
爬虫网络
下边简单总结下关于网络爬虫的乱码处理。注意,这里不仅是中文乱码,还包括一些如日文、韩文 、俄文、藏文之类的乱码处理,因为他们的解决方式 是一致的,故在此统一说明。 网络爬虫,有两种选择,一是选择nutch、hetriex,二是自写爬虫,两者在处理乱码时,原理是一致的,但前者处理乱码时,要看懂源码后进行修改才可以,所以要废劲一些;而后者更自由方便,可以在编码处理
- Xcode常用快捷键
张亚雄
xcode
一、总结的常用命令:
隐藏xcode command+h
退出xcode command+q
关闭窗口 command+w
关闭所有窗口 command+option+w
关闭当前
- mongoDB索引操作
adminjun
mongodb索引
一、索引基础: MongoDB的索引几乎与传统的关系型数据库一模一样,这其中也包括一些基本的优化技巧。下面是创建索引的命令: > db.test.ensureIndex({"username":1}) 可以通过下面的名称查看索引是否已经成功建立: &nbs
- 成都软件园实习那些话
aijuans
成都 软件园 实习
无聊之中,翻了一下日志,发现上一篇经历是很久以前的事了,悔过~~
断断续续离开了学校快一年了,习惯了那里一天天的幼稚、成长的环境,到这里有点与世隔绝的感觉。不过还好,那是刚到这里时的想法,现在感觉在这挺好,不管怎么样,最要感谢的还是老师能给这么好的一次催化成长的机会,在这里确实看到了好多好多能想到或想不到的东西。
都说在外面和学校相比最明显的差距就是与人相处比较困难,因为在外面每个人都
- Linux下FTP服务器安装及配置
ayaoxinchao
linuxFTP服务器vsftp
检测是否安装了FTP
[root@localhost ~]# rpm -q vsftpd
如果未安装:package vsftpd is not installed 安装了则显示:vsftpd-2.0.5-28.el5累死的版本信息
安装FTP
运行yum install vsftpd命令,如[root@localhost ~]# yum install vsf
- 使用mongo-java-driver获取文档id和查找文档
BigBird2012
driver
注:本文所有代码都使用的mongo-java-driver实现。
在MongoDB中,一个集合(collection)在概念上就类似我们SQL数据库中的表(Table),这个集合包含了一系列文档(document)。一个DBObject对象表示我们想添加到集合(collection)中的一个文档(document),MongoDB会自动为我们创建的每个文档添加一个id,这个id在
- JSONObject以及json串
bijian1013
jsonJSONObject
一.JAR包简介
要使程序可以运行必须引入JSON-lib包,JSON-lib包同时依赖于以下的JAR包:
1.commons-lang-2.0.jar
2.commons-beanutils-1.7.0.jar
3.commons-collections-3.1.jar
&n
- [Zookeeper学习笔记之三]Zookeeper实例创建和会话建立的异步特性
bit1129
zookeeper
为了说明问题,看个简单的代码,
import org.apache.zookeeper.*;
import java.io.IOException;
import java.util.concurrent.CountDownLatch;
import java.util.concurrent.ThreadLocal
- 【Scala十二】Scala核心六:Trait
bit1129
scala
Traits are a fundamental unit of code reuse in Scala. A trait encapsulates method and field definitions, which can then be reused by mixing them into classes. Unlike class inheritance, in which each c
- weblogic version 10.3破解
ronin47
weblogic
版本:WebLogic Server 10.3
说明:%DOMAIN_HOME%:指WebLogic Server 域(Domain)目录
例如我的做测试的域的根目录 DOMAIN_HOME=D:/Weblogic/Middleware/user_projects/domains/base_domain
1.为了保证操作安全,备份%DOMAIN_HOME%/security/Defa
- 求第n个斐波那契数
BrokenDreams
今天看到群友发的一个问题:写一个小程序打印第n个斐波那契数。
自己试了下,搞了好久。。。基础要加强了。
&nbs
- 读《研磨设计模式》-代码笔记-访问者模式-Visitor
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
import java.util.ArrayList;
import java.util.List;
interface IVisitor {
//第二次分派,Visitor调用Element
void visitConcret
- MatConvNet的excise 3改为网络配置文件形式
cherishLC
matlab
MatConvNet为vlFeat作者写的matlab下的卷积神经网络工具包,可以使用GPU。
主页:
http://www.vlfeat.org/matconvnet/
教程:
http://www.robots.ox.ac.uk/~vgg/practicals/cnn/index.html
注意:需要下载新版的MatConvNet替换掉教程中工具包中的matconvnet:
http
- ZK Timeout再讨论
chenchao051
zookeepertimeouthbase
http://crazyjvm.iteye.com/blog/1693757 文中提到相关超时问题,但是又出现了一个问题,我把min和max都设置成了180000,但是仍然出现了以下的异常信息:
Client session timed out, have not heard from server in 154339ms for sessionid 0x13a3f7732340003
- CASE WHEN 用法介绍
daizj
sqlgroup bycase when
CASE WHEN 用法介绍
1. CASE WHEN 表达式有两种形式
--简单Case函数
CASE sex
WHEN '1' THEN '男'
WHEN '2' THEN '女'
ELSE '其他' END
--Case搜索函数
CASE
WHEN sex = '1' THEN
- PHP技巧汇总:提高PHP性能的53个技巧
dcj3sjt126com
PHP
PHP技巧汇总:提高PHP性能的53个技巧 用单引号代替双引号来包含字符串,这样做会更快一些。因为PHP会在双引号包围的字符串中搜寻变量, 单引号则不会,注意:只有echo能这么做,它是一种可以把多个字符串当作参数的函数译注: PHP手册中说echo是语言结构,不是真正的函数,故把函数加上了双引号)。 1、如果能将类的方法定义成static,就尽量定义成static,它的速度会提升将近4倍
- Yii框架中CGridView的使用方法以及详细示例
dcj3sjt126com
yii
CGridView显示一个数据项的列表中的一个表。
表中的每一行代表一个数据项的数据,和一个列通常代表一个属性的物品(一些列可能对应于复杂的表达式的属性或静态文本)。 CGridView既支持排序和分页的数据项。排序和分页可以在AJAX模式或正常的页面请求。使用CGridView的一个好处是,当用户浏览器禁用JavaScript,排序和分页自动退化普通页面请求和仍然正常运行。
实例代码如下:
- Maven项目打包成可执行Jar文件
dyy_gusi
assembly
Maven项目打包成可执行Jar文件
在使用Maven完成项目以后,如果是需要打包成可执行的Jar文件,我们通过eclipse的导出很麻烦,还得指定入口文件的位置,还得说明依赖的jar包,既然都使用Maven了,很重要的一个目的就是让这些繁琐的操作简单。我们可以通过插件完成这项工作,使用assembly插件。具体使用方式如下:
1、在项目中加入插件的依赖:
<plugin>
- php常见错误
geeksun
PHP
1. kevent() reported that connect() failed (61: Connection refused) while connecting to upstream, client: 127.0.0.1, server: localhost, request: "GET / HTTP/1.1", upstream: "fastc
- 修改linux的用户名
hongtoushizi
linuxchange password
Change Linux Username
更改Linux用户名,需要修改4个系统的文件:
/etc/passwd
/etc/shadow
/etc/group
/etc/gshadow
古老/传统的方法是使用vi去直接修改,但是这有安全隐患(具体可自己搜一下),所以后来改成使用这些命令去代替:
vipw
vipw -s
vigr
vigr -s
具体的操作顺
- 第五章 常用Lua开发库1-redis、mysql、http客户端
jinnianshilongnian
nginxlua
对于开发来说需要有好的生态开发库来辅助我们快速开发,而Lua中也有大多数我们需要的第三方开发库如Redis、Memcached、Mysql、Http客户端、JSON、模板引擎等。
一些常见的Lua库可以在github上搜索,https://github.com/search?utf8=%E2%9C%93&q=lua+resty。
Redis客户端
lua-resty-r
- zkClient 监控机制实现
liyonghui160com
zkClient 监控机制实现
直接使用zk的api实现业务功能比较繁琐。因为要处理session loss,session expire等异常,在发生这些异常后进行重连。又因为ZK的watcher是一次性的,如果要基于wather实现发布/订阅模式,还要自己包装一下,将一次性订阅包装成持久订阅。另外如果要使用抽象级别更高的功能,比如分布式锁,leader选举
- 在Mysql 众多表中查找一个表名或者字段名的 SQL 语句
pda158
mysql
在Mysql 众多表中查找一个表名或者字段名的 SQL 语句:
方法一:SELECT table_name, column_name from information_schema.columns WHERE column_name LIKE 'Name';
方法二:SELECT column_name from information_schema.colum
- 程序员对英语的依赖
Smile.zeng
英语程序猿
1、程序员最基本的技能,至少要能写得出代码,当我们还在为建立类的时候思考用什么单词发牢骚的时候,英语与别人的差距就直接表现出来咯。
2、程序员最起码能认识开发工具里的英语单词,不然怎么知道使用这些开发工具。
3、进阶一点,就是能读懂别人的代码,有利于我们学习人家的思路和技术。
4、写的程序至少能有一定的可读性,至少要人别人能懂吧...
以上一些问题,充分说明了英语对程序猿的重要性。骚年
- Oracle学习笔记(8) 使用PLSQL编写触发器
vipbooks
oraclesql编程活动Access
时间过得真快啊,转眼就到了Oracle学习笔记的最后个章节了,通过前面七章的学习大家应该对Oracle编程有了一定了了解了吧,这东东如果一段时间不用很快就会忘记了,所以我会把自己学习过的东西做好详细的笔记,用到的时候可以随时查找,马上上手!希望这些笔记能对大家有些帮助!
这是第八章的学习笔记,学习完第七章的子程序和包之后