- 奇数和偶数的求法c++
今天给大家分享一个奇偶数的求法:#includeusingnamespacestd;intmain(){inta=0;cin>>a;if(a%2==0){cout>a;这是输入和声明if(a%2==0){cout<<"偶数";}else{cout<<"奇数";}这部分是判断的点个赞吧,求求了~
- 【Unity&AssetBundle】AssetBundle管理器
usingSystem;usingSystem.Collections;usingSystem.Collections.Generic;usingSystem.IO;usingSystem.Threading.Tasks;usingUnityEngine;usingUnityEngine.Events;//////AB包管理器///publicclassAssetBundle_Manager_:S
- 【Unity笔记02】订阅事件-自动开门
流程当玩家移动到触发区域的时候,门自动打开事件系统usingSystem;usingSystem.Collections;usingSystem.Collections.Generic;usingUnityEngine;publicclassEventSystem:MonoBehaviour{publicstaticEventSystemInstance{get;privateset;}publi
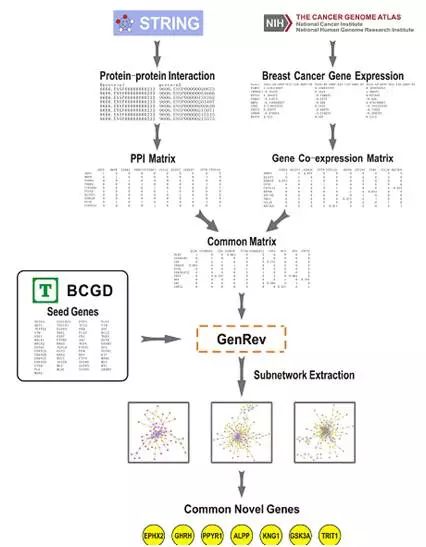
- 基于迁移学习的多视图卷积神经网络在乳腺超声自动分类中的应用
despacito,
论文精读-乳腺超声分类
BREASTCANCERCLASSIFICATIONINAUTOMATEDBREASTULTRASOUNDUSINGMULTIVIEWCONVOLUTIONALNEURALNETWORKWITHTRANSFERLEARNINGYIWANG,*,1EUNJUNGCHOI,y,1YOUNHEECHOI,*HAOZHANG,*GONGYONGJIN,yandSEOK-BUMKO*TAGGEDEND*De
- 快速排序算法
追烽少年x
数据结构数据结构
快速排序算法快速排序是一种高效的排序算法,其核心思想是通过分治法将数组分成两部分,一部分小于某个基准值,另一部分大于基准值,然后递归地对这两部分进行排序。以下是快速排序算法的C++实现:快速排序的C++实现代码:#include#includeusingnamespacestd;voidSwap(int&a,int&b){intnTemp=a;a=b;b=nTemp;}intPartition(v
- C - Large Queue
queenlll
c语言开发语言
地址:C-LargeQueue#includeusingnamespacestd;constintN=1e5+10;voidsolve(){intn;cin>>n;queue>q;while(n--){intt;cin>>t;if(t==1){longlongc,x;cin>>c>>x;q.push({x,c});}else{longlongk;cin>>k;longlongsum=0;while
- c++求同构数
*Allen*
c++算法数据结构
题目描述所谓同构数是指这样的数,即它出现在它的平方数的右端。例如,5的平方是25(即5×5=25),5是25右端的数,那么5就是同构数。又如,25的平方是625(即25×25=625),同理25也是同构数。找出通过键盘输入的两个正整数N和M(0usingnamespacestd;intn,m,t,s,a[100],b[100],sum,s1,s2,k;intmain(){cin>>n>>m;for
- 【华为od刷题(C++)】HJ35 蛇形矩阵(指针)
m0_64866459
华为odc++链表
我的代码1:#includeusingnamespacestd;intmain(){introw;//row:定义了矩阵的行数(和列数,实际上是一个正方形矩阵)while(cin>>row){//这个循环会持续执行,直到输入流被结束//每次读取一个整数并赋值给row,程序就开始执行填充操作int**a=newint*[row];//动态地为一个二维数组(a)的行分配内存/*这里a是一个指向指针的指
- undo tablespace的恢复, database能不关闭最好不要关闭
jnrjian
oracle数据库
Appliesto:EnterpriseManagerforOracleDatabase-Version9.2.0.1to11.2.0.4Informationinthisdocumentappliestoanyplatform.PurposeProblemDescription:====================Thisisarecoveryscenarioinwhichadatafile
- 【华为od刷题(C++)】HJ33 整数与IP地址间的转换
m0_64866459
华为odc++链表
我的代码:#include//这个头文件提供了输入输出流的功能,使得我们能够使用cin和cout来进行输入输出usingnamespacestd;//可以直接使用标准命名空间std中的功能//比如cout和cin,而不需要每次都写出std::intmain(){longlonginta,b,c,d;//a,b,c,d:这四个变量用来存储IP地址的四个部分//分别代表IP地址中的四个字节longlo
- 【Unity笔记01】基于单例模式的简单UI框架
单例模式的UIManagerusingSystem.Collections;usingSystem.Collections.Generic;usingUnityEngine;publicclassUIManager{privatestaticUIManager_instance;publicDictionarypathDict;publicDictionaryprefabDict;publicDi
- AtCoder ABC344 A-E题解
seanli1008
#AtCoder题解题解
传送门:ABC344咕了一个周的题解。省流:D>E+不可以总司令专场ohhhhhhhhhhhhhhhhhhhhhhhhhhProblemA:善用STL。#includeusingnamespacestd;intmain(){stringS;cin>>S;inti=S.find('|');j=S.find('|',i+1);coutusingnamespacestd;intmain(){vector
- OL9.4安装19.27RAC记录
李曰福
Oracleoracle
更新-2025-06-09在上一次安装时,文档OracleDatabase19cProactivePatchInformation(DocID2521164.1)未给出GI_MRP的链接。安装DB_MRP失败。在PrimaryNoteforDatabaseQuarterlyReleaseUpdates(DocID888.1)找到了GI_MRP.上传到/stage目录后,并修改setup.ini,加
- 交互式模式下启用 SAI(SoC 架构信息)的操作解析
一、交互式模式启用SAI的核心逻辑与流程在交互式模式下启用SAI(SoCArchitectureInformation)是通过命令行逐行输入指令,动态构建芯片架构模型并实时查看反馈的操作方式。其核心优势在于灵活性高,适合设计早期的单场景探索与命令调试,具体流程如下:二、关键操作步骤与命令详解启动SAI功能(read_sai)作用:启动物理设计环境并进入SAI交互模式。命令格式:tclread_sa
- 利用 SoC 架构信息 (SAI) 创建自动网表的解析
weixin_45371279
innovus
基于SAI(SoCArchitectureInformation)创建网表是在无完整电路网表时启动物理设计的关键技术,通过解析架构描述并生成等效网表,使Innovus能够执行布局规划与分析。以下是核心功能与实现逻辑的详细说明:一、SAI自动网表生成的技术背景与目标当设计早期缺乏RTL或门级网表时,SAI自动网表生成通过抽象模型具体化的方式,将架构描述转换为物理设计工具可识别的网表结构,实现:提前启
- 五天速成C++-----第五天
别睡了.
五天速成C++c++开发语言算法
面向对象核心1.继承定义:在已存在类的基础上,创建新的类。会拥有原有类的一些特性。通常会在原有类基础上做修改和增加操作。已经存在的类成为父类或基类新创建的类称为子类或派生类#includeusingnamespacestd;classFather{public:stringfirst_name="李";voidwork(){coutusingnamespacestd;classFather{pub
- 机器视觉通用平台之拟合圆工具类
小治视觉
算法visualstudioc#
usingSystem;usingSystem.Collections.Generic;usingSystem.Linq;usingSystem.Runtime.InteropServices;usingSystem.Text;usingSystem.Threading.Tasks;usingCvBase;usingSystem.IO;usingSystem.Drawing.Drawing2D;u
- LeetCode-5.最长回文子串 C++实现
一.问题描述给你一个字符串s,找到s中最长的回文子串(如果字符串向前和向后读都相同,则它满足回文性。)。示例1:输入:s="babad"输出:"bab"解释:"aba"同样是符合题意的答案。示例2:输入:s="cbbd"输出:"bb"提示:1usingnamespacestd;classSolution{public:stringlongestPalindrome(strings){intn=s.
- AtCoder Beginner Contest 412(ABCDE)
前言回来喽!!前一阵子期末周快复习疯了,接下来还想准备数学建模,感觉高中都没这么忙过T^T。中间参加了一场百度之星的比赛,只AC了两题,感觉好难啊还是太菜了,希望能混个牌呜呜呜。图论和数论题好难,还得多练啊……一、A-TaskFailedSuccessfully#includeusingnamespacestd;typedeflonglongll;typedefpairpii;voidsolve(
- greenplum查询超时_Greenplum常用SQL查询
抽空网上收罗Greenplum常用SQL查询语句整理备忘。欢迎各位留言补充。都是SQL命令以及数据字典的使用。熟悉数据字典非常重要。三个重要的schema:pg_catalog,pg_toolkit,information_schema,其中information_schema中的数据字典都在视图中目录一、查看表某模式所有分布键信息二、数据库运行状态查询管理1.greenplum查询正在运行的sq
- 【华为od刷题(C++)】HJ30 字符串合并处理
m0_64866459
华为odc++链表
我的代码:#include//用于输入输出流#include//用于字符串处理#include//用于动态数组的处理#include//包含排序等常见算法#include//用于字符串流的处理,可以将数据从字符串流中提取#include//提供字符处理函数,如isdigit、isalpha等#include//提供位集处理,能够将数字转换为二进制表示usingnamespacestd;charbi
- 【数据结构】栈
会的全对٩(ˊᗜˋ*)و
数据结构数据结构经验分享栈
要求:熟悉栈的定义,栈的特点以及栈的基本操作。能够根据实际情况选择合适的存储结构,解决实际问题。对任意给定的一个中缀算术表达式输出等价的后缀形式。代码实现:#include#include#includeusingnamespacestd;intprio(charop){//给运算符优先级排序intt;if(op=='*'||op=='/')t=2;if(op=='+'||op=='-')t=1;
- 踏入真实:具身智能与物理世界的认知交响
当大型语言模型在文本的海洋中纵横捭阖,生成式AI在数字画布上挥洒创意时,人工智能仍有一个根本性的疆域尚未完全征服——真实的三维物理世界。理解一个苹果,不能仅靠词向量坐标;学会行走,无法通过阅读说明书达成;在拥挤的街道导航,远非处理符号逻辑那般简单。智能的进化,自生命诞生之初,便与具身性(Embodiment)和环境交互(Interaction)密不可分。我们的认知、学习、乃至意识的雏形,都源于身体
- TypeScript-Babel-Starter 项目常见问题解决方案
董灵辛Dennis
TypeScript-Babel-Starter项目常见问题解决方案TypeScript-Babel-StarterAsamplesetupusingBabelCLItobuildTypeScriptcode,andusingTypeScriptfortype-checking.项目地址:https://gitcode.com/gh_mirrors/ty/TypeScript-Babel-Star
- TypeScript-Babel-Starter 项目推荐
周琰策Scott
TypeScript-Babel-Starter项目推荐TypeScript-Babel-StarterAsamplesetupusingBabelCLItobuildTypeScriptcode,andusingTypeScriptfortype-checking.项目地址:https://gitcode.com/gh_mirrors/ty/TypeScript-Babel-StarterTyp
- 线性代数向量内积_向量的点积| 使用Python的线性代数
cumubi7453
python线性代数机器学习numpy算法
线性代数向量内积Prerequisite:LinearAlgebra|DefiningaVector先决条件:线性代数|定义向量Linearalgebraisthebranchofmathematicsconcerninglinearequationsbyusingvectorspacesandthroughmatrices.Inotherwords,avectorisamatrixinn-dim
- DAY08 算法训练营| 字符串part01
天空的孩子
算法
344.反转字符串-力扣(LeetCode)字符串和数组算法题目思路类似反转字符串是经典双指针法(回忆反转链表,有序数组的平方,三数之和,四数之和)classSolution{public:voidreverseString(vector&s){len=s.length();for(inti=0,j=s.size()-1;iusingnamespacestd;intmain(){strings;/
- 【Unity开发】Unity实现glb模型上传到场景中使用功能
龚子亦
Unityunity游戏引擎
一、效果展示点击按钮上传glb格式的模型,进行后续移动、旋转和缩放操作,上传的模型会出现在原点(0,0,0)位置演示视频:Unity实现glb模型上传到场景中使用功能二、环境要求需要下载安装用于加载glb模型的插件,下载好后直接拖入项目中进行使用下载链接:https://github.com/Siccity/GLTFUtility/releases三、实现完整代码usingUnityEngine;
- 【Unity开发】Unity实现对模型移动、缩放、旋转操作的功能
龚子亦
Unityunity游戏引擎
一、效果展示Unity实现模型移动、旋转和缩放功能通过鼠标实现对模型的不同操作鼠标中键点击,模型变为高亮状态,表示选中状态,再次点击其他地方,则取消高亮,表示未选中状态鼠标中键上下滚动,实现模型的缩放鼠标左键点击或长按,实现模型的移动鼠标右键点击,左右滑动,实现模型的水平旋转;同时按下shift键,上下滑动,实现模型垂直旋转二、实现完整代码usingSystem.Collections;using
- 【华为od刷题(C++)】HJ17 坐标移动(continue语句、break语句)
m0_64866459
华为odc++开发语言
我的代码:#include//用于输入输出#include//用于处理字符串#include//用于存储动态数组usingnamespacestd;intmain(){strings;//用于存储输入的字符串cin>>s;//输入字符串intlen=s.size();//获取字符串的长度vectorstr;//用来存储从输入字符串中提取出的子字符串(每个指令部分)intx=0,y=0;//设置初始
- 关于旗正规则引擎中的MD5加密问题
何必如此
jspMD5规则加密
一般情况下,为了防止个人隐私的泄露,我们都会对用户登录密码进行加密,使数据库相应字段保存的是加密后的字符串,而非原始密码。
在旗正规则引擎中,通过外部调用,可以实现MD5的加密,具体步骤如下:
1.在对象库中选择外部调用,选择“com.flagleader.util.MD5”,在子选项中选择“com.flagleader.util.MD5.getMD5ofStr({arg1})”;
2.在规
- 【Spark101】Scala Promise/Future在Spark中的应用
bit1129
Promise
Promise和Future是Scala用于异步调用并实现结果汇集的并发原语,Scala的Future同JUC里面的Future接口含义相同,Promise理解起来就有些绕。等有时间了再仔细的研究下Promise和Future的语义以及应用场景,具体参见Scala在线文档:http://docs.scala-lang.org/sips/completed/futures-promises.html
- spark sql 访问hive数据的配置详解
daizj
spark sqlhivethriftserver
spark sql 能够通过thriftserver 访问hive数据,默认spark编译的版本是不支持访问hive,因为hive依赖比较多,因此打的包中不包含hive和thriftserver,因此需要自己下载源码进行编译,将hive,thriftserver打包进去才能够访问,详细配置步骤如下:
1、下载源码
2、下载Maven,并配置
此配置简单,就略过
- HTTP 协议通信
周凡杨
javahttpclienthttp通信
一:简介
HTTPCLIENT,通过JAVA基于HTTP协议进行点与点间的通信!
二: 代码举例
测试类:
import java
- java unix时间戳转换
g21121
java
把java时间戳转换成unix时间戳:
Timestamp appointTime=Timestamp.valueOf(new SimpleDateFormat("yyyy-MM-dd HH:mm:ss").format(new Date()))
SimpleDateFormat df = new SimpleDateFormat("yyyy-MM-dd hh:m
- web报表工具FineReport常用函数的用法总结(报表函数)
老A不折腾
web报表finereport总结
说明:本次总结中,凡是以tableName或viewName作为参数因子的。函数在调用的时候均按照先从私有数据源中查找,然后再从公有数据源中查找的顺序。
CLASS
CLASS(object):返回object对象的所属的类。
CNMONEY
CNMONEY(number,unit)返回人民币大写。
number:需要转换的数值型的数。
unit:单位,
- java jni调用c++ 代码 报错
墙头上一根草
javaC++jni
#
# A fatal error has been detected by the Java Runtime Environment:
#
# EXCEPTION_ACCESS_VIOLATION (0xc0000005) at pc=0x00000000777c3290, pid=5632, tid=6656
#
# JRE version: Java(TM) SE Ru
- Spring中事件处理de小技巧
aijuans
springSpring 教程Spring 实例Spring 入门Spring3
Spring 中提供一些Aware相关de接口,BeanFactoryAware、 ApplicationContextAware、ResourceLoaderAware、ServletContextAware等等,其中最常用到de匙ApplicationContextAware.实现ApplicationContextAwaredeBean,在Bean被初始后,将会被注入 Applicati
- linux shell ls脚本样例
annan211
linuxlinux ls源码linux 源码
#! /bin/sh -
#查找输入文件的路径
#在查找路径下寻找一个或多个原始文件或文件模式
# 查找路径由特定的环境变量所定义
#标准输出所产生的结果 通常是查找路径下找到的每个文件的第一个实体的完整路径
# 或是filename :not found 的标准错误输出。
#如果文件没有找到 则退出码为0
#否则 即为找不到的文件个数
#语法 pathfind [--
- List,Set,Map遍历方式 (收集的资源,值得看一下)
百合不是茶
listsetMap遍历方式
List特点:元素有放入顺序,元素可重复
Map特点:元素按键值对存储,无放入顺序
Set特点:元素无放入顺序,元素不可重复(注意:元素虽然无放入顺序,但是元素在set中的位置是有该元素的HashCode决定的,其位置其实是固定的)
List接口有三个实现类:LinkedList,ArrayList,Vector
LinkedList:底层基于链表实现,链表内存是散乱的,每一个元素存储本身
- 解决SimpleDateFormat的线程不安全问题的方法
bijian1013
javathread线程安全
在Java项目中,我们通常会自己写一个DateUtil类,处理日期和字符串的转换,如下所示:
public class DateUtil01 {
private SimpleDateFormat dateformat = new SimpleDateFormat("yyyy-MM-dd HH:mm:ss");
public void format(Date d
- http请求测试实例(采用fastjson解析)
bijian1013
http测试
在实际开发中,我们经常会去做http请求的开发,下面则是如何请求的单元测试小实例,仅供参考。
import java.util.HashMap;
import java.util.Map;
import org.apache.commons.httpclient.HttpClient;
import
- 【RPC框架Hessian三】Hessian 异常处理
bit1129
hessian
RPC异常处理概述
RPC异常处理指是,当客户端调用远端的服务,如果服务执行过程中发生异常,这个异常能否序列到客户端?
如果服务在执行过程中可能发生异常,那么在服务接口的声明中,就该声明该接口可能抛出的异常。
在Hessian中,服务器端发生异常,可以将异常信息从服务器端序列化到客户端,因为Exception本身是实现了Serializable的
- 【日志分析】日志分析工具
bit1129
日志分析
1. 网站日志实时分析工具 GoAccess
http://www.vpsee.com/2014/02/a-real-time-web-log-analyzer-goaccess/
2. 通过日志监控并收集 Java 应用程序性能数据(Perf4J)
http://www.ibm.com/developerworks/cn/java/j-lo-logforperf/
3.log.io
和
- nginx优化加强战斗力及遇到的坑解决
ronin47
nginx 优化
先说遇到个坑,第一个是负载问题,这个问题与架构有关,由于我设计架构多了两层,结果导致会话负载只转向一个。解决这样的问题思路有两个:一是改变负载策略,二是更改架构设计。
由于采用动静分离部署,而nginx又设计了静态,结果客户端去读nginx静态,访问量上来,页面加载很慢。解决:二者留其一。最好是保留apache服务器。
来以下优化:
- java-50-输入两棵二叉树A和B,判断树B是不是A的子结构
bylijinnan
java
思路来自:
http://zhedahht.blog.163.com/blog/static/25411174201011445550396/
import ljn.help.*;
public class HasSubtree {
/**Q50.
* 输入两棵二叉树A和B,判断树B是不是A的子结构。
例如,下图中的两棵树A和B,由于A中有一部分子树的结构和B是一
- mongoDB 备份与恢复
开窍的石头
mongDB备份与恢复
Mongodb导出与导入
1: 导入/导出可以操作的是本地的mongodb服务器,也可以是远程的.
所以,都有如下通用选项:
-h host 主机
--port port 端口
-u username 用户名
-p passwd 密码
2: mongoexport 导出json格式的文件
- [网络与通讯]椭圆轨道计算的一些问题
comsci
网络
如果按照中国古代农历的历法,现在应该是某个季节的开始,但是由于农历历法是3000年前的天文观测数据,如果按照现在的天文学记录来进行修正的话,这个季节已经过去一段时间了。。。。。
也就是说,还要再等3000年。才有机会了,太阳系的行星的椭圆轨道受到外来天体的干扰,轨道次序发生了变
- 软件专利如何申请
cuiyadll
软件专利申请
软件技术可以申请软件著作权以保护软件源代码,也可以申请发明专利以保护软件流程中的步骤执行方式。专利保护的是软件解决问题的思想,而软件著作权保护的是软件代码(即软件思想的表达形式)。例如,离线传送文件,那发明专利保护是如何实现离线传送文件。基于相同的软件思想,但实现离线传送的程序代码有千千万万种,每种代码都可以享有各自的软件著作权。申请一个软件发明专利的代理费大概需要5000-8000申请发明专利可
- Android学习笔记
darrenzhu
android
1.启动一个AVD
2.命令行运行adb shell可连接到AVD,这也就是命令行客户端
3.如何启动一个程序
am start -n package name/.activityName
am start -n com.example.helloworld/.MainActivity
启动Android设置工具的命令如下所示:
# am start -
- apache虚拟机配置,本地多域名访问本地网站
dcj3sjt126com
apache
现在假定你有两个目录,一个存在于 /htdocs/a,另一个存在于 /htdocs/b 。
现在你想要在本地测试的时候访问 www.freeman.com 对应的目录是 /xampp/htdocs/freeman ,访问 www.duchengjiu.com 对应的目录是 /htdocs/duchengjiu。
1、首先修改C盘WINDOWS\system32\drivers\etc目录下的
- yii2 restful web服务[速率限制]
dcj3sjt126com
PHPyii2
速率限制
为防止滥用,你应该考虑增加速率限制到您的API。 例如,您可以限制每个用户的API的使用是在10分钟内最多100次的API调用。 如果一个用户同一个时间段内太多的请求被接收, 将返回响应状态代码 429 (这意味着过多的请求)。
要启用速率限制, [[yii\web\User::identityClass|user identity class]] 应该实现 [[yii\filter
- Hadoop2.5.2安装——单机模式
eksliang
hadoophadoop单机部署
转载请出自出处:http://eksliang.iteye.com/blog/2185414 一、概述
Hadoop有三种模式 单机模式、伪分布模式和完全分布模式,这里先简单介绍单机模式 ,默认情况下,Hadoop被配置成一个非分布式模式,独立运行JAVA进程,适合开始做调试工作。
二、下载地址
Hadoop 网址http:
- LoadMoreListView+SwipeRefreshLayout(分页下拉)基本结构
gundumw100
android
一切为了快速迭代
import java.util.ArrayList;
import org.json.JSONObject;
import android.animation.ObjectAnimator;
import android.os.Bundle;
import android.support.v4.widget.SwipeRefreshLayo
- 三道简单的前端HTML/CSS题目
ini
htmlWeb前端css题目
使用CSS为多个网页进行相同风格的布局和外观设置时,为了方便对这些网页进行修改,最好使用( )。http://hovertree.com/shortanswer/bjae/7bd72acca3206862.htm
在HTML中加入<table style=”color:red; font-size:10pt”>,此为( )。http://hovertree.com/s
- overrided方法编译错误
kane_xie
override
问题描述:
在实现类中的某一或某几个Override方法发生编译错误如下:
Name clash: The method put(String) of type XXXServiceImpl has the same erasure as put(String) of type XXXService but does not override it
当去掉@Over
- Java中使用代理IP获取网址内容(防IP被封,做数据爬虫)
mcj8089
免费代理IP代理IP数据爬虫JAVA设置代理IP爬虫封IP
推荐两个代理IP网站:
1. 全网代理IP:http://proxy.goubanjia.com/
2. 敲代码免费IP:http://ip.qiaodm.com/
Java语言有两种方式使用代理IP访问网址并获取内容,
方式一,设置System系统属性
// 设置代理IP
System.getProper
- Nodejs Express 报错之 listen EADDRINUSE
qiaolevip
每天进步一点点学习永无止境nodejs纵观千象
当你启动 nodejs服务报错:
>node app
Express server listening on port 80
events.js:85
throw er; // Unhandled 'error' event
^
Error: listen EADDRINUSE
at exports._errnoException (
- C++中三种new的用法
_荆棘鸟_
C++new
转载自:http://news.ccidnet.com/art/32855/20100713/2114025_1.html
作者: mt
其一是new operator,也叫new表达式;其二是operator new,也叫new操作符。这两个英文名称起的也太绝了,很容易搞混,那就记中文名称吧。new表达式比较常见,也最常用,例如:
string* ps = new string("
- Ruby深入研究笔记1
wudixiaotie
Ruby
module是可以定义private方法的
module MTest
def aaa
puts "aaa"
private_method
end
private
def private_method
puts "this is private_method"
end
end