前情回顾
Metagenome 组装的具体处理步骤如下:
1)经过预处理后得到 Clean Data,使用 SOAP denovo 组装软件进行组装;
2)对于单个样品,首先选取一个 K-mer(默认选取55)进行组装,得到该样品的组装结果;
3)将组装得到的 Scaffolds 从 N 连接处打断,得到不含 N 的序列片段,称为 Scaftigs (i.e., continuous sequences within scaffolds);
4)将各样品质控后的 CleanData 采用 SoapAligner 软件比对至各样品组装后的 Scaftigs 上,获取未被利用上的 PE reads;
5)将各样品未被利用上的 reads 放在一起,进行混合组装,组装时,考虑到计算消耗和时间消耗,只选取一个 kmer 进行组装(默认-K 55),其他组装参数与单样品组装参数相同;
6)将混合组装的 Scaffolds 从 N 连接处打断,得到不含 N 的 Scaftigs 序列;
7)对于单样品和混合组装生成的 Scaftigs,过滤掉 500bp 以下的片段,并进行统计分析和后续基因预测;
混合组装的结果和一般样本组装的结果一样:
里面有:
NOVO_MIX/
|-- assembly.stat 【k-mer组装结果统计】
|-- NOVO_MIX.assembly.list【路径信息】
|-- NOVO_MIX.len.png 【长度信息】
|-- NOVO_MIX.len.svg
|-- NOVO_MIX.scafSeq.500.ss.txt【cutlen为500时scaffold基本信息统计】
|-- NOVO_MIX.scafSeq.fa
|-- NOVO_MIX.scaftigs.500.ss.txt
|-- NOVO_MIX.scaftigs.fa
|-- NOVO_MIX.scaftigs.len
|-- NOVO_MIX.soapdenovo.cfg【组装参数】
`-- ReadsMapping 【组装前readsmapping的的结果目录】
作为一个独立的样本参与了基因预测
NOVO_MIX/
|-- cds.length.screen.log
|-- length.screen.log
|-- NOVO_MIX.CDS.fa
|-- NOVO_MIX.CDS.fa.integrity.stat.xls
|-- NOVO_MIX.CDS.fa.len.png
|-- NOVO_MIX.CDS.fa.len.svg
|-- NOVO_MIX.CDS.fa.len.xls
|-- NOVO_MIX.CDS.fa.stat.xls
|-- NOVO_MIX.CDS.ori.fa【该样本预测基因原始CDS碱基序列】
|-- NOVO_MIX.mgm
|-- NOVO_MIX.protein.fa
|-- NOVO_MIX.protein.ori.fa
|-- NOVO_MIX.rename.mgm
|-- NOVO_MIX.rename.mgm.gff
`-- NOVO_MIX.scaf.500.fa 【按照长度500过滤后的Scaftigs
,用于进行基因预测】
CDS是编码序列(Coding sequence)的缩写。
DNA转录成mRNA,mRNA经剪接等加工后翻译出蛋白质,所谓CDS就是与蛋白质序列一 一对应的DNA序列,且该序列中间不含其它非该蛋白质对应的序列,不考虑mRNA加工等过程中的序列变化,总之,就是与蛋白质的密码子完全对应。
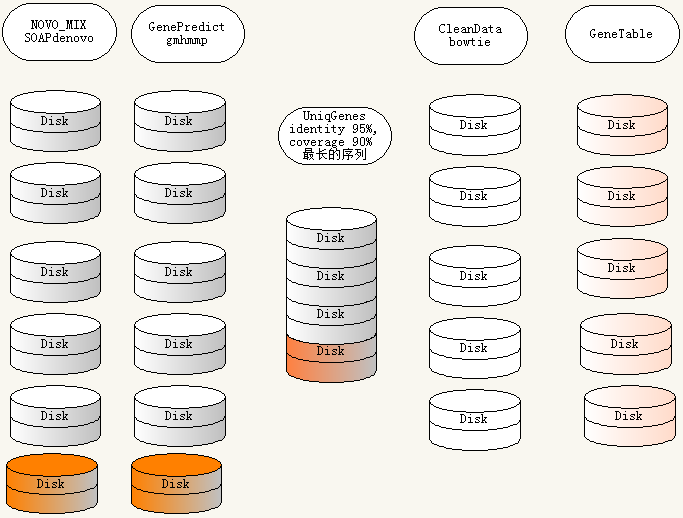
对各样品及混合组装的 ORF 预测结果,采用 CD-HIT[21,22] 软件进行去冗余,以获得非冗余的初始 gene catalogue(此处,操作上,将非冗余的连续基因编码的核酸序列称之为 genes[17])
默认以 identity 95%, coverage 90% 进行聚类,并选取最长的序列为代表性序列;采用参数[16,18]:-c 0.95, -G 0, -aS 0.9, -g 1, -d 0
03.GenePredict/UniqGenes/
|-- Unigenes.CDS.cdhit.fa 【去冗余后基因的碱基序列】
|-- Unigenes.CDS.cdhit.fa.integrity.stat.xls
|-- Unigenes.CDS.cdhit.fa.len.png
|-- Unigenes.CDS.cdhit.fa.len.svg
|-- Unigenes.CDS.cdhit.fa.len.xls【去冗余后碱基序列长度统计表】
|-- Unigenes.CDS.cdhit.fa.stat.xls
|-- Unigenes.protein.cdhit.fa【去冗余后基因的蛋白序列】
|-- Unigenes.protein.cdhit.fa.clstr
|-- Unigenes.protein.cdhit.fa.len.xls
|-- Unigenes.protein.fa
`-- Unigenes.protein.table.txt 【去冗余后代表基因的统计信息】
GeneTable/
|-- F11.1
|-- F11.2
|-- F11.3
|-- KB1
|-- KB2
|-- KB3
|-- soap.coverage.list
|-- soap.coverage.single.list
|-- soap.readsNum.list
|-- soap.unmapping.list
`-- Total
head KB2.readsNum.xls
Gene_ID Gene_Reads
F11.2_26625 8400
KB1_52315 7771
F11.1_613 7580
F11.1_29318 7472
F11.1_8330 7119
F11.1_3050 6250
Unigenes mapping到各个样本
perl 01.MGsoap/reads_mapping.pl --vf 16G --mf all.mf --mapmethod bowtie --mapopts ' --end-to-end --sensitive --no-unal -I 200 -X 400 --threads 8 ' --cutoff 2 --fq_pattern '.*\_(\d+)(\.nohost)*\.fq[12].gz' --index 03.GenePredict/UniqGenes/Unigenes.CDS.cdhit.fa --genel 03.GenePredict/UniqGenes/Unigenes.CDS.cdhit.fa.len.xls --aa 03.GenePredict/UniqGenes/Unigenes.protein.cdhit.fa --data_list 01.DataClean/Dataclean.total.list --prefix Unigenes --shdir ./ --outdir ./
混合组装前,寻找unmapping
perl 01.MGsoap/reads_mapping.pl --mapmethod bowtie --mapopts ' --end-to-end --sensitive -I 200 -X 400 --threads 8 ' --mf all.mf --data_list Dataclean.total.list --index_f total.scaftigs.list --outdir --shdir ./ --unmap
4)过滤掉在各个样品中支持 reads 数目<=2[15,23] 的基因,获得最终用于后续分析的 gene catalogue(Unigenes);
5)从比对上的 reads 数目及基因长度出发,计算得到各基因在各样品中的丰度信息
6)基于 gene catalogue 中各基因在各样品中的丰度信息,进行基本信息统计,core-pan 基因分析,样品间相关性分析,及基因数目韦恩图分析。