10xGenomics单细胞转录seurat包提取表达量画小提琴图
- 需求
- 提取表达
- 画小提琴图
- 对噪音处理
需求
其实seurat包已经有画小提琴图的函数,VlnPlot,参数也较多,基本上可以满足需求,但是如果需要对其x、y轴的文字以及大小调整,可以直接添加其他的ggplot的命名,比如命令如下:
p<-VlnPlot(object = newname.immune.combined, features.plot = as.character(gene), return.plotlist=TRUE,point.size.use = -1,cols.use=color,size.x.use = -1,do.sort = F)
p<-p+labs(x='Clusters', y= 'Gene Expresion')
p<-p+theme(panel.grid=element_blank(), legend.background = element_rect(colour = NA),
legend.title = element_blank(),legend.text = element_text(face="plain", color="black",size = 20),
axis.text.x = element_text(color="black",size=15),
axis.text.y = element_text(color="black",size=15),
axis.title.x = element_text(face="plain", color="black",size=20),
axis.title.y = element_text(face="plain", color="black",size=20))
- [x] 参数: size.x.use = -1
- [x] 参数: immune.combined为seurat对象
- [x] 参数: features.plot,为基因名称
- [x] 参数: cols.use为调整小提琴的颜色
- [x] 参数: labs添加坐标轴或者title名字,与普通的ggplot中的一样
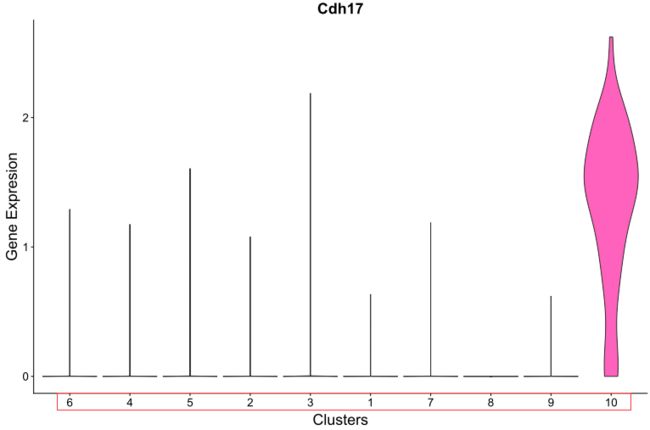
正常情况下,这样基本上满足需求,但是之前有个特殊的项目,将项目中的聚类名字修改了,改变顺序,然后画出来的图顺序不对(如下图),因此需要调整顺序。
但是VlnPlot函数中没有找到相关参数,幸亏seurat包功能比较全,那我就提取表达量数据,自己作图就ok了。
提取表达量
seurat提取表达量函数为:FetchData,提取命令也很简单:
exprs <- data.frame(FetchData(object = immune.combined, vars.all = gene))
- [x] 参数: object,为seurat分析对象
- [x] 参数: vars.all,提取数据的名称,可以是向量,比如:c("tSNE_1","tSNE_2", 基因名称)
-
[x] 参数: cells.use,提取数据的细胞barcode,这里不用
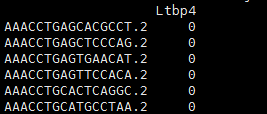
提取表达结果如下图:
这里提取的数据只有barcode和表达量,没有聚类信息,因此需要增加聚类信息:
exprs$Barcod<-rownames(exprs)
ident<-data.frame()
#barcode与聚类信息提取
ident<-data.frame(Barcod=names(newname.immune.combined@ident),orig.ident=newname.immune.combined@ident)
#通过merge函数,将表达量与聚类号对应起来
c<-merge(exprs,ident,by='Barcod')
#对其进行排序
c$orig.ident<-factor(c$orig.ident,levels=c(sort(unique(immune.combined@ident))))
最终得到如下表达量矩阵:
得到上述表达矩阵,开始画小提琴图:
ggplot(data = c,mapping = aes(x = factor(x = orig.ident),y = c[,2])) +geom_violin(scale = "width",adjust =1,trim = TRUE,mapping = aes(fill = factor(x = orig.ident)))+labs(x='Clusters', y= 'Gene Expresion',title=costmer_gene[m])+scale_colour_manual(values=mycolo)+theme(legend.position = 'none')+theme(panel.grid=element_blank(), legend.background = element_rect(colour = NA),
legend.title = element_blank(),legend.text = element_text(face="plain", color="black",size = 20),
axis.text.x = element_text(color="black",size=20),
axis.text.y = element_text(color="black",size=20),
axis.title.x = element_text(face="plain", color="black",size=20),
axis.title.y = element_text(face="plain", color="black",size=20))
ggsave('test.pdf')
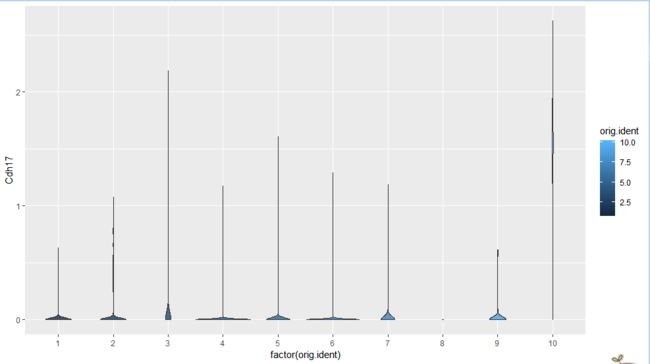
错误的小提琴图
开始按照普通的画小提琴进行小提琴绘制:
ggplot(data,aes(x=factor(orig.ident),y=Cdh17,fill=orig.ident))+geom_violin(alpha=0.8,width=1)
ggsave('test.pdf')
得到如下图小提琴图,可以看到数据分别有明显从错误。
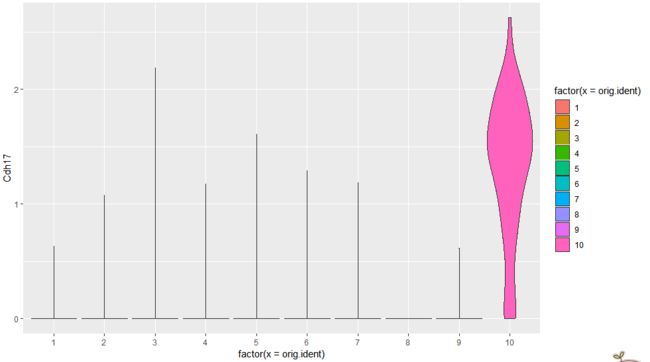
正确的小提琴图
通过修改小提琴图画法,找到正确的小提琴图命名:
ggplot(data = data,mapping = aes(x = factor(x = orig.ident),y = Cdh17)) +geom_violin(scale = "width",adjust =1,trim = TRUE,mapping = aes(fill = factor(x = orig.ident)))
ggsave('test.pdf')
得到如下小提琴图:
从图中可以看出,数据分布基本正常,聚类10的表达量明显高于其他聚类,但是与我们直接用VlnPlot函数画图出来的图还是不一样,除了聚类10以外,其他表达量也较高。
最终正确小提琴图
通过查看seurat官网在GitHub上面的SingleVlnPlot源代码,然后发现其他官方对其有一个噪音处理过程,这是与VlnPlot画出来不一样的原因,然后我们也加上噪音处理,可以得到与VlnPlot一样的图。(VlnPlot函数是调用SingleVlnPlot画图)
noise <- rnorm(n = length(x = data[,c('Cdh17')])) / 100000
data[,c('Cdh17')] <- data[, c('Cdh17')] + noise
ggplot(data = data,mapping = aes(x = factor(x = orig.ident),y = Cdh17)) +geom_violin(scale = "width",adjust =1,trim = TRUE,mapping = aes(fill = factor(x = orig.ident)))
ggsave('test.pdf')
得到与VlnPlot的几乎一致的图:
,至于颜色,legend都可以通过ggplot2画图图层修改。
到此,画小提琴图终于完成了。
2019年3月27日