鸢尾花分类matlab代码
文章目录
- 鸢尾花分类matlab代码
- 一、Iris数据集介绍
- 二、代码分析
- 2.1 标签数据化
- 2.2 可视化分析
- 2.2.1 相关系数热度图
- 2.2.2 箱型图
- 2.2.3 散点图
- 2.3 数据集的准备
- 2.3.1 训练集和测试集的划分
- 2.3.2 数据预处理
- 2.4 SVM网络的训练与预测
- 2.5 预测结果分析
- 2.5.1 svmpredict函数结果
- 2.5.2 预测结果可视化
- 三、模型优化
- 3.1 libsvm训练参数简介
- 3.2 利用PSO算法对参数进行优化
鸢尾花分类matlab代码
本文是基于matlab平台的libsvm工具箱进行的,是羊同学的练手做,代码编写不太仔细,欢迎大家斧正。
一、Iris数据集介绍
Iris 鸢尾花数据集是一个经典数据集,在统计学习和机器学习领域都经常被用作示例。数据集内包含 3 类共 150 条记录,每类各 50 个数据,每条记录都有 4 项特征:花萼长度、花萼宽度、花瓣长度、花瓣宽度,可以通过这4个特征预测鸢尾花卉属于(iris-setosa, iris-versicolour, iris-virginica)中的哪一品种。
由于本数据集十分的典型,羊同学便采用了本数据集。
我们通过以下两条命令简单的观察一下该数据集的形式:
load Iris
head Iris
得到结果如下:
ans =
8×6 table
Id SepalLengthCm SepalWidthCm PetalLengthCm PetalWidthCm Species
__ _____________ ____________ _____________ ____________ ___________
1 5.1 3.5 1.4 0.2 Iris-setosa
2 4.9 3 1.4 0.2 Iris-setosa
3 4.7 3.2 1.3 0.2 Iris-setosa
4 4.6 3.1 1.5 0.2 Iris-setosa
5 5 3.6 1.4 0.2 Iris-setosa
6 5.4 3.9 1.7 0.4 Iris-setosa
7 4.6 3.4 1.4 0.3 Iris-setosa
8 5 3.4 1.5 0.2 Iris-setosa
二、代码分析
2.1 标签数据化
为了方便,我们将标签数据化处理。我们可通过unique()函数查看类别数。
unique(Iris.Species)
可以得到结果:
ans =
3×1 categorical 数组
Iris-setosa
Iris-versicolor
Iris-virginica
所以羊同学随手把三种类别转换为了1、2、3。
%% 将类别数据化
num = length(Iris.Species);
label = [];
for i = 1:num
switch Iris.Species(i)
case 'Iris-setosa'
label(i) = 1;
case 'Iris-versicolor'
label(i) = 2;
case 'Iris-virginica '
label(i) = 3;
end
end
label = label';
data = [Iris.SepalLengthCm,Iris.SepalWidthCm,Iris.PetalLengthCm,Iris.PetalWidthCm];
2.2 可视化分析
羊同学决定对数据进行简单的可视化分析,对数据的情况有个整体的把握,主要从以下几个方面进行:
- 相关系数热度图
- 箱型图
- 散点图
2.2.1 相关系数热度图
RHO = corr(data);
name = Iris.Properties.VariableNames(2:5);
heatmap(name,name,RHO);
colormap hot
其结果如图:
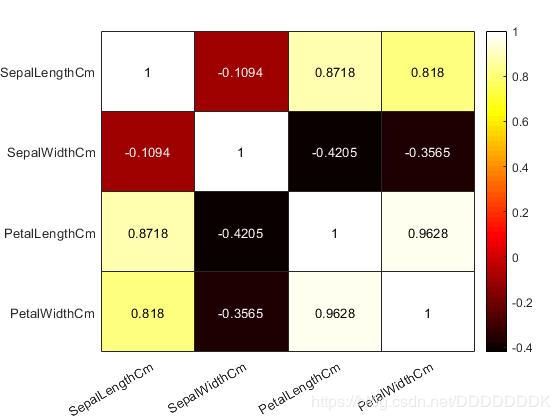
由此可发现PetalLengthCm和PetalWidthCm相关性较强,我们可以删除一组,也可以用PCA提取主成分消除独立性。不过由于羊同学很懒,就直接不做了,有兴趣的同学可以试试看,效果会不会变好。
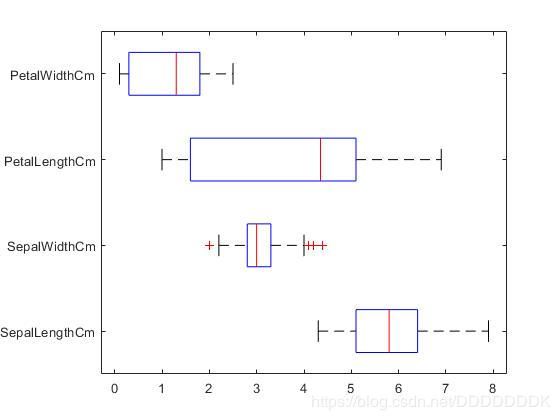
2.2.2 箱型图
boxplot(data,'orientation','horizontal','labels',name);
2.2.3 散点图
one2six = 1:4;
comb = combntns(one2six,2);
index_1 = find(label==1);
index_2 = find(label==2);
index_3 = find(label==3);
figure
hold on
for i = 1:6
subplot(2,3,i)
scatter(data(index_1,comb(i,1)),index_1,data(comb(i,2)),'fill','r');
hold on
scatter(data(index_2,comb(i,1)),index_2,data(comb(i,2)),'fill','g');
hold on
scatter(data(index_3,comb(i,1)),index_3,data(comb(i,2)),'fill','b');
title([name{comb(i,1)},' and ',name{comb(i,2)}]);
legend('Iris-setosa','Iris-versicolor','Iris-virginica','location','best');
end
hold off
2.3 数据集的准备
2.3.1 训练集和测试集的划分
羊同学随手划分了一下,由于样本数不多感兴趣的同学也可以采用k-fold crossValidation方法。
train_data = [data(1:40,:);data(51:90,:);data(101:140,:)];
train_label = [label(1:40,:);label(51:90,:);label(101:140,:)];
test_data = [data(1:40,:);data(51:90,:);data(101:140,:)];
test_label = [label(1:40,:);label(51:90,:);label(101:140,:)];
2.3.2 数据预处理
使用mapminmax函数时,注意它的归一化方法。在归一化前记得转置。
[mtrain,ntrain] = size(train_data);
[mtest,ntest] = size(test_data);
dataset = [train_data;test_data]; % mapminmax为MATLAB自带的归一化函数
[dataset_scale,ps] = mapminmax(dataset',0,1); %归一化要先转至
dataset_scale = dataset_scale';
train_data = dataset_scale(1:mtrain,:);
test_data = dataset_scale( (mtrain+1):(mtrain+mtest),: );
2.4 SVM网络的训练与预测
羊同学先不调整任何参数,直接使用默认参数进行训练,看看效果:
%% SVM网络训练
model = svmtrain(train_label, train_data,'-c 2 -g 1');
%% SVM网络预测
[predict_label, accuracy,desc_value] = svmpredict(test_label, test_data, model); % desc_value !!
注意:libsvm工具箱由于版本不同会有一定的不同,新版本的预测函数需要添加dec_value,不然会运算报错。
2.5 预测结果分析
2.5.1 svmpredict函数结果
optimization finished, #iter = 30
nu = 0.410476
obj = -48.386381, rho = 0.160529
nSV = 34, nBSV = 31
Total nSV = 43
Accuracy = 96.6667% (116/120) (classification)
由此我们可以看到正确率达到了96.6667%,有种勉勉强强的感觉呢。
2.5.2 预测结果可视化
figure;
hold on;
plot(test_label,'o');
plot(predict_label,'r*');
xlabel('测试集样本','FontSize',12);
ylabel('类别标签','FontSize',12);
legend('实际测试集分类','预测测试集分类');
title('测试集的实际分类和预测分类图','FontSize',12);
grid on;
结果如图:

通过图可以看出只有一个测试样本是被错分的。就这样简单的libsvm模型就完成了。当然还可以对其进行许多方面的优化,最重要的一点就是对于libsvm参数模型的优化。
三、模型优化
3.1 libsvm训练参数简介
Options:可用的选项即表示的涵义如下
- -s svm类型:SVM设置类型(默认0)
- 0 – C-SVC
- 1 --v-SVC
- 2 – 一类SVM
- 3 – e -SVR
- 4 – v-SVR
- -t 核函数类型:核函数设置类型(默认2)
- 0 – 线性:u’v
- 1 – 多项式:(r*u’v + coef0)^degree
- 2 – RBF函数:exp(-r|u-v|^2)
- 3 –sigmoid:tanh(r*u’v + coef0)
- -d degree:核函数中的degree设置(针对多项式核函数)(默认3)
- -g r(gama):核函数中的gamma函数设置(针对多项式/rbf/sigmoid核函数)(默认1/ k)
- -r coef0:核函数中的coef0设置(针对多项式/sigmoid核函数)((默认0)
- -c cost:设置C-SVC,e -SVR和v-SVR的参数(损失函数)(默认1)
- -n nu:设置v-SVC,一类SVM和v- SVR的参数(默认0.5)
- -p p:设置e -SVR 中损失函数p的值(默认0.1)
- -m cachesize:设置cache内存大小,以MB为单位(默认40)
- -e eps:设置允许的终止判据(默认0.001)
- -h shrinking:是否使用启发式,0或1(默认1)
- -wi weight:设置第几类的参数C为weight*C (C-SVC中的C) (默认1)
- -v n: n-fold交互检验模式,n为fold的个数,必须大于等于2
其中-g选项中的k是指输入数据中的属性数。
option -v 随机地将数据剖分为n部分并计算交互检验准确度和均方根误差。以上这些参数设置可以按照SVM的类型和核函数所支持的参数进行任意组合,如果设置的参数在函数或SVM类型中没有也不会产生影响,程序不会接受该参数;如果应有的参数设置不正确,参数将采用默认值。
training_set_file是要进行训练的数据集;model_file是训练结束后产生的模型文件,文件中包括支持向量样本数、支持向量样本以及lagrange系数等必须的参数;该参数如果不设置将采用默认的文件名,也可以设置成自己惯用的文件名。
3.2 利用PSO算法对参数进行优化
优化后的结果:
optimization finished, #iter = 40
nu = 0.518337
obj = -2741.351977, rho = 0.228981
nSV = 43, nBSV = 40
Total nSV = 52
Accuracy = 97.5% (117/120) (classification)
打印测试集分类准确率
Accuracy = 97.5% (117/120)
可视化作图:
模型得到了一定程度的优化。