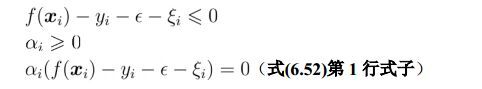西瓜书课后题——第六章(支持向量机)
课后题6.2、西瓜数据集3.0a上分别用线性核和高斯核训练一个SVM,并比较支持向量的区别。
使用LIBSVM经过训练之后发现,线性核和高斯核所得到的支持向量是一样的。LIBSVM的使用见这篇文章:LIBSVM使用
完整代码如下所示:
from libsvm.python.svmutil import *
import numpy as np
A = np.array([[0.697,0.460,1],[0.774,0.376,1],[0.634,0.264,1],[0.608,0.318,1],[0.556,0.215,1],
[0.403,0.237,1],[0.481,0.149,1],[0.437,0.211,1],[0.666,0.091,-1],[0.243,0.267,-1],
[0.245,0.057,-1],[0.343,0.099,-1],[0.639,0.161,-1],[0.657,0.198,-1],[0.360,0.370,-1],
[0.593,0.042,-1],[0.719,0.103,-1]])
y = A[:,2]
x = []
for i in range(len(y)):
dic = {}
dic[1] = A[i,0]
dic[2] = A[i,1]
x.append(dic)
lina_options = '-t 0 -c 1 -b 1' #线性核
guass_options = '-t 2 -c 4 -b 1' # 高斯核
model = svm_train(y,x,lina_options)
svm_save_model('./xiGua3.3alpha_linear',model)
model_ = svm_train(y,x,guass_options)
svm_save_model('./xiGua3.3alpha_Guass',model_)
得到的支持向量如下所示:
1 1:0.697 2:0.46
1 1:0.774 2:0.376
1 1:0.634 2:0.264
1 1:0.608 2:0.318
1 1:0.556 2:0.215
1 1:0.403 2:0.237
1 1:0.481 2:0.149
1 1:0.437 2:0.211
-1 1:0.666 2:0.091
-1 1:0.243 2:0.267
-1 1:0.343 2:0.099
-1 1:0.639 2:0.161
-1 1:0.657 2:0.198
-1 1:0.36 2:0.37
-1 1:0.593 2:0.042
-1 1:0.719 2:0.103
课后题6.3、用UCI数据集,分别用高斯核和线性核进行训练。
此处,选用了 iris数据集 中的前两类,分别标记为1和-1。用前40个样本进行训练,用后10个样本进行测试。
具体代码如下:
import numpy as np
from libsvm.python.svmutil import *
def readData(): # 读取数据
"""
Read data from txt file.
Return:
X1, y1, X2, y2, X3, y3: X is list with shape [50, 4],
y is list with shape [50,]
"""
X1 = []
y1 = []
X2 = []
y2 = []
#read data from txt file
with open("./iris.txt", "r") as f:
for line in f:
x = []
iris = line.strip().split(",")
for attr in iris[0:4]:
aa = float(attr)
x.append(aa)
if iris[4]=="Iris-setosa":
X1.append(x)
y1.append(1)
else:
X2.append(x)
y2.append(-1)
return X1, y1, X2, y2
def getX(X): # 生成LIBSVM需要的固定数据格式
A = []
X = np.array(X)
m,n = np.shape(X)
for i in range(m):
dic = {}
for j in range(n):
dic[j+1] = X[i,j]
A.append(dic)
return A
def svm_linear(x,y): # 线性核训练
lina_options = '-t 0 -c 1 -b 1' #线性核
model = svm_train(y,x,lina_options)
svm_save_model('./problem_6.3/UCI_linear',model)
def svm_guass(x,y): # 高斯核训练
guass_options = '-t 2 -c 4 -b 1' # 高斯核
model = svm_train(y,x,guass_options)
svm_save_model('./problem_6.3/UCI_guass',model)
def predic_linear(x,y): # 线性核预测
model = svm_load_model('./problem_6.3/UCI_linear')
p_label,p_acc,p_val = svm_predict(y,x,model)
return p_label,p_acc
def predic_guass(x,y): # 高斯核预测
model = svm_load_model('./problem_6.3/UCI_guass')
p_label,p_acc,p_val = svm_predict(y,x,model)
return p_label,p_acc
def main():
X1,y1,X2,y2 = readData()
x1 = getX(X1)
x2 = getX(X2)
x_train = x1[:40]
y_train = y1[:40]
x_train.extend(x2[:40])
y_train.extend(y2[:40])
x_test = x1[40:]
x_test.extend(x2[40:])
y_test = y1[40:]
y_test.extend(y2[40:])
svm_linear(x_train,y_train)
svm_guass(x_train,y_train)
p_label_lin,p_acc_lin = predic_linear(x_test,y_test)
p_label_gua,p_acc_gua = predic_guass(x_test,y_test)
print("线性预测结果")
print(p_label_lin,p_acc_lin)
print("高斯预测结果")
print(p_label_gua,p_acc_gua)
if __name__ == '__main__':
main()
最终可以发现,使用 线性核 得到的模型内容如下所示,仅有3个支持向量,第一类2个,第二类1个:
svm_type c_svc
kernel_type linear
nr_class 2
total_sv 3
rho -0.77382525930929269
label 1 -1
probA -2.4478146893743347
probB -0.35294488993985529
nr_sv 2 1
SV
0.21830366492064313 1:5.1 2:3.3 3:1.7 4:0.5
0.34115593497309121 1:4.8 2:3.4 3:1.9 4:0.2
-0.55945959989373428 1:4.9 2:2.4 3:3.3 4:1 使用高斯核得到的模型如下所示,有 7个支持向量,第一类有4个,第二类3个:
svm_type c_svc
kernel_type rbf
gamma 0.25
nr_class 2
total_sv 7
rho 0.13436257455469611
label 1 -1
probA -3.3718241673496236
probB -0.0081498851867360216
nr_sv 4 3
SV
0.67790332585451707 1:4.4 2:2.9 3:1.4 4:0.2
0.58083107807394418 1:5.7 2:4.4 3:1.5 4:0.4
0.25603598180822196 1:5.1 2:3.3 3:1.7 4:0.5
0.53119467380522756 1:4.8 2:3.4 3:1.9 4:0.2
-0.53179196812475671 1:7 2:3.2 3:4.7 4:1.4
-1.3463704603213571 1:4.9 2:2.4 3:3.3 4:1
-0.16780263109579691 1:6.7 2:3 3:5 4:1.7
但是,使用两个不同的核函数,在测试集上的精确度均为 100% 。预测的结果如下所示:
线性预测结果
[1.0, 1.0, 1.0, 1.0, 1.0, 1.0, 1.0, 1.0, 1.0, 1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0] (100.0, 0.0, 1.0)
高斯预测结果
[1.0, 1.0, 1.0, 1.0, 1.0, 1.0, 1.0, 1.0, 1.0, 1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0, -1.0] (100.0, 0.0, 1.0)课后题6.4、讨论线性判别分析和线性核支持向量机在何种条件下等价。
此题的解释参见这篇文章:https://blog.csdn.net/icefire_tyh/article/details/52135662
个人结合参考文章中博主的解释及文章末尾的评论,整理如下:
分以下几点进行一个理解:
1. LDA和线性SVM都希望能最大化异类样例间距,但LDA是异类中心间距最大化,而线性SVM考虑的是支持向量间距最大
2. LDA的目标函数考虑了同类样例的协方差,希望同类样例在投影空间尽可能靠近,而线性SVM却没有考虑这一点。
3.关于数据是否线性可分的问题,如果使用软间隔的线性SVM,线性可分这个条件是不必要的,如果是硬间隔线性SVM,那么线性可分是必要条件。但是LDA不管数据是否线性可分,都可以进行处理
4. 假如当前样本线性可分,且SVM与LDA求出的结果相互垂直。则当SVM的支持向量固定时,再加入新的非支持向量样本,并不会改变SVM中求出的w。但是新加入的样本会改变原类型数据的协方差和均值,从而导致LDA求出的结果发生改变。这个时候两者的w就不再垂直,但是数据依然是可分的。所以, 线性可分 和 LDA求出的wl与线性核支持向量机求出的ws垂直,这两个条件是不等价的。
5. 所以,该题的答案严格上来讲,应该是当线性SVM和LDA求出的w互相垂直时,两者是等价的。因为一般LDA是不带偏置项的(因为LDA的思想是投影,投影过程和偏置是没有任何关系的),所以SVM这个时候比LDA仅仅多了个偏移b。
课后题6.5、高斯核SVM和RBF之间的联系。
参考这篇博客: https://blog.csdn.net/icefire_tyh/article/details/52135662
个人整理如下几点:
1、RBF网络的径向基函数与SVM都采用高斯核,就分别得到了高斯核RBF网络与高斯核SVM。
2、神经网络是最小化累计误差,将参数 w 作为惩罚项;而SVM相反,主要是最小化参数,将误差作为惩罚项。
3、根据书上给出的公式,可以发现,在二分类问题中,如果将RBF中隐层神经元的个数设置为总样本个数,且每个隐层神经元所对应的中心设置为样本参数,并将RBF的径向基函数中的 beta 设置成高斯函数中对应的系数,则得出的RBF网络与核SVM基本等价,非支持向量在RBF中将得到很小的权重。
课后题6.6、SVM对噪声敏感的原因。
参考这篇博客:https://blog.csdn.net/Dby_freedom/article/details/82960422
个人整理如下:
1、SVM的基本形态是一个硬间隔分类器,它要求所有样本都满足硬间隔约束(即函数间隔要大于1)
2、当数据集中存在噪声点但是仍然满足线性可分的条件时,SVM为了把噪声点也划分正确,超平面就会向另外一个类的样本靠拢,这就使得划分超平面的几何间距变小,从而降低了模型的泛化性能。
3、当数据集因为存在噪声点而导致已经无法线性可分时,此时就使用了核技巧,通过将样本映射到高维特征空间使得样本线性可分,这样就会得到一个复杂模型,并由此导致过拟合(原样本空间得到的划分超平面会是弯弯曲曲的,它确实可以把所有样本都划分正确,但得到的模型只对训练集有效),泛化能力极差。
4、所以说,SVM对于噪声很敏感。因此,提出了软间隔SVM来防止由于噪声的存在而导致的过拟合问题。
课后题6.7、给出式6.52的完整KKT条件。
首先,根据KKT条件的定义,可以知道,对于拉格朗日函数中的带有拉格朗日乘子的项,每一项对应的KKT条件共有三个式子。在式6.52中,带有拉格朗日乘子的共有四项,所以完整的KKT条件应该共有12个式子。
完整的KKT条件如下所示:
课后题6.8、以密度为输入,含糖率为输出,训练一个SVR。
采用 epsilon-SVR进行回归,核函数采用高斯核,具体代码如下:
from libsvm.python.svmutil import *
import numpy as np
A = np.array([[0.697,0.460],[0.774,0.376],[0.634,0.264],[0.608,0.318],[0.556,0.215],
[0.403,0.237],[0.481,0.149],[0.437,0.211],[0.666,0.091],[0.243,0.267],
[0.245,0.057],[0.343,0.099],[0.639,0.161],[0.657,0.198],[0.360,0.370],
[0.593,0.042],[0.719,0.103]])
y = A[:,1]
x = []
for i in range(len(y)):
dic = {}
dic[1] = A[i,0]
x.append(dic)
guass_options = '-s 3 -t 2 -c 1 -b 1' # 高斯核
model = svm_train(y,x,guass_options)
svm_save_model('./problem_6.8_XG3.3alpha_guass',model)
最终训练得到的模型如下所示:
svm_type epsilon_svr
kernel_type rbf
gamma 1
nr_class 2
total_sv 9
rho -0.21442336320877067
probA 0.11761725324207717
SV
1 1:0.697
1 1:0.774
0.67138842799068921 1:0.608
-1 1:0.666
0.32861157200931085 1:0.243
-1 1:0.245
1 1:0.36
-1 1:0.593
-1 1:0.719
上面第一列其实是每一个支持向量的权值,对应于西瓜书上式6.47里面的(alpha‘ - alpha)不妨令模型中第一列为alpha,第二列为 x, 则根据书上公式6.47及式6.56,结合训练得到的模型和核函数是高斯核的情况,我们可以得到如下的含糖率和密度之间的关系 :
![]() 含糖率(x) = ∑ alphai *
含糖率(x) = ∑ alphai * exp(-|x-xi|^2) + 0.2144
(因为高斯核是exp(-gamma*|u-v|^2),而根据模型可知gamma为1,所以得到上式回归关系)