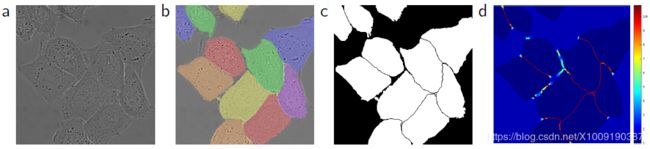
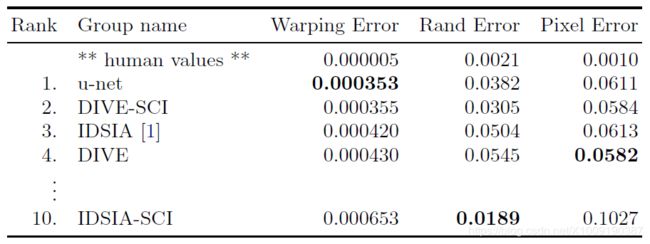
- Halcon学习之select_shape()算子参数介绍
一、算子介绍select_shape()是HALCON中用于基于形状特征筛选区域的关键算子,广泛应用于图像分割、目标检测和工业质检等领域。它允许用户根据指定的几何特征从输入区域集合中选择符合条件的区域。至于为什么单独介绍这个算子呢,因为他筛选特征的方式有太多种了,如果可以熟练的掌握这些特征,那在后面的例程学习以及实际应用中,可谓是得心应手了。二、算子参数select_shape(Regions:S
- 图像分割技术详解:从原理到实践
lanjieying
本文还有配套的精品资源,点击获取简介:图像分割是图像处理领域将图像分解为多个区域的过程,用于图像分析、特征提取等。文章介绍了图像分割的原理,并通过一个将图像划分为2*4子块的示例,展示了如何使用Python和matplotlib库中的tight_subplot函数进行图像分割和展示。文章还探讨了图像分割在不同领域的应用,以及如何在机器学习项目中作为数据预处理步骤。1.图像分割基本概念在图像处理领域
- SAM 图像分割算法计算物体表面积
loong_XL
深度学习图像CV算法SAM图像面积计算图像算法cv图像分割
参考:https://enpeicv.com/forum.php?mod=viewthread&tid=90&extra=page%3D1使用SAM算法进行图像分割,计算出分割图像有多少像素,然后根据像素数量计算实际面积局限:此方法适用于物体与参考物体一个平面内,如果物体在参考物体的前后立体位置,准确性可能不大好SAM安装及模型下载:https://github.com/facebookresea
- OpenCV图像边缘检测
慕婉0307
opencv基础opencv人工智能计算机视觉
一、边缘检测基础概念边缘检测是图像处理中最基本也是最重要的操作之一,它能识别图像中亮度或颜色急剧变化的区域,这些区域通常对应物体的边界。OpenCV提供了多种边缘检测方法,从传统的算子到基于深度学习的现代方法。1.1为什么需要边缘检测?数据降维:将图像转换为边缘表示可大幅减少数据量特征提取:边缘是图像最重要的视觉特征之一预处理步骤:为物体识别、图像分割等高级任务做准备噪声抑制:某些边缘检测方法具有
- CVPR2024 分割Segmentation相关论文37篇速览
木木阳
CVPR2024Segmentation分割论文
Paper1MFP:MakingFullUseofProbabilityMapsforInteractiveImageSegmentation摘要小结:最近的交互式分割算法中,将先前的概率图作为网络输入,以帮助当前分割轮次的预测。然而,尽管使用了先前的掩膜,概率图中包含的有用信息并没有很好地传播到当前预测中。在本文中,为了克服这一局限性,我们提出了一种新颖有效的基于点击的交互式图像分割算法MFP,
- 【EI会议征稿】东北大学主办第三届机器视觉、图像处理与影像技术国际会议(MVIPIT 2025)
诗远Yolanda
图像处理计算机视觉考研视频机器学习论文阅读
一、会议信息大会官网:www.mvipit.org官方邮箱:
[email protected]会议地点:辽宁沈阳主办单位:东北大学会议时间:2025年9月27日-9月29日二、征稿主题集中但不限于“机器视觉、图像处理与影像技术”等其他相关主题。机器视觉:视觉中的统计机器学习;立体视觉标定;几何建模与处理;人脸识别与手势识别;早期视觉和生物学启发的视觉;光流法和运动追踪;图像分割和图像分类;基于模型的视觉
- 遥感影像数据处理-大图滑窗切分为小图
GIS潮流
遥感语义分割
功能需求据所周知,遥感影像的尺寸有大有小,大的达到几万x几万像素,而图像分割算法模型在训练中尺寸适中,比如256x256,512x512,1024x1024等等,如果直接将遥感影像的原图输入模型中进行训练,大概率会提示内存和显存不足,因此针对遥感影像的模型训练,一般都需要将影像裁剪为小图。裁剪后的效果图如下:解决思路基于上面的需求,写了一套裁剪算法流程。主要考虑的是在裁剪过程中,从左往右、从上到下
- 基于OpenCV图像分割与PyTorch的增强图像分类方案
从零开始学习人工智能
opencvpytorch分类
在图像分类任务中,背景噪声和复杂场景常常会对分类准确率产生负面影响。为了应对这一挑战,本文介绍了一种结合OpenCV图像分割与PyTorch深度学习框架的增强图像分类方案。通过先对图像进行分割提取感兴趣区域(RegionofInterest,ROI),再进行分类,可以有效减少背景干扰,突出关键特征,从而提高分类准确率。该方案在多种复杂场景下表现出色,尤其适用于图像背景复杂或包含多个对象的情况。一、
- 【图像处理入门】12. 综合项目与进阶:超分辨率、医学分割与工业检测
小米玄戒Andrew
图像处理:从入门到专家图像处理人工智能深度学习算法python计算机视觉CV
摘要本周将聚焦三个高价值的综合项目,打通传统算法与深度学习的技术壁垒。通过图像超分辨率重建对比传统方法与深度学习方案,掌握医学图像分割的U-Net实现,设计工业缺陷检测的完整流水线。每个项目均包含原理解析、代码实现与性能优化,帮助读者从“技术应用”迈向“系统设计”。一、项目1:图像超分辨率重建(从模糊到清晰的跨越)1.技术背景与核心指标超分辨率(SR)是通过算法将低分辨率(LR)图像恢复为高分辨率
- 高斯混合模型(Gaussian Mixture Model, GMM)
不想秃头的程序
神经网络语音识别人工智能深度学习网络
高斯混合模型(GaussianMixtureModel,GMM)是一种概率模型,用于表示数据点由多个高斯分布(GaussianDistribution)混合生成的过程。它广泛应用于聚类分析、密度估计、图像分割、语音识别等领域,尤其适合处理非球形簇或多模态数据。以下是GMM的详细介绍:一、核心思想GMM假设数据是由多个高斯分布混合生成的,每个高斯分布代表一个簇(Cluster),并引入隐变量(Lat
- 10个基于Python的计算机视觉实战项目
云博士的AI课堂
基于Python计算机视觉python计算机视觉机器视觉人工智能
10个基于Python的计算机视觉实战项目,涵盖多个领域和应用场景,每个项目均附有GitHub地址、概述、解决的问题及应用场景:1.PCV图像处理与计算机视觉库GitHub地址:jesolem/PCV概述:提供计算机视觉基础算法的Python实现,包括图像分割、直方图均衡化、图像增强等。解决的问题:简化图像处理流程,支持快速实现算法原型。应用场景:学术研究、教学实验、图像预处理任务。2.基于朴素贝
- 用OpenCV实现图像分割:提取Logo中的特定元素并重新着色
Wave还没秃
opencv人工智能计算机视觉
用OpenCV实现图像分割:提取Logo中的特定元素并重新着色任务:将图片中三个圆形图案以及字母(共四个)单独取出,分别保存为四个新图片,图片大小与原图一致,图案尽量位于图片中心。三个圆形图案在新图片中改成不与于原来的填充色。步骤总览1.读取图片2.预处理:二值化或色彩分割找目标区域3.提取每个圆形图案(红、绿、蓝)和文字区域的掩膜4.将掩膜区域提取出来并改变颜色(对圆形)5.让内容居中对齐,保存
- CVPR 2024 图像处理方向总汇(图像去噪、图像增强、图像分割和图像恢复等)
点云SLAM
图形图像处理深度学习计算机视觉图像分割图像增强CVPR2024人工智能
1、ImageProgress(图像处理)去鬼影GeneratingContentforHDRDeghostingfromFrequencyView去阴影HomoFormer:HomogenizedTransformerforImageShadowRemoval去模糊UnsupervisedBlindImageDeblurringBasedonSelf-EnhancementLatencyCorr
- UNet改进(5):线性注意力机制(Linear Attention)-原理详解与代码实现
摸鱼许可证
人工智能计算机视觉
引言在计算机视觉领域,UNet架构因其在图像分割任务中的卓越表现而广受欢迎。近年来,注意力机制的引入进一步提升了UNet的性能。本文将深入分析一个结合了线性注意力机制的UNet实现,探讨其设计原理、代码实现以及在医学图像分割等任务中的应用潜力。UNet架构概述UNet最初由Ronneberger等人提出,主要用于生物医学图像分割。其独特的U形结构由编码器(下采样路径)和解码器(上采样路径)组成,通
- OpenCV C++ 边缘检测与图像分割
achene_ql
opencvc++计算机视觉人工智能
一、边缘检测在数字图像处理领域,边缘检测是一项至关重要的基础技术。它如同为图像赋予“骨架”,帮助计算机快速识别图像中的物体轮廓、形状与结构,广泛应用于目标识别、图像分割、图像配准等多个领域。1.1概念边缘检测的核心目标是找出图像中像素灰度发生剧烈变化的区域边界。这些边界往往对应着图像中物体的轮廓、不同物体的交界处或纹理变化明显的地方。通过提取这些边缘信息,可以有效减少图像数据量,同时保留图像中最关
- 【GitHub项目实战】Roboflow Sports 实战解析:构建多场景运动视觉系统的开源工具集与工程落地路径
观熵
GitHub开源项目实战github开源人工智能
RoboflowSports实战解析:构建多场景运动视觉系统的开源工具集与工程落地路径关键词RoboflowSports、计算机视觉、体育目标检测、关键点识别、球体追踪、OpenCV、YOLOv8、图像分割、PoseEstimation、数据集标注与训练摘要RoboflowSports是由Roboflow团队推出的面向体育分析场景的开源视觉工具集,涵盖目标检测、图像分割、关键点检测等常用任务,支持
- Matlab | matlab中的图像处理详解
北斗猿
程序语言设计(C语言C++MatlabPython等)matlab算法图像处理
MATLAB图像处理详解这里写目录标题图像处理MATLAB图像处理详解一、图像基础操作1.图像读写与显示2.图像信息获取3.图像类型转换二、图像增强技术1.对比度调整2.去噪处理3.锐化处理三、图像变换1.几何变换2.频域变换四、图像分割1.阈值分割2.边缘检测3.区域分割五、形态学操作1.基本操作2.高级形态学六、特征提取与分析1.区域属性2.纹理特征七、彩色图像处理1.色彩空间转换2.彩色分割
- 医图论文 AAAI‘25 | VOILA: 基于体素与语言交互的复杂度感知CT图像通用分割方法
小白学视觉
医学图像处理论文解读人工智能计算机视觉医学图像处理论文解读深度学习AAAI
论文信息题目:VOILA:Complexity-AwareUniversalSegmentationofCTimagesbyVoxelInteractingwithLanguageVOILA:基于体素与语言交互的复杂度感知CT图像通用分割方法作者:ZishuoWan,YuGao,WanyuanPang,DaweiDing论文创新点引入体素级对比学习:本文首次将体素级对比学习引入医学图像分割任务。通
- OpenCV C++ 图像处理教程:灰度变换与直方图分析
achene_ql
opencvc++图像处理计算机视觉人工智能
在数字图像处理领域,灰度变换与直方图分析是最基础且核心的技术,它们如同“图像的化妆师”,能够通过调整像素灰度分布显著改善图像视觉效果,为后续的目标检测、图像分割等高级任务奠定基础。无论是校正图像的亮度与对比度,还是从低质量图像中提取有效信息,掌握这些技术都是图像处理从业者的必备技能。一、点运算(PointOperation)1.概念点运算是图像处理中最基础的操作之一,指对图像中每个像素点的灰度值进
- 基于深度学习的智能图像分割系统:技术与实践
Blossom.118
机器学习与人工智能深度学习人工智能python机器学习tensorflow神经网络sklearn
前言图像分割是计算机视觉领域中的一个核心任务,其目标是将图像划分为多个有意义的区域或对象。图像分割在医学影像分析、自动驾驶、安防监控等多个领域有着广泛的应用。近年来,深度学习技术,尤其是卷积神经网络(CNN)及其变体,为图像分割带来了显著的改进。本文将详细介绍基于深度学习的智能图像分割系统的原理、实现方法以及实际应用案例。一、图像分割的基本概念1.1什么是图像分割?图像分割是一种将图像划分为多个互
- 图像的形态学操作
Suniaun原型机
OpenCV入门opencv计算机视觉c++
OpenCV中的形态学操作图像的形态学操作(MorphologicalOperations)是一种基于图像形状的处理方法,通常用于二值图像的分析和处理。形态学操作通过对图像中各个区域的结构进行改变或分析,来提取或增强图像中的形态特征(如边缘、物体、空洞等)。这些操作在许多计算机视觉任务中非常常见,例如噪声去除、边缘检测、图像分割、物体识别等。它们主要基于图像的几何形状进行分析,通过设置形态学核(通
- 医图论文 Arxiv‘24 | SEG-SAM:用于统一医学图像分割的语义引导SAM
小白学视觉
医学图像处理论文解读医学图像处理医学图像顶会Arxiv论文解读深度学习
论文信息题目:SEG-SAM:Semantic-GuidedSAMforUnifiedMedicalImageSegmentationSEG-SAM:用于统一医学图像分割的语义引导SAM作者:ShuangpingHuang,HaoLiang,QingfengWang,ChulongZhong,ZijianZhou,MiaojingShi论文创新点语义感知解码器:作者提出了一个独立的语义感知解码器(
- RV1106 图像分割 基于paddleseg
Linzhenghan
凌智视觉模块(RV1106)目标检测计算机视觉opencv人工智能
RV1106图像分割基于paddleseg图像分割1.基本知识简介1.1人像分割简介1.2人像分割常用方法2.C++API文档2.1RKNPU2Backend类2.1.1头文件2.1.2构造类函数2.1.3Initialize函数2.1.4Run函数2.1.5GetInputAttrs函数2.1.6GetOutputAttrs函数3.PP-Humanseg人像分割代码解析3.1流程图3.2核心代码
- 目标检测与图像分割:协同分析图像信息
AI天才研究院
计算AI大模型企业级应用开发实战ChatGPT计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
1.背景介绍1.1计算机视觉领域的重要任务目标检测和图像分割是计算机视觉领域中两个至关重要的任务,它们在许多应用场景中扮演着关键角色,例如自动驾驶、医疗影像分析、机器人视觉等。目标检测旨在识别图像中特定目标的位置和类别,而图像分割则将图像分割成不同的区域,每个区域代表一个特定的对象或部分。1.2从粗粒度到细粒度的图像理解目标检测提供了一种粗粒度的图像理解方式,它能够告诉我们图像中存在哪些目标以及它
- 树莓派 5 AI 套件(Hailo-8L)使用教程
kuan_li_lyg
树莓派&Jetson教程人工智能树莓派机器人开发语言嵌入式硬件python神经网络
系列文章目录目录系列文章目录前言一、人工智能模块功能二、安装三、入门3.1前提条件3.2硬件设置3.3演示3.3.1目标检测3.3.2图像分割3.3.3姿势估计四、更多资源五、产品简介前言TheRaspberryPiAIKitRaspberryPiAI套件将RaspberryPiM.2HAT+与HailoAI加速模块捆绑在一起,供RaspberryPi5使用。套件包含以下内容:包含神经处理单元(N
- 将对透视变换后的图像使用Otsu进行阈值化,来分离黑色和白色像素。这句话中的Otsu是什么意思?
黄卷青灯77
计算机视觉opencv人工智能自动化阈值OTSU
Otsu是一种自动阈值化方法,用于将图像分割为前景和背景。它通过最小化图像的类内方差或等价地最大化类间方差来选择最佳阈值。这种方法特别适用于图像的二值化处理,能够自动确定一个阈值,将图像中的像素分为黑色和白色两类。Otsu方法的原理Otsu方法的核心思想是将图像的像素分为两类(前景和背景),并通过统计分析找到一个阈值,使得这两类之间的差异最大化。具体步骤如下:计算图像的直方图:统计每个灰度值的像素
- nnUNet V2修改网络——暴力替换网络为Swin-Unet
w1ndfly
nnU-NetV2修改网络nnunet深度学习人工智能机器学习nnunetv2
更换前,要用nnUNetV2跑通所用数据集,证明nnUNetV2、数据集、运行环境等没有问题阅读nnU-NetV2的U-Net结构,初步了解要修改的网络,知己知彼,修改起来才能游刃有余。Swin-Unet是一种基于纯Transformer的U型编码器-解码器架构,专为医学图像分割任务设计。传统方法主要依赖卷积神经网络(CNN),尤其是U-Net及其变体,通过局部卷积操作和跳跃连接提取多尺度特征。然
- 使用 MMDetection 实现 Pascal VOC 数据集的目标检测项目练习(一) 开端
深蓝海拓
PascalVOC图像集的练习项目目标检测人工智能计算机视觉pytorch深度学习
一、背景知识PascalVOCPascalVOC(PatternAnalysis,StaticalModelingandComputationalLearning和VisualObjectClasses)是一个知名的计算机视觉数据集,专注于目标检测、图像分割等任务。它包含20个不同物体类别的图像注释,如人、动物、交通工具等。PascalVOC挑战赛自2005年至2012年每年举办,其中VOC200
- 跨视角差异-依赖网络用于体积医学图像分割|文献速递-生成式模型与transformer在医学影像中的应用
Title题目Cross-viewdiscrepancy-dependencynetworkforvolumetricmedicalimagesegmentation跨视角差异-依赖网络用于体积医学图像分割01文献速递介绍医学图像分割旨在从原始图像中分离出受试者的解剖结构(例如器官和肿瘤),并为每个像素分配语义类别,这在许多临床应用中起着至关重要的作用,如器官建模、疾病诊断和治疗规划(Shamsh
- 七天速成数字图像处理之五(图像分割)
ZzzZ31415926
图像处理计算机视觉算法人工智能数学建模
图像分割(ImageSegmentation)是数字图像处理中最核心、最具挑战性的任务之一,其目标是将图像划分为具有一致特征的区域,从而实现对图像中目标或结构的提取、理解与分析。下面我将从概念、分类、经典方法、实际应用四个层面为你系统性地讲解图像分割。一、什么是图像分割?定义:图像分割是指将图像划分为若干个互不重叠的区域,使得每个区域内部具有某种一致性(如灰度、纹理、颜色、边缘等),而不同区域之间
- 多线程编程之理财
周凡杨
java多线程生产者消费者理财
现实生活中,我们一边工作,一边消费,正常情况下会把多余的钱存起来,比如存到余额宝,还可以多挣点钱,现在就有这个情况:我每月可以发工资20000万元 (暂定每月的1号),每月消费5000(租房+生活费)元(暂定每月的1号),其中租金是大头占90%,交房租的方式可以选择(一月一交,两月一交、三月一交),理财:1万元存余额宝一天可以赚1元钱,
- [Zookeeper学习笔记之三]Zookeeper会话超时机制
bit1129
zookeeper
首先,会话超时是由Zookeeper服务端通知客户端会话已经超时,客户端不能自行决定会话已经超时,不过客户端可以通过调用Zookeeper.close()主动的发起会话结束请求,如下的代码输出内容
Created /zoo-739160015
CONNECTEDCONNECTED
.............CONNECTEDCONNECTED
CONNECTEDCLOSEDCLOSED
- SecureCRT快捷键
daizj
secureCRT快捷键
ctrl + a : 移动光标到行首ctrl + e :移动光标到行尾crtl + b: 光标前移1个字符crtl + f: 光标后移1个字符crtl + h : 删除光标之前的一个字符ctrl + d :删除光标之后的一个字符crtl + k :删除光标到行尾所有字符crtl + u : 删除光标至行首所有字符crtl + w: 删除光标至行首
- Java 子类与父类这间的转换
周凡杨
java 父类与子类的转换
最近同事调的一个服务报错,查看后是日期之间转换出的问题。代码里是把 java.sql.Date 类型的对象 强制转换为 java.sql.Timestamp 类型的对象。报java.lang.ClassCastException。
代码:
- 可视化swing界面编辑
朱辉辉33
eclipseswing
今天发现了一个WindowBuilder插件,功能好强大,啊哈哈,从此告别手动编辑swing界面代码,直接像VB那样编辑界面,代码会自动生成。
首先在Eclipse中点击help,选择Install New Software,然后在Work with中输入WindowBui
- web报表工具FineReport常用函数的用法总结(文本函数)
老A不折腾
finereportweb报表工具报表软件java报表
文本函数
CHAR
CHAR(number):根据指定数字返回对应的字符。CHAR函数可将计算机其他类型的数字代码转换为字符。
Number:用于指定字符的数字,介于1Number:用于指定字符的数字,介于165535之间(包括1和65535)。
示例:
CHAR(88)等于“X”。
CHAR(45)等于“-”。
CODE
CODE(text):计算文本串中第一个字
- mysql安装出错
林鹤霄
mysql安装
[root@localhost ~]# rpm -ivh MySQL-server-5.5.24-1.linux2.6.x86_64.rpm Preparing... #####################
- linux下编译libuv
aigo
libuv
下载最新版本的libuv源码,解压后执行:
./autogen.sh
这时会提醒找不到automake命令,通过一下命令执行安装(redhat系用yum,Debian系用apt-get):
# yum -y install automake
# yum -y install libtool
如果提示错误:make: *** No targe
- 中国行政区数据及三级联动菜单
alxw4616
近期做项目需要三级联动菜单,上网查了半天竟然没有发现一个能直接用的!
呵呵,都要自己填数据....我了个去这东西麻烦就麻烦的数据上.
哎,自己没办法动手写吧.
现将这些数据共享出了,以方便大家.嗯,代码也可以直接使用
文件说明
lib\area.sql -- 县及县以上行政区划分代码(截止2013年8月31日)来源:国家统计局 发布时间:2014-01-17 15:0
- 哈夫曼加密文件
百合不是茶
哈夫曼压缩哈夫曼加密二叉树
在上一篇介绍过哈夫曼编码的基础知识,下面就直接介绍使用哈夫曼编码怎么来做文件加密或者压缩与解压的软件,对于新手来是有点难度的,主要还是要理清楚步骤;
加密步骤:
1,统计文件中字节出现的次数,作为权值
2,创建节点和哈夫曼树
3,得到每个子节点01串
4,使用哈夫曼编码表示每个字节
- JDK1.5 Cyclicbarrier实例
bijian1013
javathreadjava多线程Cyclicbarrier
CyclicBarrier类
一个同步辅助类,它允许一组线程互相等待,直到到达某个公共屏障点 (common barrier point)。在涉及一组固定大小的线程的程序中,这些线程必须不时地互相等待,此时 CyclicBarrier 很有用。因为该 barrier 在释放等待线程后可以重用,所以称它为循环的 barrier。
CyclicBarrier支持一个可选的 Runnable 命令,
- 九项重要的职业规划
bijian1013
工作学习
一. 学习的步伐不停止 古人说,活到老,学到老。终身学习应该是您的座右铭。 世界在不断变化,每个人都在寻找各自的事业途径。 您只有保证了足够的技能储
- 【Java范型四】范型方法
bit1129
java
范型参数不仅仅可以用于类型的声明上,例如
package com.tom.lang.generics;
import java.util.List;
public class Generics<T> {
private T value;
public Generics(T value) {
this.value =
- 【Hadoop十三】HDFS Java API基本操作
bit1129
hadoop
package com.examples.hadoop;
import org.apache.hadoop.conf.Configuration;
import org.apache.hadoop.fs.FSDataInputStream;
import org.apache.hadoop.fs.FileStatus;
import org.apache.hadoo
- ua实现split字符串分隔
ronin47
lua split
LUA并不象其它许多"大而全"的语言那样,包括很多功能,比如网络通讯、图形界面等。但是LUA可以很容易地被扩展:由宿主语言(通常是C或 C++)提供这些功能,LUA可以使用它们,就像是本来就内置的功能一样。LUA只包括一个精简的核心和最基本的库。这使得LUA体积小、启动速度快,从 而适合嵌入在别的程序里。因此在lua中并没有其他语言那样多的系统函数。习惯了其他语言的字符串分割函
- java-从先序遍历和中序遍历重建二叉树
bylijinnan
java
public class BuildTreePreOrderInOrder {
/**
* Build Binary Tree from PreOrder and InOrder
* _______7______
/ \
__10__ ___2
/ \ /
4
- openfire开发指南《连接和登陆》
开窍的石头
openfire开发指南smack
第一步
官网下载smack.jar包
下载地址:http://www.igniterealtime.org/downloads/index.jsp#smack
第二步
把smack里边的jar导入你新建的java项目中
开始编写smack连接openfire代码
p
- [移动通讯]手机后盖应该按需要能够随时开启
comsci
移动
看到新的手机,很多由金属材质做的外壳,内存和闪存容量越来越大,CPU速度越来越快,对于这些改进,我们非常高兴,也非常欢迎
但是,对于手机的新设计,有几点我们也要注意
第一:手机的后盖应该能够被用户自行取下来,手机的电池的可更换性应该是必须保留的设计,
- 20款国外知名的php开源cms系统
cuiyadll
cms
内容管理系统,简称CMS,是一种简易的发布和管理新闻的程序。用户可以在后端管理系统中发布,编辑和删除文章,即使您不需要懂得HTML和其他脚本语言,这就是CMS的优点。
在这里我决定介绍20款目前国外市面上最流行的开源的PHP内容管理系统,以便没有PHP知识的读者也可以通过国外内容管理系统建立自己的网站。
1. Wordpress
WordPress的是一个功能强大且易于使用的内容管
- Java生成全局唯一标识符
darrenzhu
javauuiduniqueidentifierid
How to generate a globally unique identifier in Java
http://stackoverflow.com/questions/21536572/generate-unique-id-in-java-to-label-groups-of-related-entries-in-a-log
http://stackoverflow
- php安装模块检测是否已安装过, 使用的SQL语句
dcj3sjt126com
sql
SHOW [FULL] TABLES [FROM db_name] [LIKE 'pattern']
SHOW TABLES列举了给定数据库中的非TEMPORARY表。您也可以使用mysqlshow db_name命令得到此清单。
本命令也列举数据库中的其它视图。支持FULL修改符,这样SHOW FULL TABLES就可以显示第二个输出列。对于一个表,第二列的值为BASE T
- 5天学会一种 web 开发框架
dcj3sjt126com
Web框架framework
web framework层出不穷,特别是ruby/python,各有10+个,php/java也是一大堆 根据我自己的经验写了一个to do list,按照这个清单,一条一条的学习,事半功倍,很快就能掌握 一共25条,即便很磨蹭,2小时也能搞定一条,25*2=50。只需要50小时就能掌握任意一种web框架
各类web框架大同小异:现代web开发框架的6大元素,把握主线,就不会迷路
建议把本文
- Gson使用三(Map集合的处理,一对多处理)
eksliang
jsongsonGson mapGson 集合处理
转载请出自出处:http://eksliang.iteye.com/blog/2175532 一、概述
Map保存的是键值对的形式,Json的格式也是键值对的,所以正常情况下,map跟json之间的转换应当是理所当然的事情。 二、Map参考实例
package com.ickes.json;
import java.lang.refl
- cordova实现“再点击一次退出”效果
gundumw100
android
基本的写法如下:
document.addEventListener("deviceready", onDeviceReady, false);
function onDeviceReady() {
//navigator.splashscreen.hide();
document.addEventListener("b
- openldap configuration leaning note
iwindyforest
configuration
hostname // to display the computer name
hostname <changed name> // to change
go to: /etc/sysconfig/network, add/modify HOSTNAME=NEWNAME to change permenately
dont forget to change /etc/hosts
- Nullability and Objective-C
啸笑天
Objective-C
https://developer.apple.com/swift/blog/?id=25
http://www.cocoachina.com/ios/20150601/11989.html
http://blog.csdn.net/zhangao0086/article/details/44409913
http://blog.sunnyxx
- jsp中实现参数隐藏的两种方法
macroli
JavaScriptjsp
在一个JSP页面有一个链接,//确定是一个链接?点击弹出一个页面,需要传给这个页面一些参数。//正常的方法是设置弹出页面的src="***.do?p1=aaa&p2=bbb&p3=ccc"//确定目标URL是Action来处理?但是这样会在页面上看到传过来的参数,可能会不安全。要求实现src="***.do",参数通过其他方法传!//////
- Bootstrap A标签关闭modal并打开新的链接解决方案
qiaolevip
每天进步一点点学习永无止境bootstrap纵观千象
Bootstrap里面的js modal控件使用起来很方便,关闭也很简单。只需添加标签 data-dismiss="modal" 即可。
可是偏偏有时候需要a标签既要关闭modal,有要打开新的链接,尝试多种方法未果。只好使用原始js来控制。
<a href="#/group-buy" class="btn bt
- 二维数组在Java和C中的区别
流淚的芥末
javac二维数组数组
Java代码:
public class test03 {
public static void main(String[] args) {
int[][] a = {{1},{2,3},{4,5,6}};
System.out.println(a[0][1]);
}
}
运行结果:
Exception in thread "mai
- systemctl命令用法
wmlJava
linuxsystemctl
对比表,以 apache / httpd 为例 任务 旧指令 新指令 使某服务自动启动 chkconfig --level 3 httpd on systemctl enable httpd.service 使某服务不自动启动 chkconfig --level 3 httpd off systemctl disable httpd.service 检查服务状态 service h