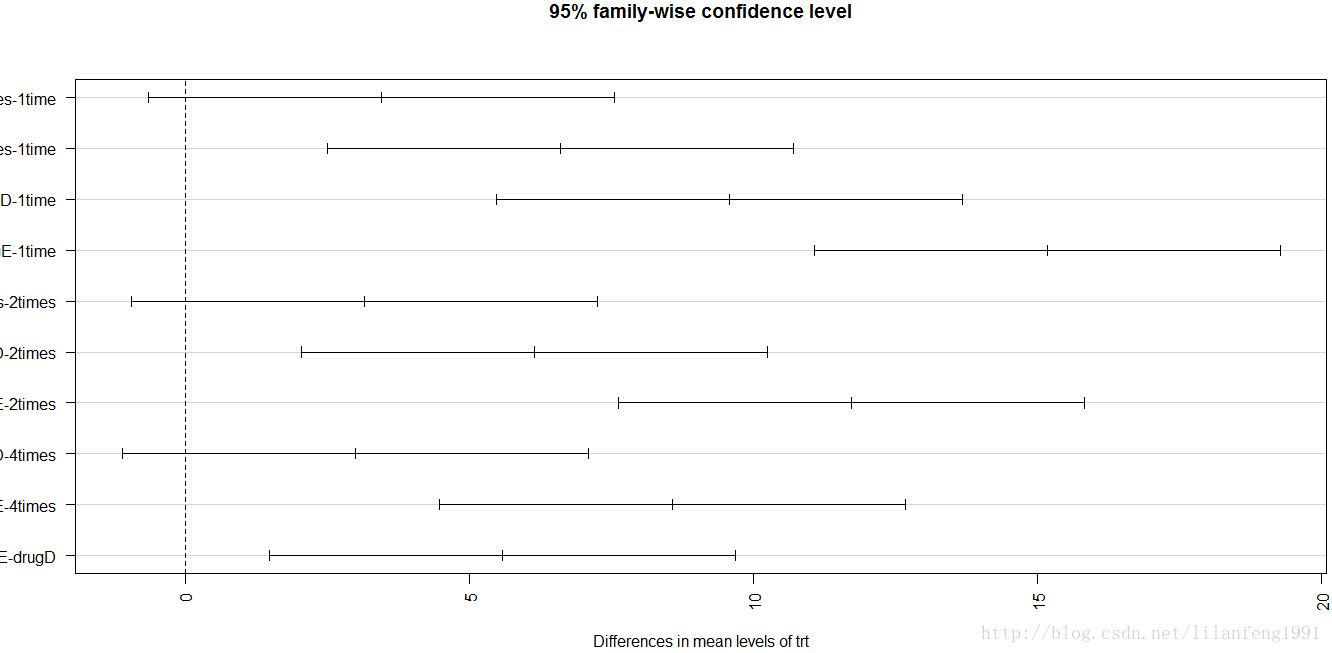R语言之方差分析篇
当包含的因子是解释变量时,通常会从预测转向 级别差异的分析,即称作方差分析(ANOVA)
组间因子
因变量
自变量
均衡设计(balanced design)
组内因子
单因素组间方差分析
单因素组内方差分析
重复测量方差分析
主效应
交叉效应
因素方差分析
混淆因素
干扰变数
协变量
协方差
1、ANOVA模型拟合
1.aov()函数
语法:aov(formula,data=dataframe)
R表达式中的特殊符号
| 符号 | 用法 |
| ~ | 分隔符号,左边为响应变量,右边为解释变量 eg:y~A+B+C |
| + | 分隔解释变量 |
| : | 表示变量的交互项 eg:y~A+B+A:B |
| * | 表示所有可能交互项 eg:y~A*B*C可展开为:y~A+B+C+A:B+A:C+B:C+A:B:C |
| ^ | 表示交互项达到次数 eg:y~(A+B+C)^2展开为:y~A+B+C+A:B+A:C+B:C |
| . | 表示包含除因变量外的所有变量 eg:若一个数据框包括变量y,A、B和C,代码y~.可展开为y~A+B+C |
常见研究设计的表达式
| 设计 | 表达式 |
| 单因素ANOVA | y~A |
| 含单个协变量的单因素ANCOVA | y~x+A |
| 双因素ANOVA | y~A*B |
| 含两个协变量的双因素ANCOVA | y~x1+x2+A*B |
| 随机化区组 | y~B+A(B是区组因子) |
| 单因素组内ANOVA | y~A+Error(Subject/A) |
| 含单个组内因子(W)和单个组间因子(B) 的重复测量ANOVA |
y~B*W+Error(Subject/W) |
非平衡设计时或存在协变量时,效应项的顺序对结果影响较大
越基础的效应应越需要放在表达式前面,首先是协变量、然后是主效应、接着是双因素的交互项,再接着是三因素的交互项
若研究不是正交的,一定要谨慎设置疚的顺序
2、单因素方差分析
(1)单因素方差分析
#单因素方差分析(感兴趣地是比较分类因子定义的两个或多个组别中的因变量均值)
install.packages("multcomp")
library(multcomp)
attach(cholesterol)
str(cholesterol)
cholesterol
table(trt)
aggregate(response,by=list(trt),FUN=mean)
aggregate(response,by=list(trt),FUN=sd)
fit<-aov(response~trt)
summary(fit)
library(gplots)
plotmeans(response~trt,xlab="Treatment",ylab="Response",main="Mean Plot\n with 95%CI")
(2)多重比较
多重比较用于解决某一组别与其他的不同
TukeyHSD()函数提供了对各组均值差异的成对检验,但与HH包存在兼容性问题((某些版本中);
TukeyHSD(fit)
par(las=2)
par(mar=c(5,4,6,2))
plot(TukeyHSD(fit))
library(multcomp)
par(mar=c(5,4,6,2))
tuk<-glht(fit,linfct=mcp(trt="Tukey"))
plot(cld(tuk,level=0.05),col="lightgrey")
(3)评估检验的假设条件
当因变量服从正态颁,各组方差相等时,可用Q-Q图来检验正态性假设
qqPlot()要求用lm()拟合,若数据落 在95%的置信区间范围内,说明满足正态性假设。
R提供的可以做方差齐性检验的函数
Bartlett检验bartlet.test()
Fligner-Killeen检验 fligner.test()
Brown-Forsythe检验
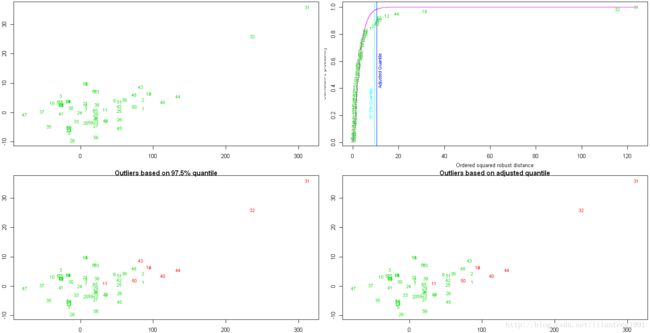
离群点检验
car包中的outlierTest()函数来检测离群点
3、单因素协方差分析
install.packages("multcomp")
library(multcomp)
head(litter,n=21)
data(litter,package="multcomp")
attach(litter)
options(digits=5)
table(litter$dose)
aggregate(weight,by=list(litter$dose),FUN=mean)
fit<-aov(weight~gesttime+litter$dose)
summary(fit)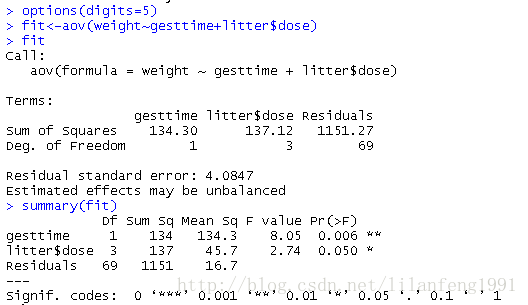
因使用了协变量,短途运输 获取调整的组均值即去除协变量疚后的组均值,可使用effects 包中的effects()函数来计算调整的均值
library(effects)
effect("dose",fit)用户定义的对照的多重比较
library(multcomp)
contrast<-rbind("no drug vs.drug"=c(3,-1,-1,-1))
summary(glht(fit,linfct=mcp(dose=contrast)))(1)评估检验的假设条件
ANCOVA与ANOVA相同,都城要正态性和同方差性假设
另ANOCVA还假定回归低低斜率相同,eg当ANCOVA模型饮食怀孕时间*剂量的交互项时,可对回归斜率的同质性进行检验。
eg
检验回归斜率的同质性
library(multcomp)
fit2<-aov(weight~gesttime*dose,data=litter)
summary(fit2)
(2)结果的可视化
install.packages("HH")
library(HH)
ancova(weight~gesttime+dose,data=litter)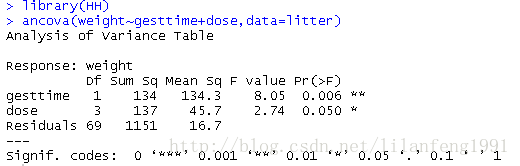
4、双因素方差分析
双因素ANOVAattach(ToothGrowth)
head(ToothGrowth)
table(supp,ToothGrowth$dose)
aggregate(len,by=list(supp,ToothGrowth$dose),FUN=mean)
aggregate(len,by=list(supp,ToothGrowth$dose),FUN=sd)
fit<-aov(len~supp*ToothGrowth$dose)
summary(fit)
detach(ToothGrowth)
可视化处理
interaction.plot(ToothGrowth$dose,supp,len,type="b",col=c("red","blue"),pch=c(16,18),main="Interaction between Dose and Supplement Type")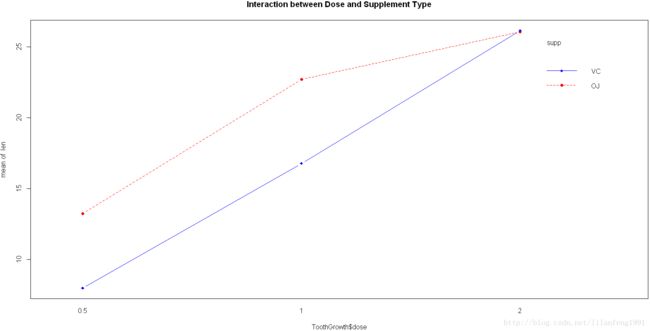
可用gplots包中的plotmans()来展示交互效应
install.packages("gplots")
library(gplots)
plotmeans(len~interaction(supp,ToothGrowth$dose,sep=" "),
connect=list(c(1,3,5),c(2,4,6)),
col=c("red","darkgreen"),
main="Interaciton Plot with 95% CIS",
xlab="Treatment and Dose Combination")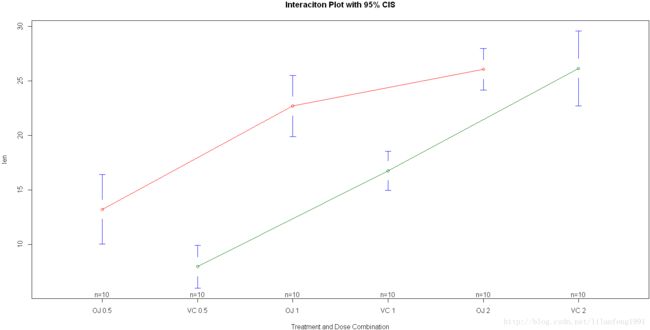
用HH包中的interaction2wt()函数来可视化结果
library(HH)
interaction2wt(len~supp*ToothGrowth$dose)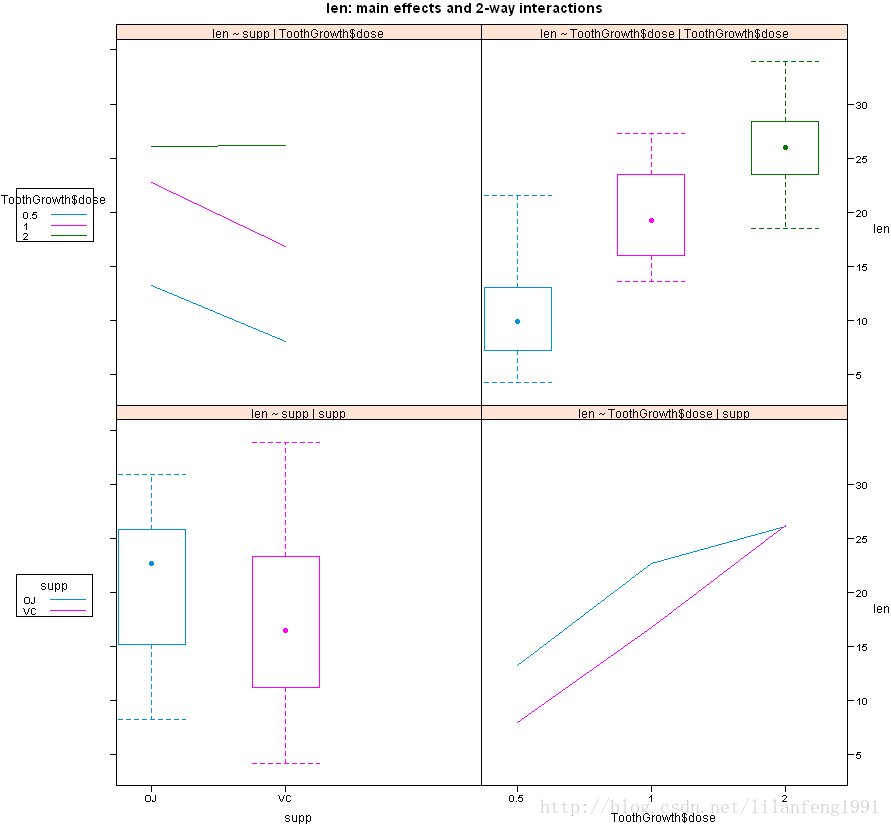
5、重复测量方差分析
所谓重复测量方差分析,即受试者被测量不止一次。
w1b1<-subset(CO2,Treatment=="chilled")
w1b1
fit<-aov(uptake~conc*Type+Error(Plant/(conc)),w1b1)
summary(fit)
par(las=2)
par(mar=c(10,4,4,2))
with(w1b1,interaction.plot(conc,Type,uptake,type="b",col=c("red","blue"),pch=c(16,18),
main="Interaction Plot for Plant Type and Concentration"))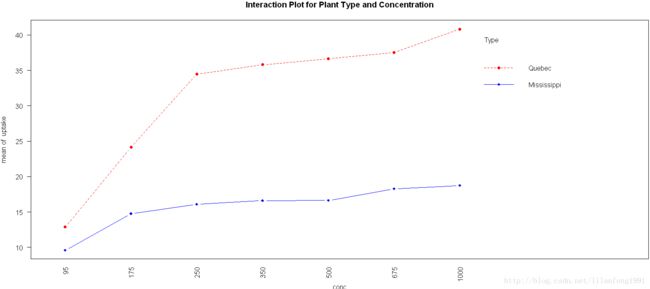
boxplot(uptake~Type*conc,data=w1b1,col=c("gold","green"),
main="Chilled Quebec and Mississippi Plants",
ylab="Carbon dioxide uptake rate umol/m^2 sec")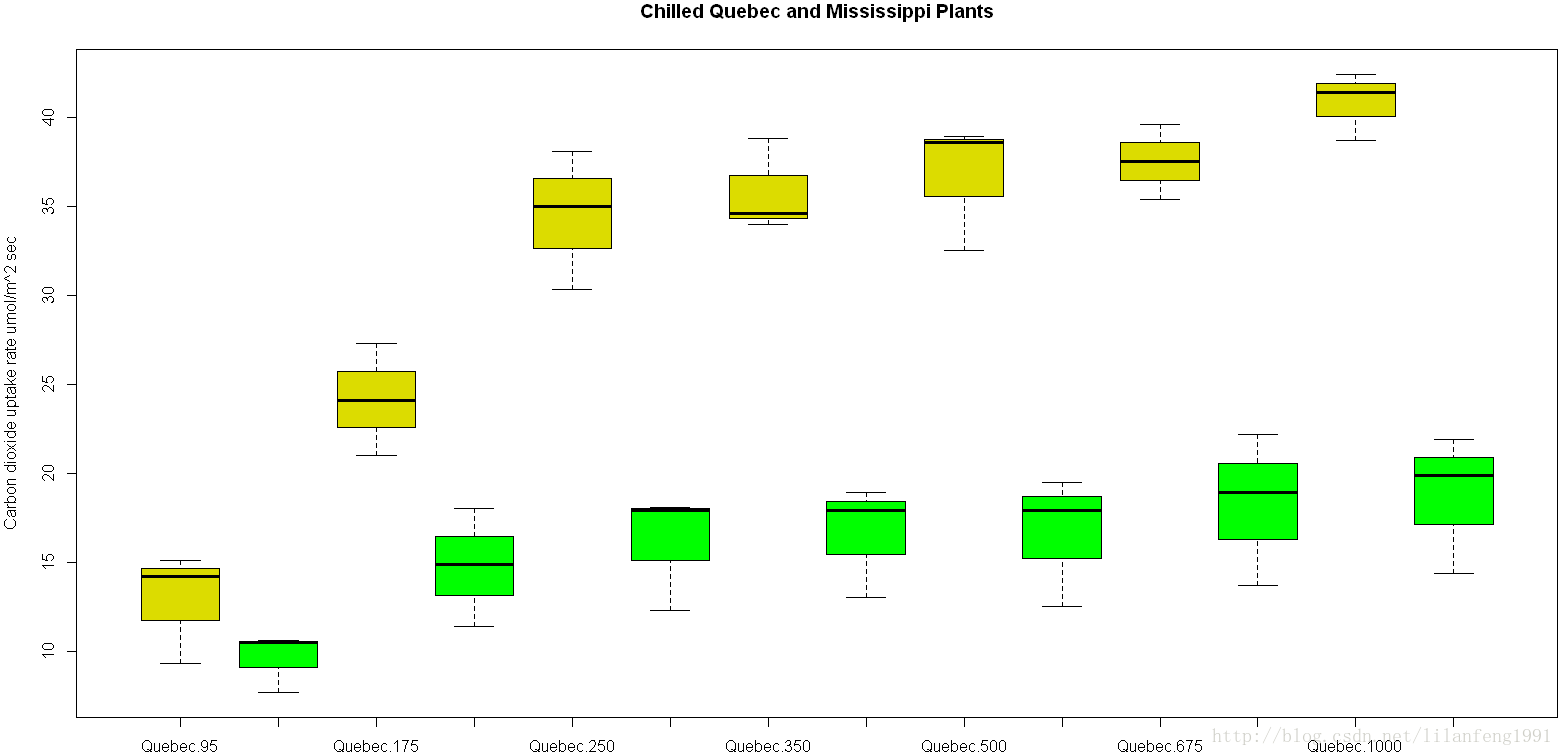
数据集
宽格式(wide format):列是变量,行是观测值,且一行一个受试对象
处理重复测量设计时,需要有长格式(long format)数据才能拟合模型;在长格式中,因变量每次测量都要放到它独有的行中。reshape包可为人正直将数据转换为相应的格式。
6、多元方差分析
(1)单因素多元方差分析
library(MASS)
head(UScereal)
attach(UScereal)
y<-cbind(calories,fat,sugars)
aggregate(y,by=list(shelf),FUN=mean)
cov(y)
fit<-manova(y~shelf)
summary(fit)
summary.aov(fit)
(2)评估假设检验
center<-colMeans(y)
n<-nrow(y)
p<-ncol(y)
cov<-cov(y)
d<-mahalanobis(y,center,cov)
coord<-qqplot(qchisq(ppoints(n),df=p),d,main="Q-Q Plot Assessing Multivariate Normality",
ylab="Mahalanobis D2")
abline(a=0,b=1)
identify(coord$x,coord$y,labels=row.names(UScereal))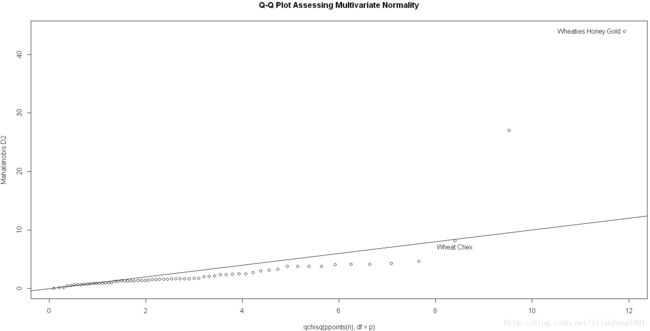
(3)稳健多元方差分析
7、用回归来做ANOVA
library(multcomp)
levels(cholesterol$trt)
fit.aov<-aov(response~trt,data=cholesterol)
summary(fit.aov)
contrasts(cholesterol$trt)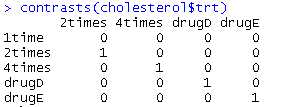
内置对照组
| 对照变量创建方法 | 描述 |
| contr.helmert | 第二个与第一个水平对照 第三个水平对照前两个均值 第四个水平对照前三个的均值 |
| contr.poly | 基于正交多项式的对照,用于趋势分析和等距水平的有序因子 |
| contr.sum | 对照变量之和限制为0,也称作偏差找对,对各水平的均值与所有水平的均值进行比较 |
| contr.treatment | 各水平对照基线水平,也称虚拟编码 |
| contr.SAS | 类似于contr.treatment,只是基线水平变成了最后一个水平 |
可通过contrasts选项,修改lm()默认的对照方法
fit.lm<-lm(response~trt,data=cholesterol,contrasts="contr.helmert")
fit.lmeg:
options(contrasts=c(contr.SAS","contr.helmert"))