在kyphosis数据集建立支持向量机分类器
1. e1701简介
R语言的e1071包提供了对libsvm的接口。库libsvm包括了常用的核,如线性,多项式,RBF,sigmoid等。多分类通过一对一的投票机制(one-against-one voting scheme)而实现。predict()是训练函数,plot()可视化数据,支持向量,决策边界(如果提供的话)。参数调整tune()。
用e1071包中svm函数可以得到与libsvm相同的结果。write.svm()更是可以把R训练得到的结果写为标准的Libsvm格式,以供其他环境下libsvm的使用。下面我们来看看svm()函数的用法。有两种格式都可以。
svm(formula,data=NULL,…,subset,na.action=na.omit,sacle=TRUE) 或者
svm(x, y = NULL, scale = TRUE, type = NULL, kernel = "radial", degree = 3, gamma = if (is.vector(x)) 1 else 1 / ncol(x), coef0 = 0, cost = 1, nu = 0.5, class.weights = NULL, cachesize = 40, tolerance = 0.001, epsilon = 0.1, shrinking = TRUE, cross = 0, probability = FALSE, fitted = TRUE, ..., subset, na.action = na.omit)
【主要参数说明】
formula:分类模型形式,在第二个表达式中可以理解为y~x,即y相当于标签,x相当于特征(变量) 。
data: 数据框。
subset: 可以指定数据集的一部分作为训练数据。 na.cation:缺失值处理,默认为删除缺失数据。
scale: 将数据标准化,中心化,使其均值为0,方差为1,将自动执行。
type: svm的形式。可分为:C-classification ,nu-classification,
one-classification (for novelty detection) ,eps-regression, nu-regression 五种形式。后面两者为做回归时用到。默认为C分类器。
kernel:在非线性可分时,我们引入核函数来做。R中提供的核函数如下:
线性核: : u'*v
多项式核: (gamma*u'*v + coef0)^degree
高斯核: exp(-gamma*|u-v|^2)
sigmoid核:tanh(gamma*u'*v + coef0)
默认为高斯核。顺带说一下,在kernel包中可以自定义核函数。
degree: 多项式核的次数,默认为3
gamma: 除去线性核外,其他核的参数,默认为1/数据维数
coef0: 多项式核与sigmoid核的参数,默认为0.
cost: C分类中惩罚项c的取值
nu: Nu分类,单一分类中nu的值
cross: 做k折交叉验证,计算分类正确性。
2. 对rpart包中的 kyphosis数据集建立支持向量机分类器
2.1 使用svm默认的参数在kyphosis数据集上建立支持向量机分类器
> library(e1071)
> library(rpart)
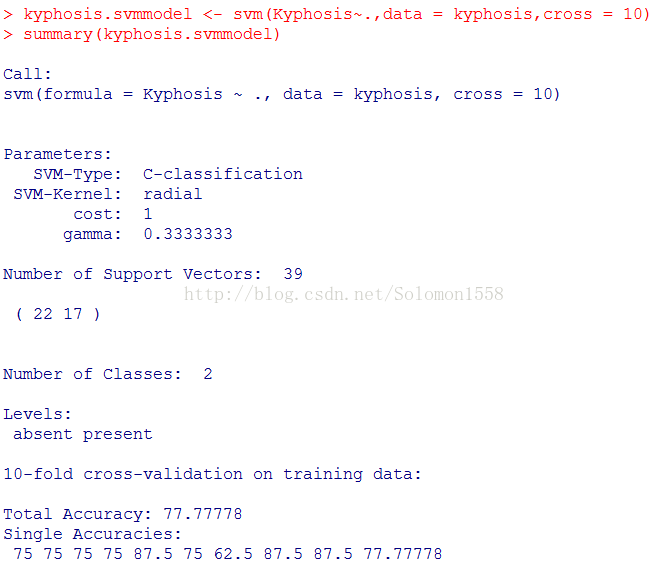
> kyphosis.svmmodel<- svm(Kyphosis~.,data = kyphosis)
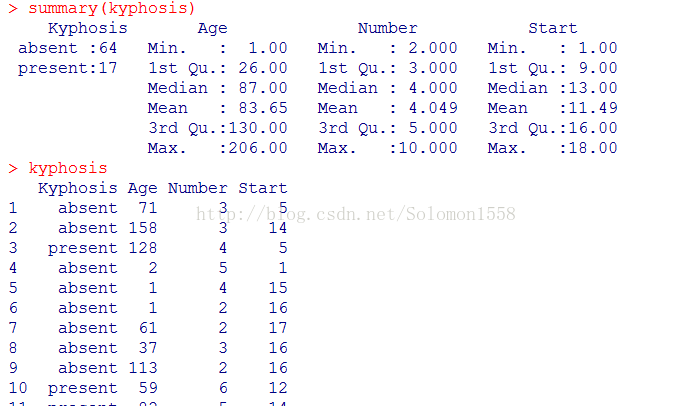
> summary(kyphosis)
图2-1 建立支持向量机分类器
kyphosis数据集是rpart自带的数据集,kyphosis是描述儿童纠正脊柱手术数据集,一共有4个变量。根据变量的英文名称,可以了解到这是以驼背、年龄、编号、开始时间为变量的数据集,一共有81行数据。
图2-2 svm分类器的描述
该模型使用了svm()默认的C分类形式,惩罚项默认为1,核函数是radial,其他核的参数gamma默认为1/数据维数,即1/3,支持向量(支持平面上的样本点)的数目一共39个。
经过十折交叉验证得到总体精确率:77.77778
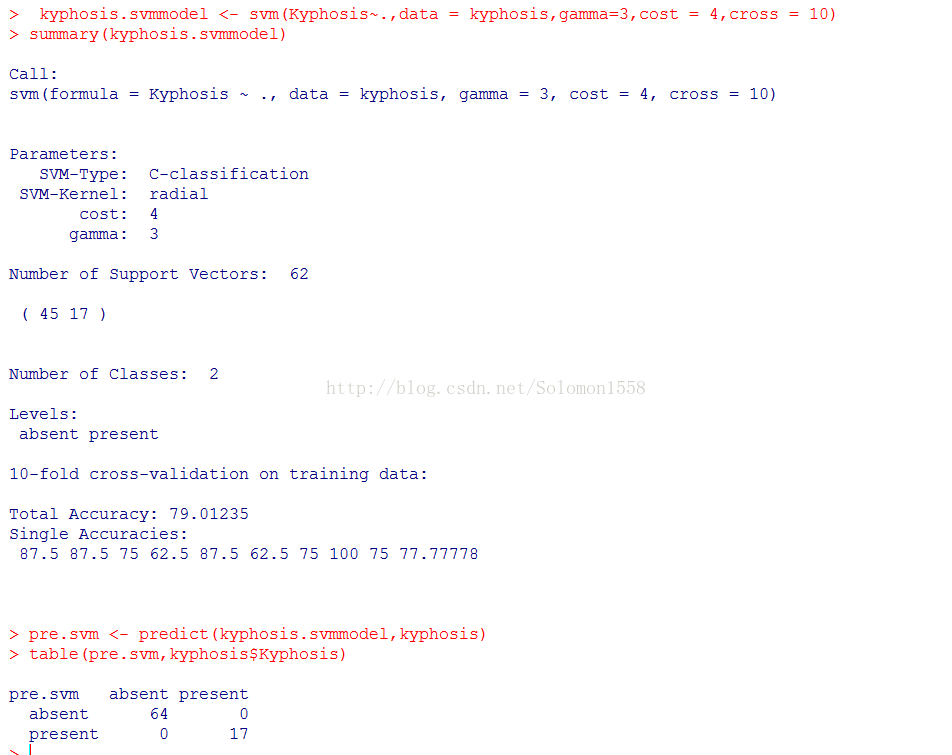
> pre.svm <-predict(kyphosis.svmmodel, kyphosis)
> table(pre.svm,kyphosis$Kyphosis)
图2-3
使用所建模型对样本进行预测,发现absent一项错分了9个样本。
结合10-折交叉验证得到的总体精确率77.77778。我们发现这一个模型并不够优。
2.2 对模型的修改
惩罚因子表示对错分的点加入多少惩罚,当C越大时,分错的点就会更少,但是过拟合的情况可能会比较严重;当C过小时,过拟合的情况相对不严重,但分错的点可能会比较多,模型的准确性将会下降。
将惩罚项cost由默认的1修改2.5。
> kyphosis.svmmodel <- svm(Kyphosis~.,data =kyphosis,cost = 2.5,cross = 10)
> summary(kyphosis.svmmodel)
图2-4
图2-5
可以看到尽管10-折交叉验证的准确率上升至81.48148,但是仍有8个样本分错。
将gamma系数由1/3改为3,将cost改为4得到新的模型。对数据即进行预测发现正确率为100%。
然而10-折交叉验证显示整体准确率为79.01235,比gamma = 1/3,cost=2.5还要低。这说明惩罚因子C可能取得有些大,出现了过拟合的情况。