- 支持向量机(SVM)在肝脏CT/MRI图像分类(肝癌检测)中的应用及实现
猿享天开
医学影像支持向量机机器学习人工智能算法
博主简介:CSDN博客专家、CSDN平台优质创作者,高级开发工程师,数学专业,10年以上C/C++,C#,Java等多种编程语言开发经验,拥有高级工程师证书;擅长C/C++、C#等开发语言,熟悉Java常用开发技术,能熟练应用常用数据库SQLserver,Oracle,mysql,postgresql等进行开发应用,熟悉DICOM医学影像及DICOM协议,业余时间自学JavaScript,Vue,
- 【论文笔记】RAGLAB: A Modular and Research-Oriented Unified Framework for Retrieval-Augmented Generation
AustinCyy
论文笔记论文阅读
论文信息论文标题:RAGLAB:AModularandResearch-OrientedUnifiedFrameworkforRetrieval-AugmentedGeneration-EMNLP24论文作者:XuanwangZhang-NanjingUniversity论文链接:https://arxiv.org/abs/2408.11381代码链接:https://github.com/fat
- 从0到1掌握OpenCV!Python图像处理实战全解析(附代码+案例)
小张在编程
Python学习opencvpython图像处理
引言你有没有想过,手机里的美颜滤镜如何精准识别五官?监控摄像头如何在人流中锁定可疑目标?医学影像软件如何从CT片中快速标注病灶?这些“神奇操作”的背后,往往藏着一个低调的“图像处理神器”——OpenCV。作为Python生态中最受欢迎的计算机视觉库,它用一行行代码将抽象的像素点变成可操作的“数字画布”。今天,我们就从最基础的图像读写开始,手把手带你解锁OpenCV的“十八般武艺”,从图像处理小白变
- Llama改进之——RoPE旋转位置编码
愤怒的可乐
NLP项目实战#LLaMARoPE旋转位置编码
引言旋转位置编码(RotaryPositionEmbedding,RoPE)将绝对相对位置依赖纳入自注意力机制中,以增强Transformer架构的性能。目前很火的大模型LLaMA、QWen等都应用了旋转位置编码。之前在[论文笔记]ROFORMER中对旋转位置编码的原始论文进行了解析,重点推导了旋转位置编码的公式,本文侧重实现,同时尽量简化数学上的推理,详细推理可见最后的参考文章。复数与极坐标复数
- Llama改进之——均方根层归一化RMSNorm
愤怒的可乐
NLP项目实战#llama
引言在学习完GPT2之后,从本文开始进入Llama模型系列。本文介绍Llama模型的改进之RMSNorm(均方根层归一化)。它是由RootMeanSquareLayerNormalization论文提出来的,可以参阅其论文笔记1。LayerNorm层归一化(LayerNorm)对Transformer等模型来说非常重要,它可以帮助稳定训练并提升模型收敛性。LayerNorm针对一个样本所有特征计算
- 基于改进扩散模型与注意力机制的影像到转基因数据预测系统
基于改进扩散模型与注意力机制的影像到转基因数据预测系统1.项目概述本系统利用改进的扩散模型结合注意力机制,从医学影像中预测转基因数据。系统采用PyTorch框架实现,包含数据预处理、模型架构、训练流程和评估指标等完整模块。importtorchimporttorch.nnasnnimporttorch.nn.functionalasFimporttorch.optimasoptimfromtorc
- 基于深度学习的线上问诊系统设计与实现(Python+Django+MySQL)
神经网络15044
深度学习算法神经网络python深度学习django机器学习人工智能算法目标检测
基于深度学习的线上问诊系统设计与实现(Python+Django+MySQL)一、系统概述本系统结合YOLOv8目标检测和ResNet50图像分类算法,构建了一个智能线上问诊平台。系统支持用户上传医学影像(皮肤照片/X光片),自动分析并生成诊断报告,同时提供医生审核功能。二、技术栈后端框架:Django4.2数据库:MySQL8.0深度学习:YOLOv8:皮肤病变区域检测ResNet50:肺炎X光
- 浅谈卷积神经网络(CNN)
cyc&阿灿
cnn人工智能神经网络
卷积神经网络(ConvolutionalNeuralNetworks,CNN)作为深度学习领域最具影响力的架构之一,已在计算机视觉、自然语言处理、医学影像分析等领域取得了革命性突破。本文将系统全面地剖析CNN的核心原理、关键组件、经典模型、数学基础、训练技巧以及最新进展,通过理论解析与代码实践相结合的方式,帮助读者深入掌握这一重要技术。一、CNN基础与核心思想1.1传统神经网络的局限性在处理图像等
- 基于OpenCv的图片倾斜校正系统详细设计与具体代码实现
AI大模型应用之禅
人工智能数学基础计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
基于OpenCv的图片倾斜校正系统详细设计与具体代码实现1.背景介绍1.1图像处理的重要性在当今数字时代,图像处理技术在各个领域都扮演着重要角色。无论是在计算机视觉、模式识别、医学影像、遥感探测还是多媒体处理等领域,图像处理都是不可或缺的核心技术。通过对图像进行预处理、增强、分割、特征提取等操作,可以从图像中获取有价值的信息,为后续的分析和决策提供支持。1.2图像倾斜问题及其影响在实际应用中,由于
- 信创时代技术栈选择与前景分析:国产替代背景下的战略路径与实践指南
猿享天开
信创开发系统安全科技创业创新开发语言
博主简介:CSDN博客专家、CSDN平台优质创作者,高级开发工程师,数学专业,10年以上C/C++,C#,Java等多种编程语言开发经验,拥有高级工程师证书;擅长C/C++、C#等开发语言,熟悉Java常用开发技术,能熟练应用常用数据库SQLserver,Oracle,mysql,postgresql等进行开发应用,熟悉DICOM医学影像及DICOM协议,业余时间自学JavaScript,Vue,
- SimpleITK——创建nrrd体素模型
bianguanyue
C#c#健康医疗算法
在介绍如何生成nrrd前,了解一下为什么医学影像上一般使用nrrd的体素模型?为什么医学影像上一般使用nrrd的体素模型?在医学影像领域,NRRD(NearlyRawRasterData)格式被广泛用于存储体素模型(如CT、MRI数据),主要基于以下技术优势:1.灵活的数据存储方式支持原始数据无损存储NRRD可直接存储未经压缩的体素数据(如16位整型、32位浮点),避免DICOM等格式
- 产教融合3.0时代:数字影像产业园‘人才+产业+创新’生态闭环构建
cdsmjt
企业微信
产教融合3.0时代,数字影像产业园构建“人才+产业+创新”生态闭环,需要从以下几个关键环节入手:一、人才培养:以产业需求为导向校企深度合作:打破传统“单向输送”模式,构建双向互动机制。高校与企业共同制定人才培养方案,课程设置紧贴产业前沿技术和岗位需求。实战化教学:建设实训基地,引入真实项目,让学生在实践中掌握技能。例如,可以参照“雅职·海信人工智能医学影像三维重建生产性实训基地”模式,打造高仿真的
- vtk和opencv和opengl直接的区别是什么?
only-lucky
opencv人工智能计算机视觉
简介VTK、OpenCV和OpenGL是三个在计算机图形学、图像处理和可视化领域广泛使用的工具库,但它们在功能、应用场景和底层技术上存在显著差异。以下是它们的核心区别和特点对比:1.核心功能与定位工具核心功能主要应用领域VTK(VisualizationToolkit)三维可视化&科学计算,提供高级渲染、体绘制、交互式可视化医学影像、地质建模、流体力学仿真OpenCV(OpenSourceComp
- 掌握Python:从基础到AI开发的首选语言
博主简介:CSDN博客专家、CSDN平台优质创作者,高级开发工程师,数学专业,10年以上C/C++,C#,Java等多种编程语言开发经验,拥有高级工程师证书;擅长C/C++、C#等开发语言,熟悉Java常用开发技术,能熟练应用常用数据库SQLserver,Oracle,mysql,postgresql等进行开发应用,熟悉DICOM医学影像及DICOM协议,业余时间自学JavaScript,Vue,
- 基于深度学习的智能图像语义分割系统:技术与实践
Blossom.118
机器学习与人工智能深度学习人工智能python分类音视频机器学习sklearn
前言图像语义分割是计算机视觉领域中的一个重要任务,其目标是将图像中的每个像素分配到预定义的语义类别中。这一技术在自动驾驶、医学影像分析、机器人视觉等多个领域有着广泛的应用。近年来,深度学习技术,尤其是卷积神经网络(CNN)及其变体,为图像语义分割带来了显著的改进。本文将详细介绍基于深度学习的智能图像语义分割系统的原理、实现方法以及实际应用案例。一、图像语义分割的基本概念1.1什么是图像语义分割?图
- 论文笔记 <交通灯><多智能体>CoLight管理交通灯
青椒大仙KI11
论文阅读
今天看的是论文Colight:学习网络级合作进行交通信号控制论文提出的CoLight模型是一种基于强化学习和图注意力网络的交通信号灯控制方法,旨在解决城市道路网络中的交通信号的写作问题,提升车辆通行效率。问题定义为:将交通信号控制问题建模为马尔可夫博弈,每个路口由一个智能体控制,智能体通过观察部分系统状态(当前相位和各车道车辆数),选择动作(下一时间段的相位),目标是最小化路口周围车道的平均队列长
- 《基于超声的深度学习模型用于降低BI-RADS 4A乳腺病变的恶性率》论文笔记 MobileNet
往事随风、、
论文笔记机器学习深度学习论文阅读人工智能机器学习健康医疗
《APPLICATIONOFDEEPLEARNINGTOREDUCETHERATEOFMALIGNANCYAMONGBI-RADS4ABREASTLESIONSBASEDONULTRASONOGRAPHY》《基于超声的深度学习模型用于降低BI-RADS4A乳腺病变的恶性率》原文地址:链接文章目录摘要简介方法患者图像获取与处理深度学习模型统计分析结果讨论结论摘要本研究旨在开发一个基于超声(US)图像
- 基于深度学习的智能图像分割系统:技术与实践
Blossom.118
机器学习与人工智能深度学习人工智能python机器学习tensorflow神经网络sklearn
前言图像分割是计算机视觉领域中的一个核心任务,其目标是将图像划分为多个有意义的区域或对象。图像分割在医学影像分析、自动驾驶、安防监控等多个领域有着广泛的应用。近年来,深度学习技术,尤其是卷积神经网络(CNN)及其变体,为图像分割带来了显著的改进。本文将详细介绍基于深度学习的智能图像分割系统的原理、实现方法以及实际应用案例。一、图像分割的基本概念1.1什么是图像分割?图像分割是一种将图像划分为多个互
- 图像灰度化处理技术与应用
mkmk00
本文还有配套的精品资源,点击获取简介:灰度化是图像处理的基础技术,能将彩色图像转换为数据量更小的灰阶图像。本文将深入探讨灰度化的原理和方法,包括直接平均法、权重平均法、最大值法、最小值法以及YUV色彩空间转换等,并分析灰度化在图像处理中的作用,例如数据量减少、简化算法、提高处理速度和易于分析。文章还介绍了灰度化在医学影像、文档扫描、车牌识别等实际应用中的例子,以及如何根据具体需求选择灰度化策略。1
- 论文笔记--Language Models are Unsupervised Multitask Learners
Isawany
论文阅读论文阅读语言模型transformerchatgpt自然语言处理
论文笔记GPT-2--LanguageModelsareUnsupervisedMultitaskLearners1.文章简介2.文章导读2.1概括2.2文章重点技术2.2.1数据集WebText2.2.2分词方法3.GPT-1&GPT-24.文章亮点5.原文传送门6.References1.文章简介标题:LanguageModelsareUnsupervisedMultitaskLearners
- You Only Look Once Unified, Real-Time Object Detection论文笔记
__Lo__
目标检测论文阅读深度学习
文章结构统一检测框架(UnifiledDetection)核心思想YOLO将目标检测视为一个端到端的回归问题,输入的图像经过SingleForwardPass,直接输出物体的信息(边界框的位置、边界框的置信度、类别概率);优势在于速度快,全局理解上下文,这里全局理解上下文的意思是识别物体和背景的关系,减少误检。网络设计网格划分(GridDivision)将图像划分为一个S×S的网格,文中S=7;共
- 基于大模型预测急性横贯性脊髓炎的综合技术方案研究报告大纲
LCG元
大模型医疗研究-方案大纲方案大纲
目录一、引言二、技术方案总体架构三、术前预测与决策四、术中监测与决策支持五、术后护理与康复指导六、统计分析与技术验证七、实验验证与证据支持八、健康教育与指导九、结论与展望一、引言(一)研究背景急性横贯性脊髓炎的临床现状与挑战阐述急性横贯性脊髓炎的发病率、致残率以及对患者生活质量的严重影响,强调准确预测和精准治疗的重要性。大模型技术在医疗领域的应用前景简述大模型在医学影像分析、疾病诊断与预测等方面的
- 计算机视觉生物启发视觉算法:如何模拟人眼与大脑?
xcLeigh
计算机视觉CV计算机视觉算法人工智能AI视觉生物
计算机视觉生物启发视觉算法:如何模拟人眼与大脑?一、前言二、人眼与大脑视觉系统基础概念2.1人眼的生理结构与功能2.2大脑视觉皮层的信息处理机制三、生物启发视觉算法的核心技术3.1视网膜启发的特征提取算法3.2视觉皮层启发的分层特征学习算法3.3注意力机制的生物启发实现四、生物启发视觉算法的应用案例4.1智能监控与安防4.2自动驾驶4.3医学影像分析五、生物启发视觉算法面临的挑战与解决方案5.1生
- 基于KAN+Transformer的专业领域建模方法论
乡土老农
transformer深度学习人工智能
一、专业领域KAN方法创新路径领域函数分解策略•数学建模:针对专业领域特性设计专用基函数组合•医学影像:采用小波变换基函数分解图像特征```pythonclassWaveletKAN(nn.Module):def__init__(self):self.wavelet_basis=nn.Parameter(torch.randn(8,32,3))#8通道小波基defforward(self,x):r
- 【论文笔记】UnifiedQA:新SOTA,生成模型一统问答任务
iLuz
深度学习自然语言处理
目录引言模型介绍1.输入格式2.实验结果总结引言问答任务有多种形式,常见的有抽取式问答(EX)、摘要式问答(AB)、多选题式问答(MC)、判断式问答(YN)。一般的解决方案是针对不同形式的问答任务设计不同的模型。例如,抽取式问答、多选题式问答、判断式问答可以转化为分类任务,摘要式问答可以转换为生成任务。尽管任务形式不同,但模型所需的语义理解和推理能力是共通的,或许不需要format-special
- [论文笔记] [2008] [ICML] Extracting and Composing Robust Features with Denoising Autoencoders
Alexzhuan
DL神经网络机器学习
在06年以前,想要去训练一个多层的神经网络是比较困难的,主要的问题是超过两层的模型,当时没有好的策略或方法使模型优化的很好,得不到预期的效果。在06年,Hinton提出的stackedautoencoders改变了当时的情况,那时候的研究者就开始关注各种自编码模型以及相应的堆叠模型。这篇的作者提出的DAE(DenoisingAutoencoders)就是当时蛮有影响力的工作。那个时候多层模型效果得
- 【凌智视觉模块】RV1106上部署YOLOv5目标检测及量化原理简介
位东风
视觉模型部署实践YOLO目标检测人工智能嵌入式硬件计算机视觉
目标检测简介目标检测是计算机视觉中的一个重要领域,它不仅要求模型能够识别图像中出现的对象属于哪一类(分类问题),还要求确定这些对象在图像中的具体位置(通常通过边界框来表示)。目标检测可以应用于多种场景,如自动驾驶、安防监控、医学影像分析等。YOLO(YouOnlyLookOnce)是一种流行的目标检测算法系列,而YOLOv5是其中的一个版本。YOLOv5由Ultralytics公司开发,并不是正式
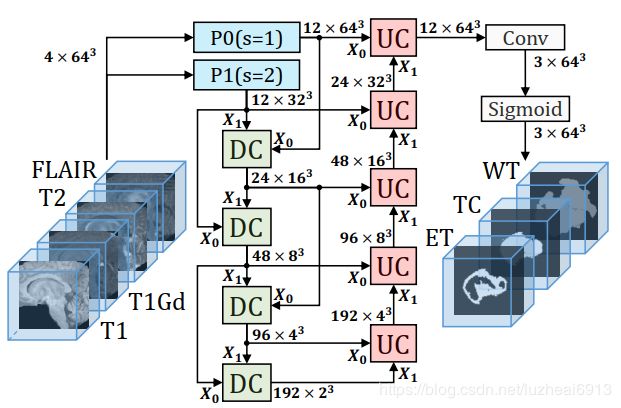
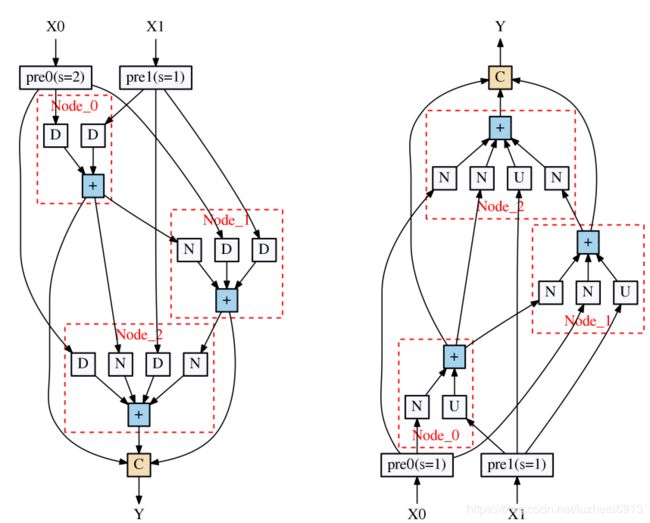
- 跨视角差异-依赖网络用于体积医学图像分割|文献速递-生成式模型与transformer在医学影像中的应用
Title题目Cross-viewdiscrepancy-dependencynetworkforvolumetricmedicalimagesegmentation跨视角差异-依赖网络用于体积医学图像分割01文献速递介绍医学图像分割旨在从原始图像中分离出受试者的解剖结构(例如器官和肿瘤),并为每个像素分配语义类别,这在许多临床应用中起着至关重要的作用,如器官建模、疾病诊断和治疗规划(Shamsh
- Docker 部署医学影像 DICOM 服务器 Orthanc
Pole丶逐
Dockerdocker服务器容器
Orthanc是一个轻量级的,基于REST的DICOM服务器,主要用于卫生保健和医疗研究。Orthanc可将任意运行了Windows和Linux的计算机变成DICOM存储(或者说是一个小的PACS系统),其架构是轻量级的,没有复杂的数据库管理,不依赖于第三方软件。1.下载Orthanc镜像#拉取镜像:选择有Web页面的版本dockerpulljodogne/orthanc-plugins2.创建挂
- 医学影像基础与实践:基于传统算法的CT影像探索
t0_54program
大数据与人工智能算法个人开发
在医学影像领域,人们往往容易将目光聚焦于人工智能(AI)技术,然而,理解和掌握基础的图像处理算法同样至关重要。这不仅有助于我们深入了解医学影像的本质,还能在不依赖深度学习的情况下,明确其应用边界。本次,我们就一同通过实践教程,深入了解医学影像,特别是计算机断层扫描(CT)技术。CT成像基础CT扫描的物理学原理CT利用X射线束来获取人体的3D像素强度。具体来说,加热的阴极释放出高能电子束,这些电子束
- 开发者关心的那些事
圣子足道
ios游戏编程apple支付
我要在app里添加IAP,必须要注册自己的产品标识符(product identifiers)。产品标识符是什么?
产品标识符(Product Identifiers)是一串字符串,它用来识别你在应用内贩卖的每件商品。App Store用产品标识符来检索产品信息,标识符只能包含大小写字母(A-Z)、数字(0-9)、下划线(-)、以及圆点(.)。你可以任意排列这些元素,但我们建议你创建标识符时使用
- 负载均衡器技术Nginx和F5的优缺点对比
bijian1013
nginxF5
对于数据流量过大的网络中,往往单一设备无法承担,需要多台设备进行数据分流,而负载均衡器就是用来将数据分流到多台设备的一个转发器。
目前有许多不同的负载均衡技术用以满足不同的应用需求,如软/硬件负载均衡、本地/全局负载均衡、更高
- LeetCode[Math] - #9 Palindrome Number
Cwind
javaAlgorithm题解LeetCodeMath
原题链接:#9 Palindrome Number
要求:
判断一个整数是否是回文数,不要使用额外的存储空间
难度:简单
分析:
题目限制不允许使用额外的存储空间应指不允许使用O(n)的内存空间,O(1)的内存用于存储中间结果是可以接受的。于是考虑将该整型数反转,然后与原数字进行比较。
注:没有看到有关负数是否可以是回文数的明确结论,例如
- 画图板的基本实现
15700786134
画图板
要实现画图板的基本功能,除了在qq登陆界面中用到的组件和方法外,还需要添加鼠标监听器,和接口实现。
首先,需要显示一个JFrame界面:
public class DrameFrame extends JFrame { //显示
- linux的ps命令
被触发
linux
Linux中的ps命令是Process Status的缩写。ps命令用来列出系统中当前运行的那些进程。ps命令列出的是当前那些进程的快照,就是执行ps命令的那个时刻的那些进程,如果想要动态的显示进程信息,就可以使用top命令。
要对进程进行监测和控制,首先必须要了解当前进程的情况,也就是需要查看当前进程,而 ps 命令就是最基本同时也是非常强大的进程查看命令。使用该命令可以确定有哪些进程正在运行
- Android 音乐播放器 下一曲 连续跳几首歌
肆无忌惮_
android
最近在写安卓音乐播放器的时候遇到个问题。在MediaPlayer播放结束时会回调
player.setOnCompletionListener(new OnCompletionListener() {
@Override
public void onCompletion(MediaPlayer mp) {
mp.reset();
Log.i("H
- java导出txt文件的例子
知了ing
javaservlet
代码很简单就一个servlet,如下:
package com.eastcom.servlet;
import java.io.BufferedOutputStream;
import java.io.IOException;
import java.net.URLEncoder;
import java.sql.Connection;
import java.sql.Resu
- Scala stack试玩, 提高第三方依赖下载速度
矮蛋蛋
scalasbt
原文地址:
http://segmentfault.com/a/1190000002894524
sbt下载速度实在是惨不忍睹, 需要做些配置优化
下载typesafe离线包, 保存为ivy本地库
wget http://downloads.typesafe.com/typesafe-activator/1.3.4/typesafe-activator-1.3.4.zip
解压r
- phantomjs安装(linux,附带环境变量设置) ,以及casperjs安装。
alleni123
linuxspider
1. 首先从官网
http://phantomjs.org/下载phantomjs压缩包,解压缩到/root/phantomjs文件夹。
2. 安装依赖
sudo yum install fontconfig freetype libfreetype.so.6 libfontconfig.so.1 libstdc++.so.6
3. 配置环境变量
vi /etc/profil
- JAVA IO FileInputStream和FileOutputStream,字节流的打包输出
百合不是茶
java核心思想JAVA IO操作字节流
在程序设计语言中,数据的保存是基本,如果某程序语言不能保存数据那么该语言是不可能存在的,JAVA是当今最流行的面向对象设计语言之一,在保存数据中也有自己独特的一面,字节流和字符流
1,字节流是由字节构成的,字符流是由字符构成的 字节流和字符流都是继承的InputStream和OutPutStream ,java中两种最基本的就是字节流和字符流
类 FileInputStream
- Spring基础实例(依赖注入和控制反转)
bijian1013
spring
前提条件:在http://www.springsource.org/download网站上下载Spring框架,并将spring.jar、log4j-1.2.15.jar、commons-logging.jar加载至工程1.武器接口
package com.bijian.spring.base3;
public interface Weapon {
void kil
- HR看重的十大技能
bijian1013
提升能力HR成长
一个人掌握何种技能取决于他的兴趣、能力和聪明程度,也取决于他所能支配的资源以及制定的事业目标,拥有过硬技能的人有更多的工作机会。但是,由于经济发展前景不确定,掌握对你的事业有所帮助的技能显得尤为重要。以下是最受雇主欢迎的十种技能。 一、解决问题的能力 每天,我们都要在生活和工作中解决一些综合性的问题。那些能够发现问题、解决问题并迅速作出有效决
- 【Thrift一】Thrift编译安装
bit1129
thrift
什么是Thrift
The Apache Thrift software framework, for scalable cross-language services development, combines a software stack with a code generation engine to build services that work efficiently and s
- 【Avro三】Hadoop MapReduce读写Avro文件
bit1129
mapreduce
Avro是Doug Cutting(此人绝对是神一般的存在)牵头开发的。 开发之初就是围绕着完善Hadoop生态系统的数据处理而开展的(使用Avro作为Hadoop MapReduce需要处理数据序列化和反序列化的场景),因此Hadoop MapReduce集成Avro也就是自然而然的事情。
这个例子是一个简单的Hadoop MapReduce读取Avro格式的源文件进行计数统计,然后将计算结果
- nginx定制500,502,503,504页面
ronin47
nginx 错误显示
server {
listen 80;
error_page 500/500.html;
error_page 502/502.html;
error_page 503/503.html;
error_page 504/504.html;
location /test {return502;}}
配置很简单,和配
- java-1.二叉查找树转为双向链表
bylijinnan
二叉查找树
import java.util.ArrayList;
import java.util.List;
public class BSTreeToLinkedList {
/*
把二元查找树转变成排序的双向链表
题目:
输入一棵二元查找树,将该二元查找树转换成一个排序的双向链表。
要求不能创建任何新的结点,只调整指针的指向。
10
/ \
6 14
/ \
- Netty源码学习-HTTP-tunnel
bylijinnan
javanetty
Netty关于HTTP tunnel的说明:
http://docs.jboss.org/netty/3.2/api/org/jboss/netty/channel/socket/http/package-summary.html#package_description
这个说明有点太简略了
一个完整的例子在这里:
https://github.com/bylijinnan
- JSONUtil.serialize(map)和JSON.toJSONString(map)的区别
coder_xpf
jqueryjsonmapval()
JSONUtil.serialize(map)和JSON.toJSONString(map)的区别
数据库查询出来的map有一个字段为空
通过System.out.println()输出 JSONUtil.serialize(map): {"one":"1","two":"nul
- Hibernate缓存总结
cuishikuan
开源sshjavawebhibernate缓存三大框架
一、为什么要用Hibernate缓存?
Hibernate是一个持久层框架,经常访问物理数据库。
为了降低应用程序对物理数据源访问的频次,从而提高应用程序的运行性能。
缓存内的数据是对物理数据源中的数据的复制,应用程序在运行时从缓存读写数据,在特定的时刻或事件会同步缓存和物理数据源的数据。
二、Hibernate缓存原理是怎样的?
Hibernate缓存包括两大类:Hib
- CentOs6
dalan_123
centos
首先su - 切换到root下面1、首先要先安装GCC GCC-C++ Openssl等以来模块:yum -y install make gcc gcc-c++ kernel-devel m4 ncurses-devel openssl-devel2、再安装ncurses模块yum -y install ncurses-develyum install ncurses-devel3、下载Erang
- 10款用 jquery 实现滚动条至页面底端自动加载数据效果
dcj3sjt126com
JavaScript
无限滚动自动翻页可以说是web2.0时代的一项堪称伟大的技术,它让我们在浏览页面的时候只需要把滚动条拉到网页底部就能自动显示下一页的结果,改变了一直以来只能通过点击下一页来翻页这种常规做法。
无限滚动自动翻页技术的鼻祖是微博的先驱:推特(twitter),后来必应图片搜索、谷歌图片搜索、google reader、箱包批发网等纷纷抄袭了这一项技术,于是靠滚动浏览器滚动条
- ImageButton去边框&Button或者ImageButton的背景透明
dcj3sjt126com
imagebutton
在ImageButton中载入图片后,很多人会觉得有图片周围的白边会影响到美观,其实解决这个问题有两种方法
一种方法是将ImageButton的背景改为所需要的图片。如:android:background="@drawable/XXX"
第二种方法就是将ImageButton背景改为透明,这个方法更常用
在XML里;
<ImageBut
- JSP之c:foreach
eksliang
jspforearch
原文出自:http://www.cnblogs.com/draem0507/archive/2012/09/24/2699745.html
<c:forEach>标签用于通用数据循环,它有以下属性 属 性 描 述 是否必须 缺省值 items 进行循环的项目 否 无 begin 开始条件 否 0 end 结束条件 否 集合中的最后一个项目 step 步长 否 1
- Android实现主动连接蓝牙耳机
gqdy365
android
在Android程序中可以实现自动扫描蓝牙、配对蓝牙、建立数据通道。蓝牙分不同类型,这篇文字只讨论如何与蓝牙耳机连接。
大致可以分三步:
一、扫描蓝牙设备:
1、注册并监听广播:
BluetoothAdapter.ACTION_DISCOVERY_STARTED
BluetoothDevice.ACTION_FOUND
BluetoothAdapter.ACTION_DIS
- android学习轨迹之四:org.json.JSONException: No value for
hyz301
json
org.json.JSONException: No value for items
在JSON解析中会遇到一种错误,很常见的错误
06-21 12:19:08.714 2098-2127/com.jikexueyuan.secret I/System.out﹕ Result:{"status":1,"page":1,&
- 干货分享:从零开始学编程 系列汇总
justjavac
编程
程序员总爱重新发明轮子,于是做了要给轮子汇总。
从零开始写个编译器吧系列 (知乎专栏)
从零开始写一个简单的操作系统 (伯乐在线)
从零开始写JavaScript框架 (图灵社区)
从零开始写jQuery框架 (蓝色理想 )
从零开始nodejs系列文章 (粉丝日志)
从零开始编写网络游戏
- jquery-autocomplete 使用手册
macroli
jqueryAjax脚本
jquery-autocomplete学习
一、用前必备
官方网站:http://bassistance.de/jquery-plugins/jquery-plugin-autocomplete/
当前版本:1.1
需要JQuery版本:1.2.6
二、使用
<script src="./jquery-1.3.2.js" type="text/ja
- PLSQL-Developer或者Navicat等工具连接远程oracle数据库的详细配置以及数据库编码的修改
超声波
oracleplsql
在服务器上将Oracle安装好之后接下来要做的就是通过本地机器来远程连接服务器端的oracle数据库,常用的客户端连接工具就是PLSQL-Developer或者Navicat这些工具了。刚开始也是各种报错,什么TNS:no listener;TNS:lost connection;TNS:target hosts...花了一天的时间终于让PLSQL-Developer和Navicat等这些客户
- 数据仓库数据模型之:极限存储--历史拉链表
superlxw1234
极限存储数据仓库数据模型拉链历史表
在数据仓库的数据模型设计过程中,经常会遇到这样的需求:
1. 数据量比较大; 2. 表中的部分字段会被update,如用户的地址,产品的描述信息,订单的状态等等; 3. 需要查看某一个时间点或者时间段的历史快照信息,比如,查看某一个订单在历史某一个时间点的状态, 比如,查看某一个用户在过去某一段时间内,更新过几次等等; 4. 变化的比例和频率不是很大,比如,总共有10
- 10点睛Spring MVC4.1-全局异常处理
wiselyman
spring mvc
10.1 全局异常处理
使用@ControllerAdvice注解来实现全局异常处理;
使用@ControllerAdvice的属性缩小处理范围
10.2 演示
演示控制器
package com.wisely.web;
import org.springframework.stereotype.Controller;
import org.spring