sklearn实现支持向量机
需要用到的函数
- 引入sklearn自带数据集:sklearn.datasets.make_blobs(n_samples=100, n_features=2, centers=3, cluster_std=1.0, center_box=(-10.0, 10.0), shuffle=True, random_state=None)
- sklearn.datasets.samples_generator.make_circles() 引入圆形数据集
n_samples: 待生成的样本的总数。
centers: 要生成的样本中心(类别)数,或者是确定的中心点。
cluster_std: 每个类别的方差,例如我们希望生成2类数据,其中一类比另一类具有更大的方差,可以将cluster_std设置为[1.0,3.0]。
- 径向基函数sklearn.svm.SVC()
model=svm.SVC() #引入分类器
model.fit(X,y) #训练并生成模型
代码
首先做一个简单的线性可分的例子,这里直接用的sklearn中的数据集。
利用 sklearn.datasets.make_blobs生成数据
from sklearn.datasets.samples_generator import make_blobs #生成数据集
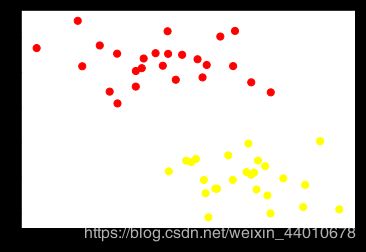
X,y=make_blobs(n_samples=50,centers=2,random_state=0,cluster_std=0.6) #n_samples=50意思取50个点,centers=2意思是将数据分为两
plt.scatter(X[:,0],X[:,1],c=y,s=50,cmap='autumn') #将图像展示出来
plt.show()
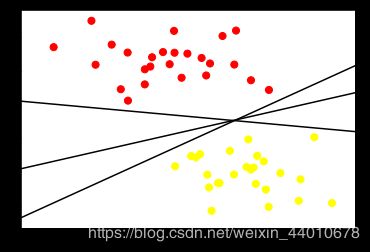
xfit = np.linspace(-1, 3.5)
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plt.plot(xfit, xfit+0.65, '-k')
plt.plot(xfit, 0.5*xfit+1.6, '-k')
plt.plot(xfit, -0.2*xfit+2.9, '-k')
plt.xlim(-1, 3.5)
plt.show()
from sklearn.svm import SVC # "Support vector classifier" #支持向量机分类器
model = SVC(kernel='linear', C=1E10)
model.fit(X, y)
构造出支持向量机
def plot_svc_decision_function(model, ax=None, plot_support=True):
"""Plot the decision function for a 2D SVC"""
if ax is None:
ax = plt.gca()
xlim = ax.get_xlim()
ylim = ax.get_ylim()
x = np.linspace(xlim[0], xlim[1], 30)
y = np.linspace(ylim[0], ylim[1], 30)
Y, X = np.meshgrid(y, x)
xy = np.vstack([X.ravel(), Y.ravel()]).T
P = model.decision_function(xy).reshape(X.shape)
ax.contour(X, Y, P, colors='k',levels=[-1, 0, 1], alpha=0.5,linestyles=['--', '-', '--'])
if plot_support:
ax.scatter(model.support_vectors_[:, 0],model.support_vectors_[:, 1],s=300, linewidth=1, facecolors='none');
ax.set_xlim(xlim)
ax.set_ylim(ylim)
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(model);
plt.show()
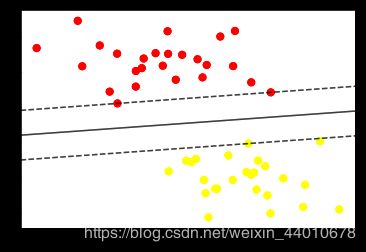
其中在边界上的两个红点和一个黄点在决策边界上,是支持向量,其α值不为0。这三个点的坐标可以由model.support_vectors_ 得出。
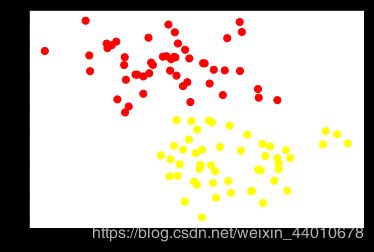
这个分类器的成功的关键在于:为了拟合,只有支持向量的位置是重要的;任何远离边距的点,都不会影响拟合。边界之外的点无论有多少都不会对其造成影响,下面来对比一下数据为60和120时的区别。
def plot_svm(N=10, ax=None):
X, y = make_blobs(n_samples=200, centers=2,
random_state=0, cluster_std=0.60)
X = X[:N]
y = y[:N]
model = SVC(kernel='linear', C=1E10)
model.fit(X, y)
ax = ax or plt.gca()
ax.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
ax.set_xlim(-1, 4)
ax.set_ylim(-1, 6)
plot_svc_decision_function(model, ax)
fig, ax = plt.subplots(1, 2, figsize=(16, 6))
fig.subplots_adjust(left=0.0625, right=0.95, wspace=0.1)
for axi, N in zip(ax, [60, 120]):#左侧的数据60个样本点,右侧的数据为120样本点。
plot_svm(N, axi)
axi.set_title('N = {0}'.format(N))
plt.show()

由上图可以看出它们的样本密度不一样,但是它们的决策边界却是一模一样的。这就意味着样本多和样本少没什么差别,这主要是因为没有引入新得支持向量,意思就是说只要边界上的点不变就不会对决策边界造成影响。
核函数
接下来引入核函数,来看看核函数的威力,真的感觉好厉害!
引入一个线性不可分的数据集。
from sklearn.datasets.samples_generator import make_circles
X, y = make_circles(100, factor=.1, noise=.1)
clf = SVC(kernel='linear').fit(X, y)
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(clf, plot_support=False);
plt.show()
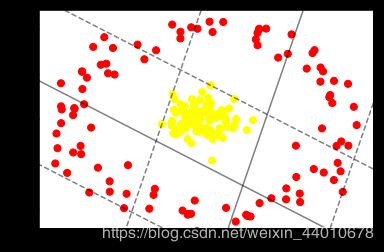
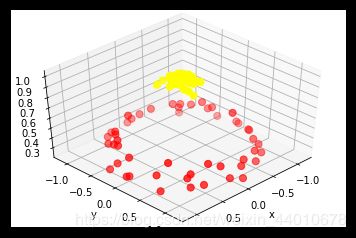
可以看出来用线性分类无论怎么画线也不可能分好。所以要使用核变换来分类,进行核变换之前,先看一看在数据在高维空间下的映射:
from mpl_toolkits import mplot3d
r=np.exp(-(X**2).sum(1))
def plot_3D(elev=30, azim=30, X=X, y=y):
ax = plt.subplot(projection='3d')
ax.scatter3D(X[:, 0], X[:, 1], r, c=y, s=50, cmap='autumn')
ax.view_init(elev=elev, azim=azim)
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.set_zlabel('z')
plot_3D(elev=45,azim=45,X=X,y=y)
plt.show()
clf = SVC(kernel='rbf', C=1E6) #引入径向基 函数
clf.fit(X, y)
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(clf)
plt.scatter(clf.support_vectors_[:, 0], clf.support_vectors_[:, 1],
s=300, lw=1, facecolors='none');
plt.show()
效果如下:
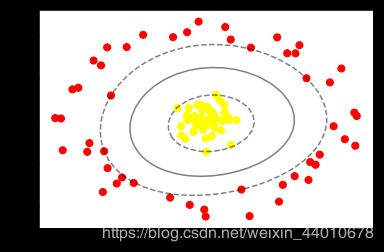
可以清楚的看出来效果要比刚才好的多,将线性不可分的两堆数据分割开来。
调节参数-软间隔问题
SVM模型有两个非常重要的参数C与gamma。其中 C是惩罚系数,即对误差的宽容度。c越高,说明越不能容忍出现误差,容易过拟合。C越小,容易欠拟合。C过大或过小,泛化能力变差
gamma是选择RBF函数作为kernel后,该函数自带的一个参数。隐含地决定了数据映射到新的特征空间后的分布,gamma越大,支持向量越少,gamma值越小,支持向量越多。
分别调节C和gamma来看一下对结果的影响:
调节参数C
C趋近于无穷大时,意味着分类严格不能有错误。C趋于很小时,意味着可以有更大的容忍。
引入一个离散度较大的数据集:
X, y = make_blobs(n_samples=100, centers=2,
random_state=0, cluster_std=0.8) #将离散度改为0.8
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn');
plt.show()
X, y = make_blobs(n_samples=100, centers=2,random_state=0, cluster_std=0.8)
fig, ax = plt.subplots(1, 2, figsize=(16, 6))
fig.subplots_adjust(left=0.0625, right=0.95, wspace=0.1)
for axi, C in zip(ax, [20, 0.2]): #将C分别设定为20和0.2,看其对结果的影响。
model = SVC(kernel='linear', C=C).fit(X, y)
axi.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(model, axi)
axi.scatter(model.support_vectors_[:, 0],model.support_vectors_[:, 1],s=300, lw=1, facecolors='none');
axi.set_title('C = {0:.1f}'.format(C), size=14)
plt.show()
效果如下:
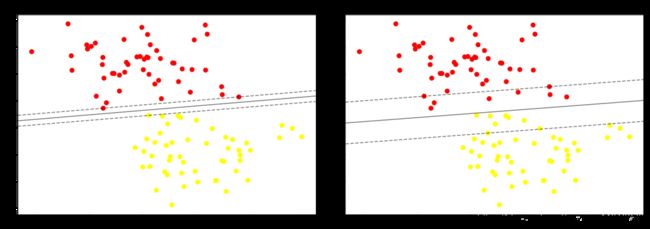
左边这幅图C值比较大,要求比较严格,不能分错东西,隔离带中没有进入任何一个点,但是隔离带的距离比较小,泛化能力比较差。右边这幅图C值比较小,要求相对来说比较松一些,隔离带较大,但是隔离带中进入了很多的黄点和红点。那么C大一点好还是小一点好呢?这需要考虑实际问题,可以进行K折交叉验证来得出最合适的C值。
调节参数gamma
X, y = make_blobs(n_samples=100, centers=2,random_state=0, cluster_std=1.1)
fig, ax = plt.subplots(1, 2, figsize=(16, 6))
fig.subplots_adjust(left=0.0625, right=0.95, wspace=0.1)
for axi, gamma in zip(ax, [20, 0.1]): #比较了一下gamma为20和0.1对结果的影响
model = SVC(kernel='rbf', gamma=gamma).fit(X, y)
axi.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(model, axi)
axi.scatter(model.support_vectors_[:, 0],model.support_vectors_[:, 1],s=300, lw=1, facecolors='none');
axi.set_title('gamma = {0:.1f}'.format(gamma), size=14)
plt.show()
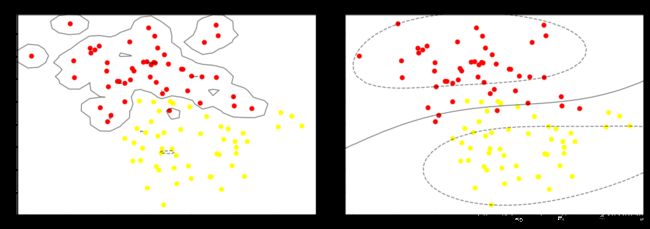
左边的图边界比较复杂,这也意味着泛化能力更弱,右边的图比较精简,泛化能力较强。一般会选择泛化能力较强的。
完整代码
# 导入标准库
import numpy as np
import matplotlib.pyplot as plt
from scipy import stats
from sklearn.datasets.samples_generator import make_blobs
#导入数据集
X,y=make_blobs(n_samples=50,centers=2,random_state=0,cluster_std=0.6) #n_samples=50意思取50个点,centers=2意思是将数据分为两
plt.scatter(X[:,0],X[:,1],c=y,s=50,cmap='autumn')
#绘制决策边界
xfit = np.linspace(-1, 3.5)
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plt.plot(xfit, xfit+0.65, '-k')
plt.plot(xfit, 0.5*xfit+1.6, '-k')
plt.plot(xfit, -0.2*xfit+2.9, '-k')
plt.xlim(-1, 3.5)
#构造支持向量机
from sklearn.svm import SVC # "Support vector classifier"
model = SVC(kernel='linear', C=1E10)
model.fit(X, y)
def plot_svc_decision_function(model, ax=None, plot_support=True):
"""Plot the decision function for a 2D SVC"""
if ax is None:
ax = plt.gca()
xlim = ax.get_xlim()
ylim = ax.get_ylim()
x = np.linspace(xlim[0], xlim[1], 30)
y = np.linspace(ylim[0], ylim[1], 30)
Y, X = np.meshgrid(y, x)
xy = np.vstack([X.ravel(), Y.ravel()]).T
P = model.decision_function(xy).reshape(X.shape)
# 绘制决策边界
ax.contour(X, Y, P, colors='k',
levels=[-1, 0, 1], alpha=0.5,
linestyles=['--', '-', '--'])
if plot_support:
ax.scatter(model.support_vectors_[:, 0],model.support_vectors_[:, 1],s=300, linewidth=1, facecolors='none');
ax.set_xlim(xlim)
ax.set_ylim(ylim)
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(model);
#引入核函数
from sklearn.datasets.samples_generator import make_circles
X, y = make_circles(100, factor=.1, noise=.1)
clf = SVC(kernel='linear').fit(X, y)
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(clf, plot_support=False);
#高维空间下数据的可视化
from mpl_toolkits import mplot3d
r=np.exp(-(X**2).sum(1))
def plot_3D(elev=30, azim=30, X=X, y=y):
ax = plt.subplot(projection='3d')
ax.scatter3D(X[:, 0], X[:, 1], r, c=y, s=50, cmap='autumn')
ax.view_init(elev=elev, azim=azim)
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.set_zlabel('z')
plot_3D(elev=45,azim=45,X=X,y=y)
# 引入径向基函数
clf = SVC(kernel='rbf', C=1E6)
clf.fit(X, y)
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(clf)
plt.scatter(clf.support_vectors_[:, 0], clf.support_vectors_[:, 1],s=300, lw=1, facecolors='none');
#调C参数和gamma参数
#C参数
X, y = make_blobs(n_samples=100, centers=2,random_state=0, cluster_std=0.8)
fig, ax = plt.subplots(1, 2, figsize=(16, 6))
fig.subplots_adjust(left=0.0625, right=0.95, wspace=0.1)
for axi, C in zip(ax, [20, 0.2]):
model = SVC(kernel='linear', C=C).fit(X, y)
axi.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(model, axi)
axi.scatter(model.support_vectors_[:, 0],model.support_vectors_[:, 1],s=300, lw=1, facecolors='none');
axi.set_title('C = {0:.1f}'.format(C), size=14)
#gamma参数
X, y = make_blobs(n_samples=100, centers=2,random_state=0, cluster_std=1.1)
fig, ax = plt.subplots(1, 2, figsize=(16, 6))
fig.subplots_adjust(left=0.0625, right=0.95, wspace=0.1)
for axi, gamma in zip(ax, [20, 0.1]):
model = SVC(kernel='rbf', gamma=gamma).fit(X, y)
axi.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='autumn')
plot_svc_decision_function(model, axi)
axi.scatter(model.support_vectors_[:, 0],model.support_vectors_[:, 1],s=300, lw=1, facecolors='none');
axi.set_title('gamma = {0:.1f}'.format(gamma), size=14)