基于卷积神经网络的X光片检测患者肺炎
基于卷积神经网络的X光片预测患者肺炎情况
[!] 该任务是本人在大一下学期初完成,专业知识尚未成熟
1 任务目标
-
了解肺脏与肺部疾病类型
-
X射线检测和计算机断层扫描的原理
-
了解COVID-19疾病
-
掌握卷积神经网络概念及基础结构
-
掌握卷积神经网络在X光片检测患者肺炎系统的应用
-
使用卷积神经网络搭建X光片检测患者肺炎系统
2 任务描述
2.1 肺脏与肺部疾病
2.1.1 肺脏
- 呼吸系统由呼吸道(鼻、咽、喉、气管和各级支气管)和肺泡组成。肺脏是呼吸系统的主要器官,肺部疾病属于呼吸系统疾病。人体为了完成新陈代谢需要不断从空气中摄取氧气和排出二氧化碳(气体交换),这种气体交换称呼吸。肺与外界环境的气体交换和肺换气──肺泡与血液之间的气体交换称外呼吸(亦叫肺呼吸),气体经过血液运输到达组织后血液与组织细胞或组织液之间的气体交换称内呼吸(亦叫组织呼吸)。故肺脏与心血管系统有着密切的联系。肺脏除了主管呼吸功能外还具备非呼吸性的防御、免疫及内分泌代谢功能。
2.1.1 肺部疾病定义
- 肺部疾病是肺脏本身的疾病或是全身性疾病的肺部表现。
2.1.2 常见疾病类型
- 感染性肺部疾病
- 与大气污染和吸烟有关的肺部疾病
- 与职业有关的肺部疾病
- 与免疫有关的肺部疾病
- 与遗传有关的肺部疾病
- 肺部肿瘤
- 原因不明的肺部疾病
- 全身性疾病的肺部表现
2.1.3 鉴别诊断方法
- X射线检测和计算机断层扫描(CT)提供了对肺部阴影、肿块形态、性质的更明确的了解。纤维支气管镜检查,必要时还可经皮肤穿刺肺活检组织进行病理、细菌、生化的检验,大大提高了病因学的确诊率。支气管肺泡灌洗液可在镜下进行细胞计数、分类和测定液体内免疫抗体、补体与酶类,以帮助对疾病性质的了解。利用放射性核素Ga(镓)进行肺扫描:Ga可以集聚在新陈代谢活跃的部位,临床上用以诊断肺癌、结节病、间质性肺泡炎等。并可用以鉴别肺梗塞与肺炎。肺动脉造影术有助于对肺梗塞的诊断。在本系统中,我们将辅助X线断层摄影和计算机断层扫描来对患者肺炎进行预测分析。
2.2 X射线检测和计算机断层扫描(CT)
注:以下可能含有引起不适的内容
2.2.1 X射线检测原理
- X射线检测主要依据X射线的穿透作用、差别吸收、感光作用和荧光作用。由于X射线穿过人体时,受到不同程度的吸收,如骨骼吸收的X射线量比肌肉吸收的量要多,那么通过人体后的X射线量就不一样,这样便携带了人体各部密度分布的信息,在荧光屏上或摄影胶片上引起的荧光作用或感光作用的强弱就有较大差别,因而在荧光屏上或摄影胶片上(经过显影、定影)将显示出不同密度的阴影。根据阴影浓淡的对比,结合临床表现、化验结果和病理诊断,即可判断人体某一部分是否正常。

2.2.2 计算机断层扫描(CT)原理
- 传统的X光检查最多只能产生一幅大脑阴影的图像,这样的影像分辨力不高。为了解决这个问题,研究者设计了计算机断层扫描(computedtomography,简称CT)。CT是以X射线从多个方向沿着头部某一选定断层层面进行照射,测定透过的X射线量,数字化后经过计算机算出该层面组织各个单位容积的吸收系数,然后重建图像的一种技术。这是一种图质好、诊断价值高而又无创伤、无痛苦、无危险的诊断方法。它使我们能够在任何深度或任何角度重建脑的各种层面结构。它具有较传统CT扫描范围大、图像质量好、成像速度快等优点。
2.3 COVID-19
2.3.1 简介
- CONVID-19是一种由严重急性呼吸系统综合征冠状病毒2(缩写:SARS-CoV-2)引发的传染病。该病首名公开出现于2019年末在中国大陆湖北省武汉市,其后此病在全球各国急速扩散,截至2020年4月28日已经波及226个国家和地区,感染超过300万人并已导致其中逾20万名病人病逝,被世卫组织认定为人类自二战以来最具毁灭性的瘟疫。
2.3.2 临床表现
- COVID-19的症状及严重程度因人而异,本疾病存在无症状感染者,有症状患者主要以轻症居多(约81%)。大多数患者的表现以类流感症状为主,常见临床表现包括发热、四肢乏力、干咳等症状,其他表现包含鼻塞、打喷嚏、流鼻水、头痛、咽痛、咳血,咳痰、肌痛,或腹泻等。有报导显示轻症初期症状可能以嗅觉及味觉丧失表现,部分轻症患者无肺炎表现,仅表现为低热、轻微乏力等。严重症状包括呼吸困难、持续性胸痛、意识混乱、步行困难或面唇发黑。

-
严重并发症包含急性呼吸窘迫综合征(ARDS)、脓毒症休克、全身炎症反应综合征、难以纠正的代谢性酸中毒、急性心肌损伤、凝血功能障碍,甚至死亡等。
-
疾病潜伏期通常约在暴露后4-5天左右,一般认为不会超过14天。97.5%的患者会在感染后11.5天内出现症状。目前认为无症状患者也具有传播疾病的能力。
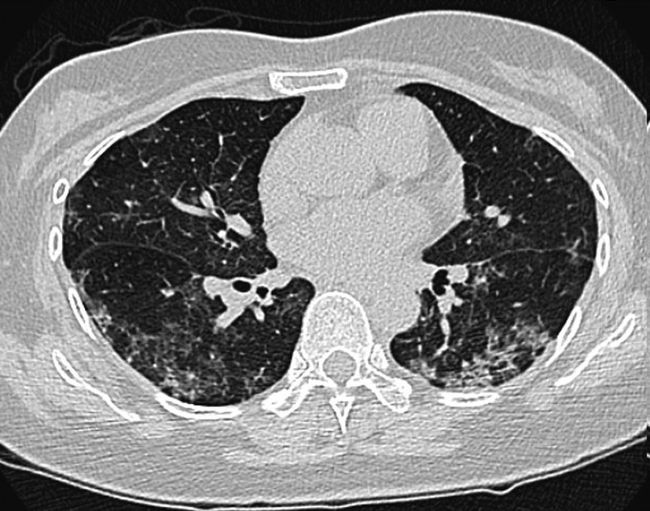
2.3.3 影像检测
- 早期患者的肺部会呈现多发小斑片影及间质改变,以肺外带明显。经发展后,肺炎患者被观察到双肺多发毛玻璃状病变、浸润影。严重者则会进一步发展为次节叶或大叶性肺实变影像表现,胸腔积液少见。有一部分的病人,可能在聚合酶连锁反应测试(RT-PCR)阴性的情形下,在电脑断层上却出现早期典型的肺实变。

3 卷积神经网络
3.1 简述
- 卷积神经网络(Convolutional Neural Network, CNN)是一种前馈神经网络,它的人工神经元可以响应一部分覆盖范围内的周围单元,对于大型图像处理有出色表现。
3.2 结构
卷积神经网络由一个或多个卷积层和顶端的全连通层(对应经典的神经网络)组成,同时也包括关联权重和池化层(pooling layer)
3.2.1 卷积层
- 卷积层是一组平行的特征图,它由多个卷积核构成,能够让卷积核在输入图像上滑动并进行一定的运算从而得到一个值并投影到输出特征图中。
3.2.2 ReLU层
-
ReLU即线性整流层 f ( x ) = m a x ( 0 , x ) f(x)=max{(0,x)} f(x)=max(0,x),与Sigmoid等激活函数相比,ReLU能够有效地避免梯度断裂情况。同时,ReLU激活函数模拟了生物学中的神经元兴奋行为,能够将影响因素放大,提高神经网络的训练速度
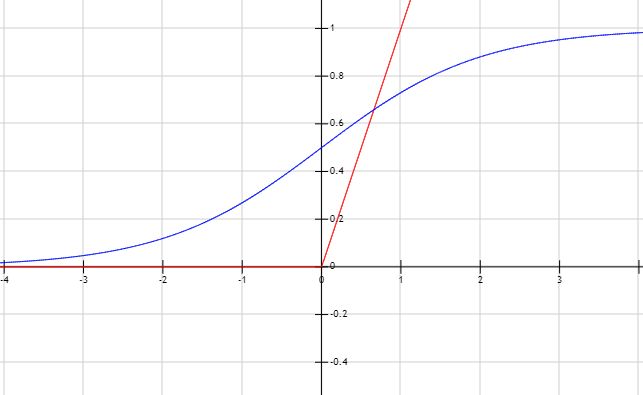
图中红色线为ReLU激活函数 f ( x ) = m a x ( 0 , x ) f(x)=max(0,x) f(x)=max(0,x),蓝色线为Sigmoid激活函数 f ( x ) = 1 1 + e − x f(x)=\frac{1}{1+e^{-x}} f(x)=1+e−x1
3.2.3 池化层
- 池化是一种非线性形式的降采样,一般的卷积神经网络都会采用“最大池化”进行操作,即将输入图像划分为若干个矩形区域,对每个子区域输出最大值。
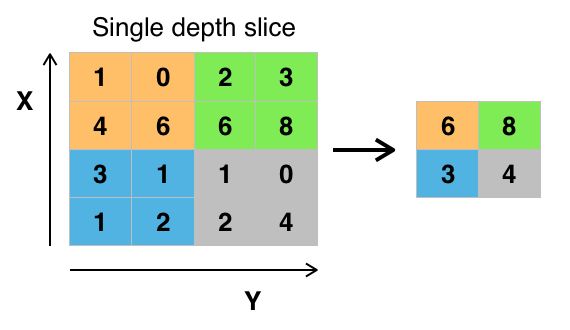
3.2.4 全连接层
- 在经过多轮卷积-池化操作与平铺操作(flatten)后,卷积神经网络将会得到一个一维张量进行处理。全连接层则是卷积神经网络中用于进行高级推理过程的结构,如同多层感知机一样运作,即进行仿射变换操作。
3.2.5 损失函数层
- 损失函数层一般用于计算模型在训练过程中的好坏度,并将该结果反馈于模型使其能进行调整。在本系统中,我们将会采用稀疏张量交叉熵损失函数来表述网络的预测结果核真实结果之间的差异。
4 任务实现
X光片检测患者肺炎情况本质为经典的图像多分类任务,基于任务描述及知识储备,我们以蒙特利尔大学博士后 Joseph Cohen 所整理的X光片图像数据集作为病患数据源,以及来源于Kaggle的胸部健康X射线图像数据集作为健康数据源
4.1 实现思路
- 预处理数据集
- 构建卷积神经网络模型
- 对卷积神经网络进行训练
- 测试卷积神经网络模型
本次任务所需模块如下:
import tensorflow as tf
from tensorflow.keras import Sequential,layers
import matplotlib.pyplot as plt
import matplotlib.image as mpimg
import numpy as np
import pandas as pd
import glob,os,random,pathlib
4.2 具体实现
4.2.1 读取数据
数据集由两个数据集组成,且Kaggle胸部健康X射线图像数据集具有少许噪声,以下为两个数据集内容对比。

系统的文件储存路径为

#加载数据集1
data1 = pd.read_csv('./data/metadata.csv',encoding='gb18030')
#查看第一张图片
temp = mpimg.imread('./data/images/' +data1.filename[0])
plt.imshow(temp)
plt.show()
4.2.2 整理路径与标签
通过Python字典来将标签转换为索引
#finding和filename为csv文件中的标签
labelToIndex = dict((name,number) for number,name in enumerate(list(set(data1.finding))))
"""
{'Chlamydophila': 0,
'Pneumocystis': 1,
'Streptococcus': 2,
'No Finding': 3,
'Klebsiella': 4,
'SARS': 5,
'COVID-19': 6,
'COVID-19, ARDS': 7,
'ARDS': 8,
'E.Coli': 9,
'Legionella': 10}
"""
随后使用Python两个平行列表来分别储存图像的位置和对应的标签
#打包路径和标签
dataPaths1 = ['./data/images/' + path for path in data1.filename]
dataLabels1 = [labeltoIndex[name] for name in data1.finding]
4.2.3 处理健康数据集
根据csv中可知前1300个样本皆为健康样本,故可以按顺序抽取其中若干份进行处理,且能看出因为Joseph Cohen并不包含健康情况,因此需要额外增加标签
#加载数据集2
data2 = pd.read_csv('./data/seMetadata.csv',encoding='gb18030')
#打包路径和标签
SIZE = 150
SIZE = min(1300,SIZE)
unormalType = len(list(set(data1.finding))) #不正常的种类
labelToIndex['Normal'] = UNORMAL #增加正常数据集
dataPaths2 = ['./data/seImages/' + path for path in data2.X_ray_image_name[:SIZE]]
dataLabels2 = [unormalType] * SIZE
4.2.4 整合及打乱数据集
随后,只需要把两组一一对应的列表相加即可得到完整的源数据集
#整合两个数据集
dataPaths = dataPaths1 + dataPaths2
dataLabels = dataLabels1 + dataLabels2
由于两份数据集接按顺序进行排列,若不进行打乱处理,会造成严重的过拟合现象
#打乱数据集
seed = random.randint(1,100)
random.seed(seed)
random.shuffle(dataPaths)
random.seed(seed)
random.shuffle(dataLabels)
#抽取前10个查看效果
print(dataPaths[:10],dataLabels[:10])
输出如下:
['./data/images/FE9F9A5D-2830-46F9-851B-1FF4534959BE.jpeg',
'./data/images/1.CXRCTThoraximagesofCOVID-19fromSingapore.pdf-002-fig3a.png',
'./data/images/covid-19-pneumonia-20.jpg',
'./data/images/covid-19-pneumonia-14-PA.png',
'./data/seImages/IM-0216-0001.jpeg',
'./data/images/streptococcus-pneumoniae-pneumonia-temporal-evolution-1-day2.jpg',
'./data/images/A7E260CE-8A00-4C5F-A7F5-27336527A981.jpeg',
'./data/seImages/IM-0207-0001.jpeg',
'./data/seImages/IM-0137-0001.jpeg',
'./data/seImages/IM-0154-0001.jpeg']
[6, 6, 6, 6, 11, 2, 6, 11, 11, 11]
经检查后确认无误
4.2.5 创建Dataset类
在Tensorflow中支持用户将数据整合为Dataset,并且可以直接使用Dataset类在keras中进行训练
#将数据集按照3:1:1的比例进行分类
SPILT = 0.6
trainData = tf.data.Dataset.from_tensor_slices((dataPaths[:int(SPILT*len(dataLabels))],dataLabels[:int(SPILT*len(dataLabels))]))
testData = tf.data.Dataset.from_tensor_slices((dataPaths[int(SPILT*len(dataLabels)):],dataLabels[int(SPILT*len(dataLabels)):]))
随后构建函数对图像进行预处理
def prePicPNG(path,label):
"""预处理图像"""
temp = tf.io.read_file(path)
temp = tf.cond(tf.image.is_jpeg(temp),
lambda: tf.image.decode_jpeg(temp, channels=3),
lambda: tf.image.decode_png(temp, channels=3))
temp = tf.cast(temp,tf.float32)
temp /= 255.0
temp = tf.image.resize(temp,[192,192])
return temp,label
使用map()遍历Dataset即可,同时对数据集进行打乱和打包
SHUFFLE = 1000
BATCH = 4
trainData = trainData.map(prePicPNG).shuffle(SHUFFLE)
trainData = trainData.batch(BATCH)
testData = testData.map(prePicPNG).shuffle(SHUFFLE).batch(1)
vaildData = vaildData.map(prePicPNG).shuffle(SHUFFLE).batch(1)
至此,数据已经准备完毕,可以进入到使用阶段
4.2.6 构建CNN模型
鉴于任务较为简单,因此使用模仿LeNet-5的经典网络模型来预测
#构建模型
model = Sequential()
model.add(layers.Conv2D(32,(5,5),padding = 'SAME',activation='relu',input_shape=[192,192,3]))
model.add(layers.MaxPooling2D((2,2)))
model.add(layers.Conv2D(64,(3,32),padding = 'SAME',activation='relu'))
model.add(layers.MaxPooling2D((2,2)))
model.add(layers.Flatten())
model.add(layers.Dense(12,activation='softmax'))
model.compile(optimizer='adam',
loss='sparse_categorical_crossentropy',
metrics=['accuracy'])
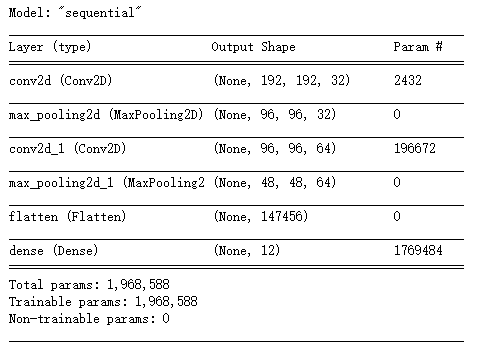
model.summary()
输出结果为:
4.2.7 训练模型
将训练集和测试集载入到模型中,配置好基本的超参数即可开始训练
#训练模型并保存历史记录
history = model.fit(trainData,shuffle=True,epochs=10,validation_data=testData)

至此已经完成了模型的训练
4.2.8 可视化数据及验证集
使用matplotlib和vaildData来完成分析
#将数据展示
plt.plot(history.history['accuracy'],label = 'accuracy')
plt.plot(history.history['val_accuracy'],label = 'val_accuracy')
plt.plot(history.history['loss'],label = 'loss')
plt.plot(history.history['val_loss'],label = 'val_loss')
plt.legend(loc='best')
plt.show()

#预测验证集
model.predict(vaildData)

能够看出虽然有83.48%的准确率,损失值也有失控的趋势
4.3 实例测试
4.3.1 处理图片
为了能够让卷积神经网络预测图片,因此需要对图片进行预处理
def editInput(path):
"""解码处理图片"""
temp = tf.io.read_file(path)
temp = tf.image.decode_jpeg(temp,channels = 3) #RGB图对应3个特征图
temp = tf.image.resize(temp,[192,192])
return tf.reshape(temp,(1,192,192,3))
4.3.2 功能封装
将功能封装起来,方便后续的使用
#索引-标签列表
indexName = [name for name,val in labelToIndex.items()]
def predictPic(path):
"""预测一个X光片的病例"""
test = editInput(path)
ans = model.predict(test).tolist()[0]
ans = ans.index(max(ans))
return indexName[ans]
5 数据分析与优化
5.1 数据分析
5.1.1 主要问题
- 过拟合:在训练中可以看到,模型在第3次训练后便出现了过拟合的现象
- 训练效果不佳:在十次训练中,测试集错误率在持续上升
5.1.2 改进思路
- 调整网络结构
- 使用回调函数储存最佳模型
- 增加数据集
5.2 优化实现
5.2.1 调整网络结构
对网络结构做了略微的修改,增加了Inception模块以及Dropout层
def Inception(x,n=1):
"""Inception模块"""
Inception1 = layers.Conv2D(n * 2,(1,1),padding='SAME',activation = 'relu')(x)
Inception1 = layers.Conv2D(n * 8,(1,3),padding='SAME',activation = 'relu')(Inception1)
Inception1 = layers.Conv2D(n * 8,(3,1),padding='SAME',activation = 'relu')(Inception1)
Inception1 = layers.Conv2D(n * 8,(1,3),padding='SAME',activation = 'relu')(Inception1)
Inception1 = layers.Conv2D(n * 8,(3,1),padding='SAME',activation = 'relu')(Inception1)
Inception2 = layers.Conv2D(n * 16,(1,1),padding='SAME',activation = 'relu')(x)
Inception3 = layers.Conv2D(n * 4,(1,1),padding='SAME',activation = 'relu')(x)
Inception3 = layers.Conv2D(n * 32,(1,3),padding='SAME',activation = 'relu')(Inception3)
Inception3 = layers.Conv2D(n * 32,(3,1),padding='SAME',activation = 'relu')(Inception3)
Inception4 = layers.MaxPooling2D((3,3),strides=(1, 1),padding = 'SAME')(x)
Inception4 = layers.Conv2D(n * 4,(1,1),padding='SAME',activation = 'relu')(Inception4)
return layers.Concatenate()([Inception1,Inception2,Inception3,Inception4])
整体结构代码如下:
inputs = layers.Input(shape=(192,192,3))
x = Inception(x)
x = layers.MaxPooling2D((2,2),strides=(2,2),padding = 'SAME')(x)
x = Inception(x,2)
x = layers.MaxPooling2D((2,2),strides=(2,2),padding = 'SAME')(x)
x = Inception(x,2)
x = layers.MaxPooling2D((2,2),strides=(2,2),padding = 'SAME')(x)
x = Inception(x,3)
x = layers.MaxPooling2D((2,2),strides=(2,2),padding = 'SAME')(x)
x = Inception(x,3)
x = layers.MaxPooling2D((2,2),strides=(2,2),padding = 'SAME')(x)
x = Inception(x,4)
x = layers.MaxPooling2D((2,2),strides=(2,2),padding = 'SAME')(x)
x = layers.Flatten()(x)
x = layers.Dense(1024,activation = 'relu')(x)
x = layers.Dense(512,activation = 'relu')(x)
x = tf.keras.layers.Dropout(0.5)(x)
outputs = layers.Dense(12,activation='softmax')(x)
model = Model(inputs=inputs,outputs=outputs)
训练效果如下:

能够看出有效缓解了过拟合情况,准确率维持在85%附近,并且测试集与测试集准确率和损失值保持在相同水平,预计为样本数目不足导致的准确度无法进一步提高
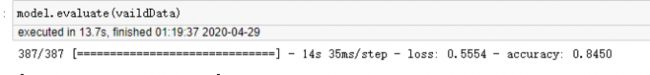
在验证集中能看出损失值降到了一个相对较低的水平,准确率也与预期相同
5.2.2 回调函数
使用Callbacks中对验证集损失值进行监视,并且回收损失值最小的模型
callback = tf.keras.callbacks.EarlyStopping(monitor='val_loss', patience=3,restore_best_weights=True)
history = model.fit(trainData,shuffle=True,epochs=12,validation_data=testData,steps_per_epoch=120,callbacks=[callback])
5.2.3 扩大数据集
能够看出数据集中样本数非常少,因此可以调整Kaggle中加入的健康数据集数目,来增加数据集的数目
训练效果如下:

能够看出准确率提高并稳定在90%左右,loss值进一步下降
使用验证集进行验证:
能够看出与分析内容大抵相同