Kmeans&HCA + iris数据集+python实现
基本的聚类分析算法
-
K均值 (K-means):
基于原型的、划分的距离技术,它试图发现用户指定个数(K)的簇。
a. 随机选取k个中心点
b. 遍历所有数据,将每个数据划分到最近的中心点中
c. 计算每个聚类的平均值,并作为新的中心点
d. 重复2-3,直到这k个中线点不再变化(收敛了),或执行了足够多的迭代
时间复杂度:O(Inkm)
空间复杂度:O(nm) -
层次凝聚聚类算法(HCA - Hierarchical Agglomerative Clustering):
主要思想就是,先把每一个样本点当做一个聚类,然后不断重复的将其中最近的两个聚类合并(就是凝聚的含义),直到满足迭代终止条件。
a. 将训练样本集中的每个数据点都当做一个聚类;
b. 计算每两个聚类之间的距离,将距离最近的或最相似的两个聚类进行合并;
c. 重复上述步骤,直到得到的当前聚类数是合并前聚类数的10%,即90%的聚类都被合并了;当然还可以设置其他终止条件,这样设置是为了防止过度合并。 -
DBSCAN:
一种基于密度的划分距离的算法,簇的个数有算法自动的确定,低密度中的点被视为噪声而忽略,因此其不产生完全聚类。
自己尝试过。。。好像对iris数据集不太好用,有空再研究
https://blog.csdn.net/weixin_43909872/article/details/85342540
用python+sklearn+iris数据集去实验一下:
- K均值 (K-means)
data = pd.read_csv("iris.csv")
data = np.mat(data)
y_pred = KMeans(n_clusters=3).fit(data[:, 1:5])
colors = 'gbycm'
y_pred_color = []
category = []
for pred in y_pred.labels_:
if pred == -1:
color = 'r'
else:
color = colors[pred]
y_pred_color.append(color)
for type in data[:, 5]:
if type == 'setosa':
category.append(0)
elif type == 'versicolor':
category.append(1)
elif type == 'virginica':
category.append(2)
plt.scatter(data[:, 0].tolist(), category, c=y_pred_color)
plt.show()
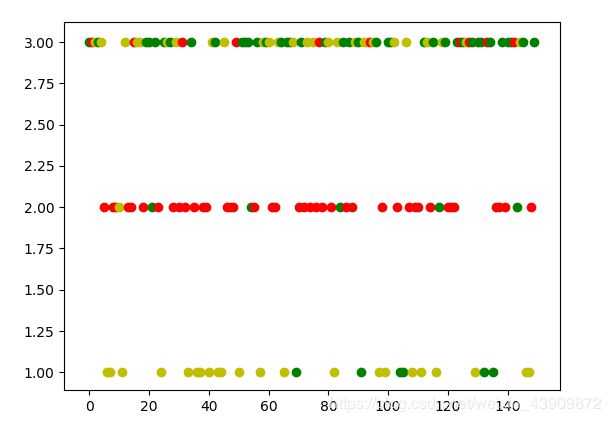
实验结果:
蛮好的分成了三类(绿蓝黄),第二类和第三类里面有些点和iris数据集里的点不一样,但感觉比DBScan的输出好多了
- 层次凝聚聚类算法(HCA - Hierarchical Agglomerative Clustering):
a. 导入iris数据集并计算每两条数据间的欧式距离:
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
from scipy.cluster.hierarchy import dendrogram, linkage, fcluster, maxinconsts
def euclideanDistance(trainingInstance, testInstance):
distance = 0
for x in range(len(trainingInstance)):
distance += np.square(trainingInstance[0,x] - testInstance[0,x])
return np.sqrt(distance);
def calculateDistances(data):
distances = []
for x in range(len(data)):
distancesForRow = []
for y in range(len(data)):
dist = euclideanDistance(data[x, :], data[y, :])
distancesForRow.append(dist)
distances.append(distancesForRow)
return distances
data = pd.read_csv("iris.csv")
data = np.mat(data)
np.random.shuffle(data)
distances = calculateDistances(data[:, 1:5])
b. 进行聚类分析
先一直计算到最后一个簇,这里用了scipy.cluster.hierarchy的linkage函数,计算每一步的聚类
debug得到下面的数据,每一行代表了一次聚类,比如第一行,是把第0个和第3个数据合起来了,合成后的这个簇里有2个数据点
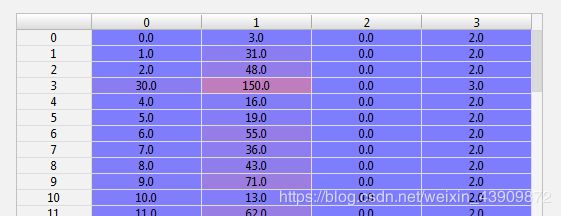
然后用fcluster函数去得到我们想要的聚类,这里我们用了maxclust的方式,也就是聚类到最后剩下三个簇,用以和原始的iris数据做对比
fcluster(Z, maxCluster, criterion = ‘maxclust’)
def HCA(data, method='average', maxCluster = 5):
'''HCA
Arguments:
data [[0, float, ...], [float, 0, ...]] -- distances between each document
Keyword Arguments:
method {str} -- [linkage method: single、complete、average、centroid、median、ward] (default: {'average'})
threshold {float} -- the cluster No. to stop
Return:
maxCluster int -- max cluster No.
cluster [[idx1, idx2,..], [idx3]] -- the index of each cluster
'''
data = np.array(data)
Z = linkage(data, method=method)
# assignments = fcluster(Z, threshold, criterion='distance')
assignments = fcluster(Z, maxCluster, criterion = 'maxclust')
clusterNo = assignments.max()
indices = getClasterIndices(assignments)
return clusterNo, indices
def getClasterIndices(assignments):
n = assignments.max()
indices = []
for cluster_number in range(1, n + 1):
indices.append(np.where(assignments == cluster_number)[0])
return indices
clustersNo, indices = HCA(distances, maxCluster = 3)
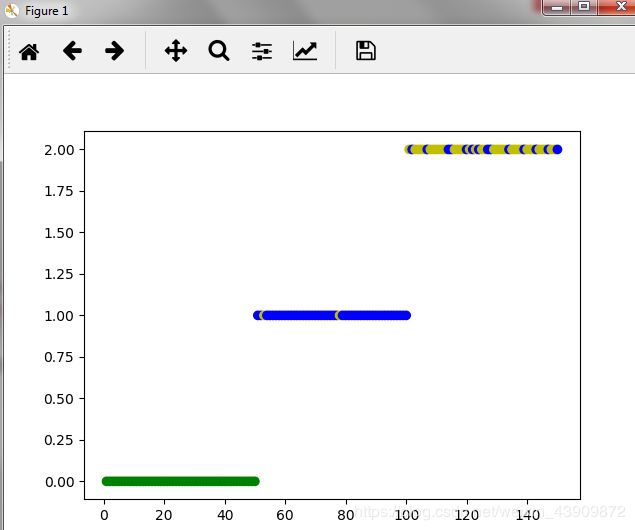
c.显示聚类结果并且和iris数据集原有数据对比
Y坐标代表用HCA分类的结果,颜色代表原来iris数据集里的种类
可以看到效果还可以
type = 0
color = 'b'
for indice in indices:
type += 1
for nodeIndex in indice:
if data[nodeIndex, 5] == 'setosa':
color = 'r'
elif data[nodeIndex, 5] == 'versicolor':
color = 'g'
elif data[nodeIndex, 5] == 'virginica':
color = 'y'
plt.scatter(nodeIndex, type, c = color)
plt.show()