机器学习sklearn(15)TSNE降维
TSNE概念部分参考:http://bindog.github.io/blog/2016/06/04/from-sne-to-tsne-to-largevis/#0x02-%E4%BB%8Esne%E8%AF%B4%E8%B5%B7
sklearn.manifold.TSNE(n_components=2, perplexity=30.0, early_exaggeration=12.0, learning_rate=200.0, n_iter=1000, n_iter_without_progress=300, min_grad_norm=1e-07, metric=’euclidean’, init=’random’, verbose=0, random_state=None, method=’barnes_hut’, angle=0.5)
参数:
n_components:int,可选(默认值:2)嵌入式空间的维度。
perplexity:浮点型,可选(默认:30)较大的数据集通常需要更大的perplexity。考虑选择一个介于5和50之间的值。由于t-SNE对这个参数非常不敏感,所以选择并不是非常重要。
early_exaggeration:float,可选(默认值:4.0)这个参数的选择不是非常重要。
learning_rate:float,可选(默认值:1000)学习率可以是一个关键参数。它应该在100到1000之间。如果在初始优化期间成本函数增加,则早期夸大因子或学习率可能太高。如果成本函数陷入局部最小的最小值,则学习速率有时会有所帮助。
n_iter:int,可选(默认值:1000)优化的最大迭代次数。至少应该200。
n_iter_without_progress:int,可选(默认值:300,必须是50倍数)在我们中止优化之前,没有进展的最大迭代次数。
0.17新版功能:参数n_iter_without_progress控制停止条件。
min_grad_norm:float,可选(默认值:1E-7)如果梯度范数低于此阈值,则优化将被中止。
metric:字符串或可迭代的,可选,计算特征数组中实例之间的距离时使用的度量。如果度量标准是字符串,则它必须是scipy.spatial.distance.pdist为其度量标准参数所允许的选项之一,或者是成对列出的度量标准.PAIRWISE_DISTANCE_FUNCTIONS。如果度量是“预先计算的”,则X被假定为距离矩阵。或者,如果度量标准是可调用函数,则会在每对实例(行)上调用它,并记录结果值。可调用应该从X中获取两个数组作为输入,并返回一个表示它们之间距离的值。默认值是“euclidean”,它被解释为欧氏距离的平方。
init:字符串,可选(默认值:“random”)嵌入的初始化。可能的选项是“随机”和“pca”。 PCA初始化不能用于预先计算的距离,并且通常比随机初始化更全局稳定。
random_state:int或RandomState实例或None(默认)
伪随机数发生器种子控制。如果没有,请使用numpy.random单例。请注意,不同的初始化可能会导致成本函数的不同局部最小值。
method:字符串(默认:'barnes_hut')
默认情况下,梯度计算算法使用在O(NlogN)时间内运行的Barnes-Hut近似值。 method ='exact'将运行在O(N ^ 2)时间内较慢但精确的算法上。当最近邻的误差需要好于3%时,应该使用精确的算法。但是,确切的方法无法扩展到数百万个示例。0.17新版功能:通过Barnes-Hut近似优化方法。
angle:float(默认值:0.5)
仅当method ='barnes_hut'时才使用这是Barnes-Hut T-SNE的速度和准确性之间的折衷。 'angle'是从一个点测量的远端节点的角度大小(在[3]中称为theta)。如果此大小低于'角度',则将其用作其中包含的所有点的汇总节点。该方法对0.2-0.8范围内该参数的变化不太敏感。小于0.2的角度会迅速增加计算时间和角度,因此0.8会快速增加误差。
| attributes | description |
|---|---|
| embedding_ | 嵌入向量 |
| kl_divergence | 最后的 KL 散度 |
| n_iter_ | 迭代的次数 |
| Methods | description |
|---|---|
| fit | 将 X 投影到一个嵌入空间 |
| fit_transform | 将 X 投影到一个嵌入空间并返回转换结果 |
| get_params | 获取 t-SNE 的参数 |
| set_params | 设置 t-SNE 的参数 |
from sklearn.manifold import TSNE
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
iris = load_iris()
iris_data = iris.data
iris_target = iris.target
from sklearn.preprocessing import StandardScaler
sc = StandardScaler()
data_std = sc.fit_transform(iris_data)
tsne = TSNE(n_components=2, learning_rate=100)
tsne.fit_transform(data_std)
array([[-22.37778 , -14.470559 ],
[-19.074526 , -12.40457 ],
[-19.71702 , -13.648663 ],
[ 10.526178 , 6.301651 ],
[ 12.626265 , 4.926107 ],
[ 6.4893355 , 8.0801115 ]], dtype=float32)
import numpy as np
data = np.array(tsne.embedding_)
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
data_pca = pca.fit_transform(data_std)
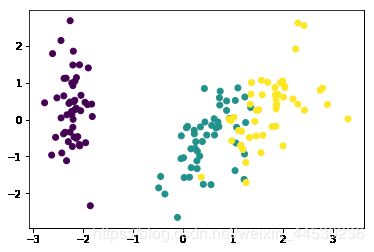
plt.scatter(data_pca[:,0], data_pca[:,1], c=iris_target)
plt.show()