Stanford机器学习课程笔记4-Kmeans与高斯混合模型
这一部分属于无监督学习的内容,无监督学习内容主要包括:Kmeans聚类算法、高斯混合模型及EM算法、Factor Analysis、PCA、ICA等。本文是Kmeans聚类算法、高斯混合模型的笔记,EM算法是适用于存在latent/hidden变量的通用算法,高斯混合模型仅仅是EM算法的一种特殊情况,关于EM算法的推到参见Andrew Ng讲义。由于公式太多,最近时间又忙实习的事就简单写一些,回头看时还得参考Ng的笔记和自己的打印Notes上的笔记,这里的程序对理解可能能提供另外的一些帮助。
Kmeans聚类
其实聚类算法除了Kmeans,还有其它的聚类方式,记得曾经接触到的就有层次聚类、基于模糊等价关系的模糊聚类。Kmeans只不过是这些聚类算法中最为常见的一中,也是我用得最多的一种。Kmeans本身的复杂度也高(O(N^3)),因此Kmeans有很多其它的变种:
- 针对浮点型数据的运算,为提高运算效率,将浮点型Round成整形(我操作的话一般先乘一个系数,以使数据分布在整个整数空间,降低Round的数据损失)
- 层次化的Kmeans聚类(HIKM)
这两种改进都能在VLFeat里见到C代码实现。作为完善基础知识,我还是愿意先来复习下最基本的Kmeans聚类,后面再根据自己的经验补充点HIKM的东西。这是Stanford机器学习课程中第一个非监督学习算法,为严谨一些,先给出一般Kmeans聚类问题的描述:
- 给定数据集:
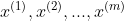
- 将数据group成K个clusters
请注意:相对于监督学习算法,Kmeans虽然没有label信息,但聚类数K却是已知的,也就是说:使用Kmeans我们得计划好要聚成多少个cluster。当然也有一些Kmeans基础上的改进方法可以通过阈值直接判断聚类数目,这里不讨论。Kmeans算法的流程是:
先随机初始化K个聚类中心,计算样本数据到聚类中心的距离,用聚类结果计算新的中心,如此迭代。提出两个问题:
- 随机初始化聚类中心怎么个随机法:这K个点是数据点还是非数据点。其实除了一般的随机,还有一种叫K-means++(参考2)的初始化方式
凡是迭代算法都有一个问题值得讨论:收敛准则。白话描述Kmeans收敛准则就是:聚类中心u稳定,不再发生太大的变化。严谨一些,定义Distortion Function:
J表示每个训练样本到被分配到的聚类中心的距离之和。当J最小时则Kmeans算法收敛。然而,由于J是非凸函数,利用坐标下降算法无法保证能收敛到全局最优解(坐标下降算法在Andrew Ng在SVM讲义部分中有讨论,是梯度下降算法的多维情况:先固定A变量,B变量用梯度下降迭代;再固定B变量,A变量用梯度下降迭代)。然而,在实际应用中,对譬如(1)存在多个局部最有之间波动的情况很少见(2)即使收敛到局部最有Kmeans算法,对于实际问题这个局部最优解也一般能够接受。所以Kmeans依然是使用最广泛的无监督聚类算法。
Matlab的Statistics and Machine Learning Toolbox自带kmeans算法,更多内容可以参考一下Matlab中的doc kmeans或者http://cn.mathworks.com/help/stats/kmeans.html 。
混合高斯模型(Mixture of Gaussian)
尖子班和普通班的成绩混在一起了,要把这两个班的成绩分开。只知道2个班的成绩都满足高斯分布,但不知道每个班的高斯分布的平均分和方差波动。如何通过无监督聚类的方法将这两个班的数据分开?上面的例子就是高斯混合模型的一个例子。
回想GDA(高斯判别分析)的生成模型,当时建立的模型是:p(y)服从伯努利实验,p(x|y)服从高斯分布,即隐藏在数据背后的高斯模型差生了训练数据,通过极大似然法使联合概率p(x,y)最大,求解得到伯努利参数phi,高斯分布参数u,sigma。混合高斯模型由于是无监督的数据,没有y,所以建模时假定一个latent/hidden(隐藏)变量z(等价GDA中的y),利用GDA的完全一样的极大似然法求解phi(z)、u(z)、sigma(z),但此时的pht、u、sigma都是和latent变量z有关的不确定的数。这就需要一种迭代算法:先估计z,更确切的说,这里是要来估计z的概率;再用z的估计值计算phi、u和sigma;更新z的估计,再迭代。。。。
高斯混合模型是EM算法的一个特例,迭代算法分为E-Step(估计z的概率)和M-Step(似然函数最大化),
在E-Step中,估计的是z的后验概率,可以先通过初始化的phi、u、sigma计算似然概率和先验概率,再用Bayes Rule得到z的后验估计。EM算法与Kmeans算法一样可能收敛到局部最优,有点不同的是EM算法的聚类中心数是可以自动决定的而Kmeans是预先给定的。下面是从 http://www.mathworks.com/matlabcentral/fileexchange/26184-em-algorithm-for-gaussian-mixture-model 找到的一份高斯混合模型的EM代码,也可以下载完整的EM Example在Matlab上运行
function [label, model, llh] = emgm(X, init)
% Perform EM algorithm for fitting the Gaussian mixture model.
% X: d x n data matrix
% init: k (1 x 1) or label (1 x n, 1<=label(i)<=k) or center (d x k)
% Written by Michael Chen ([email protected]).
%% initialization
fprintf('EM for Gaussian mixture: running ... \n');
R = initialization(X,init);
[~,label(1,:)] = max(R,[],2);
R = R(:,unique(label));
tol = 1e-10;
maxiter = 500;
llh = -inf(1,maxiter);
converged = false;
t = 1;
while ~converged && t < maxiter
t = t+1;
model = maximization(X,R);
[R, llh(t)] = expectation(X,model);
[~,label(:)] = max(R,[],2);
u = unique(label); % non-empty components
if size(R,2) ~= size(u,2)
R = R(:,u); % remove empty components
else
converged = llh(t)-llh(t-1) < tol*abs(llh(t));
end
end
llh = llh(2:t);
if converged
fprintf('Converged in %d steps.\n',t-1);
else
fprintf('Not converged in %d steps.\n',maxiter);
end
function R = initialization(X, init)
[d,n] = size(X);
if isstruct(init) % initialize with a model
R = expectation(X,init);
elseif length(init) == 1 % random initialization
k = init;
idx = randsample(n,k);
m = X(:,idx);
[~,label] = max(bsxfun(@minus,m'*X,dot(m,m,1)'/2),[],1);
[u,~,label] = unique(label);
while k ~= length(u)
idx = randsample(n,k);
m = X(:,idx);
[~,label] = max(bsxfun(@minus,m'*X,dot(m,m,1)'/2),[],1);
[u,~,label] = unique(label);
end
R = full(sparse(1:n,label,1,n,k,n));
elseif size(init,1) == 1 && size(init,2) == n % initialize with labels
label = init;
k = max(label);
R = full(sparse(1:n,label,1,n,k,n));
elseif size(init,1) == d %initialize with only centers
k = size(init,2);
m = init;
[~,label] = max(bsxfun(@minus,m'*X,dot(m,m,1)'/2),[],1);
R = full(sparse(1:n,label,1,n,k,n));
else
error('ERROR: init is not valid.');
end
function [R, llh] = expectation(X, model)
mu = model.mu;
Sigma = model.Sigma;
w = model.weight;
n = size(X,2);
k = size(mu,2);
logRho = zeros(n,k);
for i = 1:k
logRho(:,i) = loggausspdf(X,mu(:,i),Sigma(:,:,i));
end
logRho = bsxfun(@plus,logRho,log(w));
T = logsumexp(logRho,2);
llh = sum(T)/n; % loglikelihood
logR = bsxfun(@minus,logRho,T);
R = exp(logR);
function model = maximization(X, R)
[d,n] = size(X);
k = size(R,2);
nk = sum(R,1);
w = nk/n;
mu = bsxfun(@times, X*R, 1./nk);
Sigma = zeros(d,d,k);
sqrtR = sqrt(R);
for i = 1:k
Xo = bsxfun(@minus,X,mu(:,i));
Xo = bsxfun(@times,Xo,sqrtR(:,i)');
Sigma(:,:,i) = Xo*Xo'/nk(i);
Sigma(:,:,i) = Sigma(:,:,i)+eye(d)*(1e-6); % add a prior for numerical stability
end
model.mu = mu;
model.Sigma = Sigma;
model.weight = w;
function y = loggausspdf(X, mu, Sigma)
d = size(X,1);
X = bsxfun(@minus,X,mu);
[U,p]= chol(Sigma);
if p ~= 0
error('ERROR: Sigma is not PD.');
end
Q = U'\X;
q = dot(Q,Q,1); % quadratic term (M distance)
c = d*log(2*pi)+2*sum(log(diag(U))); % normalization constant
y = -(c+q)/2;参考
- Andrew Ng Lecture Notes.
- D. Arthur and S. Vassilvitskii. k-means++: The advantages of careful seeding. In Proc. ACM-SIAM Symp. on Discrete Algorithms, 2007.
