使用libsvm分类和预测详细说明(python)
Libsvm使用详细介绍
optimization finished, #iter= 162 //iter为迭代次数,
nu = 0.431029 //nu是你选择的核函数类型的参数
obj = -100.877288, rho =0.424462 // rho为判决函数的偏置项b
// obj为SVM文件转换为的二次规划求解得到的最小值
nSV = 132, nBSV = 107 // nSV为标准支持向量个数(0
//nBSV为边界上的支持向量个数(a[i]=c)
Total nSV = 132
//TotalnSV为支持向量总个数(对于两类来说,因为只有一个分类模型TotalnSV = nSV,但是对于多类,这个是各个分类模型的nSV之和)。
用法:
svmscale[-l lower] [-u upper] //将数据进行归一化处理
[-y y_lower y_upper]
[-s save_filename]
[-r restore_filename]filename
其中,[]中都是可选项:
-l:设定数据下限;lower:设定的数据下限值,缺省为-1
-u:设定数据上限;upper:设定的数据上限值,缺省为 1
-y:是否对目标值同时进行缩放;y_lower为下限值,y_upper为上限值;
-ssave_filename:表示将缩放的规则保存为文件save_filename;
-rrestore_filename:表示将按照已经存在的规则文件restore_filename进行缩放;
filename:待缩放的数据文件,文件格式按照libsvm格式。
首先打开cmd,进入libsvm>windows文件夹
默认情况下,只需要输入要缩放的文件名就可以了:比如(已经存在的文件为test.txt)
svm-scaletest.txt
这时,test.txt中的数据已经变成[-1,1]之间的数据了。但是,这样原来的数据就被覆盖了,为了让规划好的数据另存为其他的文件,我们用一个dos的重定向符 > 来另存为(假设为out.txt):
svm-scale test.txt > out.txt
运行后,我们就可以看到目录下多了一个out.txt文件,那就是规范后的数据。假如,我们想设定数据范围[0,1],并把规则保存为test.range文件:
svm-scale –l 0 –u 1 –s test.range test.txt > out.txt
这时,目录下又多了一个test.range文件,可以用记事本打开,下次就可以用
-r test.range来载入了。
grid.py //暴力试参
首先进入libsvm>tools文件夹,找到grid.py,打开源代码修改gnuplot_exe(需要另下载)的路径
打开cmd,进入libsvm>tools文件夹
输入 python grid.py test.txt
可选参数[-log2cbegin,end,step] [-log2g begin,end,step] [-v fold]
//用户自定义的参数 c和g 的范围 begin~end 以及步长 step,几折交叉验证
得到参数c和g的值以及交叉验证准确率
注意:text.txt里面的数据,每一行的的键必须从小到大依次排列,否则报错,libsvm其他方法处理的时候,则不需要
使用时调入模块
(1)svm_read_problem() : read the data from a LIBSVM-format file
(2)svm_problem():![]()
(3)svm_parameter():参数为字符串
![]()
其中的c和g参数根据之前调试的参数进行修改
svm_train的参数:
-s SVM的类型(svm_type)
0 -- C-SVC(默认)使用惩罚因子(Cost)的处理噪声的多分类器
1 -- nu-SVC(多分类器)按照错误样本比例处理噪声的多分类器
2 -- one-class SVM一类支持向量机,可参见"SVDD"的相关内容
3 -- epsilon-SVR(回归)epsilon支持向量回归
4 -- nu-SVR(回归)
-t 核函数类型(kernel_type)
0 -- linear(线性核):u'*v
1 -- polynomial(多项式核):(gamma*u'*v +coef0)^degree
2 -- radial basisfunction(RBF,径向基核/高斯核):exp(-gamma*|u-v|^2)
3 -- sigmoid(S型核):tanh(gamma*u'*v + coef0)
4 -- precomputed kernel(预计算核):
核矩阵存储在training_set_file中
下面是调整SVM或核函数中参数的选项:
-d 调整核函数的degree参数,默认为3
-g 调整核函数的gamma参数,默认为1/num_features
-r 调整核函数的coef0参数,默认为0
-c 调整C-SVC, epsilon-SVR 和 nu-SVR中的Cost参数,默认为1
-n 调整nu-SVC, one-class SVM 和 nu-SVR中的错误率nu参数,默认为0.5
-p 调整epsilon-SVR的loss function中的epsilon参数,默认0.1
-m 调整内缓冲区大小,以MB为单位,默认100
-e 调整终止判据,默认0.001
-wi调整C-SVC中第i个特征的Cost参数
调整算法功能的选项:
• -b是否估算正确概率,取值0 - 1,默认为0
• -h是否使用收缩启发式算法(shrinkingheuristics),取值0- 1,默认为0
• -v交叉校验
• -q静默模式
(4)svm_train()
svm_train有3个重载:
![]()
l model = svm_train(y, x [,'training_options'])
![]()
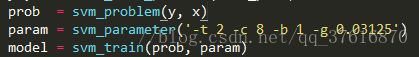
l model = svm_train(prob [,'training_options'])
l model = svm_train(prob, param)
(5)svm_save_model() : save model to a file.
将训练好的svm_model存储到文件中:
svm_save_model('model_file',model)
model_file的内容:
svm_typec_svckernel_typelinearnr_class 2 total_sv 2 rho 0 label 1 -1probA 0.693147 probB 2.3919e-16 nr_sv 1 1 SV 0.25 1:1 2:1-0.25 1:-1 2:-1
(6)svm_load_model() : load a LIBSVM model.
读取存储在文件中的svm_model:
model =svm_load_model('model_file')
(7)svm_predict()
调用语法:
p_labs, p_acc, p_vals =svm_predict(y, x, model [,'predicting_options'])
参数:
y测试数据的标签x测试数据的输入向量model为训练好的SVM模型。
返回值:
p_labs是存储预测标签的列表。
p_acc存储了预测的精确度,均值和回归的平方相关系数。
p_vals在指定参数'-b 1'时将返回判定系数(判定的可靠程度)。
这个函数不仅是测试用的接口,也是应用状态下进行分类的接口。比较奇葩的是需要输入测试标签y才能进行预测,因为y不影响预测结果可以用0向量代替。