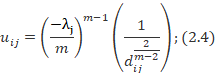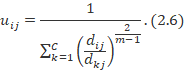Python--Kmeans和FCM聚类效果对比(对图像与数据处理)
1、Kmeans
Kmeans是聚类算法中较为经典的算法之一,由于其效率高,所以一般大规模的数据进行聚类的时候都会被广泛应用。
算法的目的是,先指定聚类的数目c,然后将输入的数据划分为c类,值簇内的数据之间具有较高的相似程度,而簇之间的相似程度较低。
下面简单介绍下Kmeans算法的实现,具体的网上都可以找到。
Kmeans的目标函数是:
c是聚类的中心,目的就是让每个点到它所属于的中心的距离之和最小。
![]()
因此对目标函数求偏导可以得到如下,其中Nj是第j类中数据点的个数。
![]()
然后就是对所有数据进行repeat直到中心点不再发生变化或者达到了最大的遍历次数。
2、FCM
FCM是一种基于模糊集合为基础的聚类方法,它是以隶属度来确定每个数据点从属于某个中心。像Kmeans这类算法称为硬聚类,而FCM则称为软聚类,是传统硬聚类的一种改进。为什么叫软跟硬,因为FCM在聚类的时候,会计算每个样本点到中心的隶属度,这个隶属度是一个0~100%的数值,而硬聚类则只有0%和100%,FCM通过这个隶属度可以使我们更加直观的了解一个数据点到中心的可信度。
因而这里就要提到一个隶属度矩阵了,为了对比Kmeans,也给Kmeans设置了一个隶属度的矩阵:
![]()
![]()
通过上面的对比就可看出关于这个隶属度矩阵的作用了。
下面说一下关于FCM算法的思路:
FCM的目标函数如下:
![]()
其中m是加权指数,一般的应用区间是[1.5,2.5]。网上也有很多研究是关于FCM中这个m的优化的。
可以看出,FCM目标函数就是在Kmeans中目标函数的基础
模糊C均值(FCM)算法设图像像素值组成n个样本的集合X={x1![]() , x2
, x2![]() ,…,xn
,…,xn![]() },则图像分割问题就转化为将这n个样本分成c个聚类的问题.根据图像中像素和聚类中心的加权相似测度,对目标函数进行迭代优化以确定最佳聚类.FCM的目标函数为:
},则图像分割问题就转化为将这n个样本分成c个聚类的问题.根据图像中像素和聚类中心的加权相似测度,对目标函数进行迭代优化以确定最佳聚类.FCM的目标函数为:
约束项:i=1Cuij=1,uij∈0,1![]()
(j∈[1,n],i∈[1,c])
式中:m∈(1,∞)为加权指数(图像处理一般取m=2); uij![]() 是第i类中样本xj
是第i类中样本xj![]() 的隶属度;dij=xj-vi
的隶属度;dij=xj-vi![]() 为样本与聚类中心的欧式距离。vi
为样本与聚类中心的欧式距离。vi![]() 是第i个聚类中心.
是第i个聚类中心.
聚类要达到的最终效果就是类内相似度最大,类间相似度最小,这个时候点和中心的加权距离之和就是最小的。所以我们只要使得目标函数取得最小值.
利用拉格朗日乘子法结合约束项,得:
F=j=1Ni=1Cuijmdij 2+j=1Nλji=1Cuij-1;(2.2)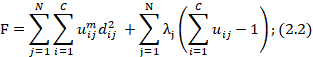
- 对uij
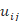 求偏导数:
求偏导数:
令(3)式为0,求得:
根据约束项,求得:
带入(4)式。得:
- 对vi
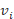 求偏导数:
求偏导数:
令(7)式为0,求得:
图像的优化分割就是通过迭代寻找聚类中心vi![]() 与隶属度值uij
与隶属度值uij![]() ,使目标函数F取最小.,最终的聚类中心就通过最后的隶属度函数来确定。
,使目标函数F取最小.,最终的聚类中心就通过最后的隶属度函数来确定。
前提准备
没有安装skfuzzy的话,可以先pip install skfuzzy。其中skfuzzy是python中用于研究模糊推理、模糊神经网络的模块,其中有很多实现好的算法和函数。
sklearn.KMeans
先看一下Kmeans这个类的参数:
def __init__(self, n_clusters=8, init='k-means++', n_init=10, max_iter=300,
tol=1e-4, precompute_distances='auto',
verbose=0, random_state=None, copy_x=True, n_jobs=1):1、n_clusters就是k值,一般需要通过测试来选择最好的聚类数目。
2、max_iter最大迭代数目,如果是凸函数的话,求导可以得到极值,因而可以不用管,但是如果是非凸函数的话,可能会不收敛,此时可以指定最大的迭代次数。
3、init即初始值选择的方式,可以是radom随机选择,或者优化过的k-means++,或者自己指定的初始化的质心。
创建了Kmeans对象之后,接着调用fit()函数来训练模型,然后通过predict()可以得到每个数据对应的label。
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
from skfuzzy.cluster import cmeans
cp = np.random.uniform(1, 100, (100, 2))
train = cp[:50]
test = cp[50:]
km = KMeans(n_clusters=3)
km.fit(train)
result = km.predict(train)
for i in range(50):
if result[i] == 0:
plt.scatter(train[i,0],train[i,1], c='r')
elif result[i] == 1:
plt.scatter(train[i, 0],train[i,1], c='g')
elif result[i] == 2:
plt.scatter(train[i, 0],train[i,1], c='b')
plt.show()
![]()
skfuzzy.cmeans
先看一下这个函数的参数跟返回值:
def cmeans(data, c, m, error, maxiter, init=None, seed=None):data : 2d array, size (S, N)
Data to be clustered. N is the number of data sets; S is the number
of features within each sample vector.
c : int
Desired number of clusters or classes.
m : float
Array exponentiation applied to the membership function u_old at each
iteration, where U_new = u_old ** m.
error : float
Stopping criterion; stop early if the norm of (u[p] - u[p-1]) < error.
maxiter : int
Maximum number of iterations allowed.
init : 2d array, size (S, N)
Initial fuzzy c-partitioned matrix. If none provided, algorithm is
randomly initialized.
seed : int
If provided, sets random seed of init. No effect if init is
provided. Mainly for debug/testing purposes.
1、data就是训练的数据。这里需要注意data的数据格式,shape是类似(特征数目,数据个数),与很多训练数据的shape正好是相反的。
2、c是需要指定的聚类个数。
3、m也就是上面提到的隶属度的指数,是一个加权指数。
4、error就是当隶属度的变化小于此的时候提前结束迭代。
5、maxiter最大迭代次数。
Returns
-------
cntr : 2d array, size (S, c)
Cluster centers. Data for each center along each feature provided
for every cluster (of the `c` requested clusters).
u : 2d array, (S, N)
Final fuzzy c-partitioned matrix.
u0 : 2d array, (S, N)
Initial guess at fuzzy c-partitioned matrix (either provided init or
random guess used if init was not provided).
d : 2d array, (S, N)
Final Euclidian distance matrix.
jm : 1d array, length P
Objective function history.
p : int
Number of iterations run.
fpc : float
Final fuzzy partition coefficient.
返回值:
1、cntr聚类的中心。
2、u是最后的的隶属度矩阵。
3、u0是初始化的隶属度矩阵。
4、d是最终的每个数据点到各个中心的欧式距离矩阵。
5、jm是目标函数优化的历史。
6、p是迭代的次数。
7、fpc全称是fuzzy partition coefficient,是一个评价分类好坏的指标。它的范围是0到1,1是效果最好。后面可以通过它来选择聚类的个数。
代码如下:
train = train.T
center, u, u0, d, jm, p, fpc = cmeans(train, m=2, c=3, error=0.005, maxiter=1000)
for i in u:
label = np.argmax(u, axis=0)
print(label)
for i in range(50):
if label[i] == 0:
plt.scatter(train[0][i], train[1][i], c='r')
elif label[i] == 1:
plt.scatter(train[0][i], train[1][i], c='g')
elif label[i] == 2:
plt.scatter(train[0][i], train[1][i], c='b')
plt.show()![]()
在进行图像处理时,应该明白处理的也是一堆数据,所以我们可以将tu'x图像的像素值读取,再代入库函数:
import numpy as np
import PIL.Image as image
from sklearn.cluster import KMeans
from skfuzzy.cluster import cmeans
from pylab import*
import pydicom
def loadData(filepath):
f = open(filepath, 'rb')
data = []
img = image.open(f)
m, n = img.size
for i in range(m):
for j in range(n):
x, y, z = img.getpixel((i, j))
data.append([x / 256.0, y / 256.0, z / 256.0])
f.close()
return np.mat(data), m, n
imgData, row, col = loadData('02.jpg')
print(imgData, row, col)
# label = KMeans(n_clusters=5).fit_predict(imgData)
# dcm = pydicom.read_file("D:\\Pychram\\PATIENT_DICOM\\image_1")
# dcm.image = dcm.pixel_array * dcm.RescaleSlope + dcm.RescaleIntercept #将灰度值转化为CT值
# slices = []
# slices.append(dcm)
# img = slices[int(len(slices)/2)].image.copy()
#
# row, col = img.shape
# for i in range(row):
# for j in range(col):
# img[i, j] /= 256
# data = []
# for i in range(row):
# for j in range(col):
# x= y=z = img((i, j))
# data.append([x / 256.0, y / 256.0, z / 256.0])
# print(img)
imgData = imgData.T
center, u, u0, d, jm, p, fpc = cmeans(imgData, m=2, c=6, error=0.0001, maxiter=1000)
for i in u:
label = np.argmax(u, axis=0) # 取得列的最大值
label = label.reshape([row, col]) # 转化维度
print(fpc)
print(center)
print(np.max(label))
pic_new = image.new('L', (row, col))
print(pic_new)
# 最终利用聚类中心点的RGB值替换原图中每一个像素点的值,便得到了最终的分割后的图片
for i in range(row):
for j in range(col):
pic_new.putpixel((i, j), int(256 / (label[i][j] + 1)))
# pic_new.save('result.jpg')
plt.imshow(pic_new)
plt.show()注释的代码是医学图像读取,感兴趣可以看看我的其他博客。