这篇文章的题目是《江豚种群基因组学揭示了一种适应淡水环境的鲸目动物》,主要内容是利用全基因组测序及重测序研究江豚种群分化和独特的适应性差异,解决了江豚的分类问题。
原文链接
PMCID: PMC5893588 DOI: 10.1038/s41467-018-03722-x
1 Introduction
鲸鱼、海豚、 鼠海豚统称为鲸豚类动物,这是一类适应多样性水生环境的哺乳动物,从海洋到淡水都有分布。 鲸豚类有着与众不同的进化历史,从陆入海,这是可以说是进化历史上非常奇迹的事件,故而引起研究人员的兴趣。
江豚是一群小型鲸豚类动物,其特征是:具有铁锹状的牙齿,短而钝的鼻子、缺乏真正的背鳍,栖息在亚洲南部和东部海岸。江豚的起源: 一些分子证据表明江豚起源于海洋祖先,随后逐渐占据不同的水生生态位。
在中国,江豚主要分布在中国的沿海海域,但有一个特殊的种群分布在长江,这个特殊的江豚种群成为了世界上唯一一个生活在淡水环境中的江豚种群,这使得长江江豚成为研究鲸豚类动物对淡水的进化适应的理想的自然模型。(白鱀豚以前也是生活在长江流域,但自2006年进行大规模调查以来,在野外没有发现任何个体,已被确认为功能性灭绝)。长江江豚自2013年以来,它一直被IUCN 视为极度濒危物种,如果不紧急执行有效的保护措施,它可能最终灭绝。
江豚有3个地理种群:①生活在印度洋沿海附近的宽脊江豚;②生活在淡水环境中的窄脊江豚(长江江豚);③生活在南海、黄海及日本海中部的窄脊江豚。先前利用mtDNA单倍型和形态学特征对这3个地理种群的分类地位提出了两种假说:(1)三亚种假说:三者属于同一物种的不同亚种;(2)两物种假说:印度洋沿岸的两个物种——宽脊江豚和窄脊江豚,后者包括两个亚种,即生活在淡水环境(中国长江流域)的窄脊江豚和生活在海水环境中的一种窄脊江豚。
本文的主要目的是利用全基因组测序来解决这个分类问题,以促进当前和未来的保护工作。
2 Materials and Methods
2.1样本与测序
所有用于测序的江豚都是在1979年至2009年期间被发现搁浅的,样本保存在-20℃冰箱中。从骨骼肌中提取基因组DNA。采用二代测序,测序平台为Illumina HiSeq 2000。用于从头组装的雄性窄脊江豚(长江江豚)测序深度为106×;另有48只江豚用于重测序,平均测序深度为14.5×(这48只江豚分别采自南海、黄海和长江流域)。de novo测序的原始数据在过滤掉接头、低质量和重复的reads之后,得到了总共265.5 GB的高质量数据,再被用于基因组组装。转录组测序,采集了两只长江江豚的新鲜血液用于rna-seq分析。
2.2基因家族和正选基因分析
下载了弓头鲸的基因组序列,利用TreeFam构建了基因家族。利用长江江豚、 其他6种鲸目动物和10种其他哺乳动物的3911单拷贝基因,使用RAxML软件最大似然算法重建了系统发育树。分歧时间基于四重简并密码子位点,利用Reltime 进行估算。分支位点模型用于检测沿着目标分支的阳性选择。每一个基因的p值是用似然值来计算的。人工检测似然值(LRTs)和P值小于0.05的基因序列。
2.3序列比对和寻找变异
利用BWA将高质量的reads比对到组装好的长江江豚的基因组上,并使用Samtools实现的Bayes方法处理对齐。利用Picard和GATK基于HMM似然功能寻找变异。为了确定SNPs的pseudo-chromosome
上的位置,对长江江豚的的scaffold进行定位,并根据其与牛基因组的同源性将其组装到染色体上。
2.4系统发育与种群结构
利用RAxML中最大似然法和VCF-kit中邻接法对48只长江江豚进行系统发育分析。用EIGENSOFT3.0进行主成分分析(PCA),用Tracey-Widom检验确定特征向量的意义。FDIST 用来确定在PCA分析和系统发育重建的中是否存在中性SNPs 。用ADMIXTURE 推断种群结构。长江江豚的祖先序列是用程序框架推断出来的,没有预先假定它们的结构。采用BEAGLE和连锁不平衡模式对ibd单倍型进行了分析,计算任意两个位点间的决定系数,对每个群体或遗传聚类进行分析。
2.5种群历史重建与基因流
psmc模型,利用整个基因组的杂合子位点来估计有效种群大小的变化,这种方法在其他哺乳动物中得到了广泛的应用。
msmc模型,用于估算种群分化的时间。
2.6在选择下检测SNPs
为了研究无鳍海豚的选择信号,首先使用Sweepfinder 2扫描基因组,寻找阳性选择的目标区域。提取了与扫描区重叠或接近的基因(在1 Mb范围内)。使用XP-Ehh确定长江与海中的窄脊江豚之间基因组的选定区域进行独立性测试。
3 Results
3.1 江豚基因组组装和基因组变异
组装了2.3Gb的基因组,contig N50为26.7,scaffold N50为 6.3 Mb。使用同源基因、重测序以及血液转录组测序,预测到了22014个蛋白编码基因,也就是说江豚基因组中有93%的基因有功能分类。利用这些蛋白编码基因生成了哺乳动物进化的时间尺度,并利用分支位点模型在江豚中鉴定出了57个正选择基因。采集并测序了48只分布在长江中下游以及沿海地区的窄脊江豚,测序平均深度为14.5×,鉴定出了1.33百万个SNPs。
3.2 江豚的系统发育与种群结构
通过系统发育重建和主成分分析(PCA)利用平均遗传距离对个体进行聚类。紫色代表南海的样本,绿色代表黄海的样本,蓝色代表长江的样本。通过系统发育重建可以发现,来自南海和黄海中的宽脊江豚聚为了一支,南海和黄海的窄脊江豚聚为了一支,长江流域的窄脊江豚聚为了一支。PCA分析也得到了同样的结果。
单倍型同源一致性的测定,表明长江江豚内部个体之间共享较大的单倍型片段。
k代表的是祖先成分数量,当k=2时,宽脊江豚和窄脊江豚分开了;当k=3时,长江中的窄脊江豚和海洋中的窄脊江豚分开了。
以上几个分析交叉验证,初步表明江豚已经分化为了三个不同的种。
3.3 种群历史和物种划分
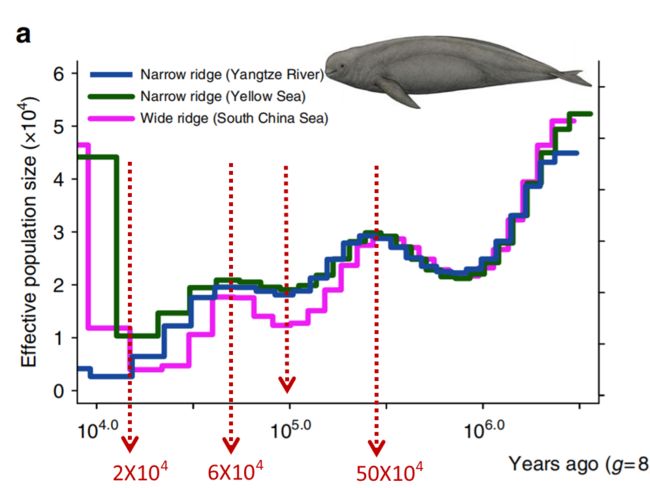
为了重建江豚的种群历史,首先采用PSMC方法。Ne表示的是有效种群规模,三个主要群体的Ne值一直到2万年前都是高度相关的。在大约50万年前和大约6万年前观测到了Ne的两个峰值,并在大约10万年前观察到了种群规模的缩小。从50万年前到2万年前这个时间段中,窄脊江豚相比宽脊江豚都表现出相对较高的Ne值,两者的Ne差异在10万年前时达到最大值。
窄脊江豚的两条Ne曲线在大约两万年前发生分离,这可能意味着此时种群发生了分化。但也有可能是因为在利用psmc法预测近期有效种群数量变化的误差较大所产生的结果。
进一步使用MSMC模型推测种群分离时间。MSMC曲线使用RCCR值做出来的,RCCR值代表两群体间的分化时间和速率:RCCR=1,表示两群体完全均匀混合;RCCR=0.5,表示两种群开始分化;RCCR=0,表示两种群完全分离。
从图上可以看出,RCCR值在最近一段时间都趋近于零,表明在过去的5千年,各个群体之间已经完全分离,没有基因流动。曲线在4万年和10万之前分别达到0.5,并分别在10万年和50万左右的达到最大值。由此,能得到两个结论:根据紫色曲线可以知道,窄脊江豚和宽脊江豚大约在5000到10万前逐渐分化开来的;根据绿色曲线可以知道,海洋窄脊江豚和淡水窄脊江豚大约是在5000到4万年前开始逐渐分化开来的。
推测造成宽脊江豚和窄脊江豚分化的原因应该是由于更新世末期的冰川作用造成的江豚祖先分离;而淡水窄脊江豚种群与海洋窄脊江豚种群的分离则可能是由于末次盛冰期前后的海平面振荡造成的。
另外,使用了三种群检验,表明长江江豚和其他江豚种群中都没有古老的混合体;三组江豚还利用Bayes因子进行物种划分分析也支持了他们之间存在完全的生殖隔离。
综上,三亚种假说和两物种假说都是错误的,江豚已经分化为了3个独立的物种了。
3.4 低氧相关基因阳性选择的特征
尽管台湾海峡和周围水域是共通的,但是宽脊江豚和窄脊江豚在形态上却有着很大的差异,例如结节区的宽度和背部疣粒的数量都是明显不同的。为了检查形态上的差异是否在基因上会也存在正选择,利用SweepFinder2对基因组进行检测。在39个具有强选择性扫描信号的宽脊和窄脊群体中鉴定出了218个基因和144个候选基因。
这些候选基因在突触后膜电位的调节、神经元动作电位、神经肽信号通路以及对生物刺激的反应等生物过程中显著富集。
进一步研究宽脊江豚的KCNMA1、VCAM1、TXN2、ADSL、CAPN2、ERCC3、PLAU基因,发现它们大多与低氧适应有关。而非常有趣的一点就是,两种江豚的典型栖息地在海平面上有很大的差异,这可能与低氧有关。窄脊江豚主要生活在台湾海峡北至韩国和日本中部的温带沿海水域,并且通常是在浅海区域,几乎不到150米深。然而,宽脊江豚主要生活在印度洋和东南亚的热带和亚热带水域中,平均海平面相对较深。表明这些基因与宽脊江豚的适应低氧环境有关。
3.5 长江江豚对淡水的遗传适应
窄脊江豚可以栖息在淡水和海水环境。为了确定长江江豚和海洋环境中的窄脊江豚的基因中是否存在阳性突变,于是采用了XP-EHH 中性检测,这幅图代表的是归一化了的XP-Ehh值,其阈值是在3.91−2.70这个范围内,高于或低于这个阈值都将被划到候选突变中去,于是总共确定了6035个候选SNPs。这些基因在“肾脏发育”和“输尿管发育”等功能中明显富集,这可能有助于淡水和海洋窄脊江豚独特的肾脏结构形成。
例如,ADCY1, DYNC2H1和 SLC14A2这3个基因 都是海洋窄脊江豚的候选基因,它们与尿素输送等功能相关。
其中,SLC14A2这个基因含有一个内含子候选SNP,这个基因在这个突变位点处的XP-EHH值明显高于阈值,Fst代表两种群差异,第三个图代表这个基因候选突变附近的频率差异,它们的值都很高,表明海洋中的窄脊江豚和场长江江豚在此处的差异是非常明显的,后面两幅图也说明了同样的情况。此外,肾转录组分析显示SLC14A2在淡水和海洋的窄脊江豚之间存在差异表达,其在长江江豚中表现为低表达。东亚江豚相比长江江豚,其尿量显著增加,而血清尿素水平却较低。同样,长江江豚的尿液渗透压低于东亚江豚,这与其生活在淡水环境有关。
综上,表明基因的进化对于江豚适应不同的生境至关重要。调节肾功能的基因变异驱动着江豚对淡水的适应。
4 Discussion
以上分析表明,这三个主要群体之间存在生殖分离,在这几千年的时间里都没有基因交流。它们显示出独特的个体化特征,以适应不同的环境。
①比较窄脊江豚和宽脊江豚,找到了大量与低氧相关的候选基因表现出强烈的正选择,可能是这两个群体的栖息环境中的海平面差异所导致的结果。
②长江江豚和生活在海洋窄脊江豚表现出许多与肾功能有关的选择特征,这可能与适应海洋和淡水不同的生境相关系。
尽管长江江豚最初是在20世纪70年代才作为一个独立的物种被对待的,但到目前为止,仍然有许多科研人员并不接受这一观点。然而,目前关于江豚种群分化显著、缺乏基因流动和独特的适应性差异的发现表明,江豚的种群分化是明显的。 因此,窄脊江豚被划分为两个物种,分别是长江江豚Neophocoena asiaeorientalis Pilleri and Gihr, 1972 和东亚江豚 Neophocoena sunameri Pilleri and Gihr, 1972 。
写在最后 | 这是第一次看种群基因组学的文章,收获颇丰,但仍然有许多问题经过查资料也还是不懂的。 ̄□ ̄||尤其是一些模型的原理,相信今后通过知识的积累会逐步搞懂的。其中若是有错误的地方,欢迎各位指出。还有就是,找了一些比较奈斯的图片,天使的微笑确实能让人心情愉悦。