【小白python】将excel表中的碱基频率可视化
目录
- 写代码的小背景
- 题目
- 预览结果
- 分析
- 1.将表格读到内存里
- 2.循环统计每列每个碱基的出现次数并记录
- 3.作图
- 完整代码
写代码的小背景
单纯的就是完成个作业,之前的小作业都是借鉴的同学的代码(别向我学这个hhhhh),第一次写博客,请多指教!
下面是正题:
题目
附表等位基因中的行表示10个病人样本,列表示DNA片段中的15个位点,试着将碱基出现频率可视化(有时间我把表格上传到资源里大家可以试着练一下)
表格为10(人数)*15(等位基因的位点)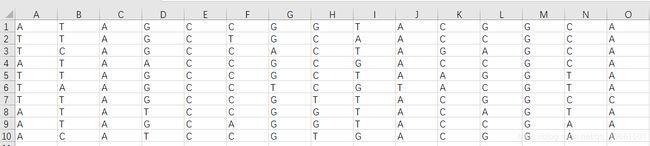
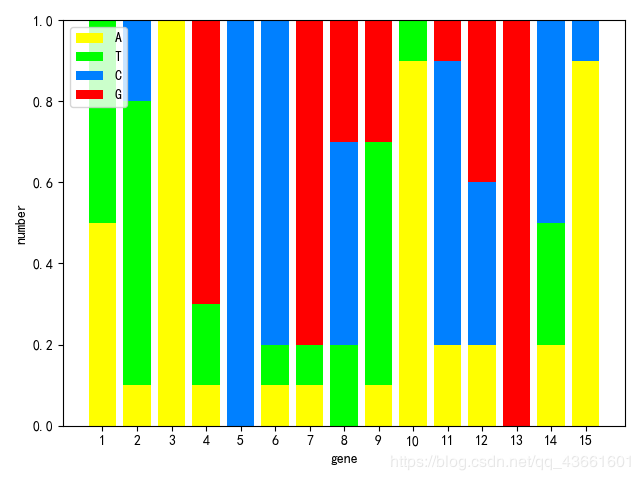
预览结果
分析
首先,分析一下需要做的工作有哪些:
1.将表格读到内存里
2.循环统计每一列每一个碱基出现次数并记录下来
3.作图
1.将表格读到内存里
import pandas as pd
data = pd.read_excel('gene.xlsx')
顺便简单介绍一下相关表格操作:
参考博客:利用Python处理Excel数据 这里只列出了需要的一部分功能
- 查看数据的行数和列数
df.shape
输出:(10,15)
意义:10行15列
- 按位置进行提取(iloc函数)
- 按区域提取
#行数0-4,列数0-5
df.iloc[:4, :5]
- 按位置提取
#[0, 2, 5] 代表指定的行,[ 4, 5 ] 代表指定的列
df.iloc[[0,2,5],[4,5]]
2.循环统计每列每个碱基的出现次数并记录
先定义4个空列表A、T、C、G,然后循环一次往里面添加一组数据(出现次数*0.1=每个碱基的频率)
A = []
T = []
G = []
C = []
for i in range(15):#15列
x.append(i)
seq = data.iloc[0:10, i]#每次读取一列
base = {'A': 0, 'T': 0, 'C': 0, 'G': 0}
for nucl in range(len(seq)):
if seq[nucl] in base:
base[seq[nucl]] += 1
for each in sorted(base.keys()):
if(each == 'A'):
A.append(base[each]*0.1)
elif (each == 'T'):
T.append(base[each]*0.1)
elif (each == 'C'):
C.append(base[each]*0.1)
elif (each == 'G'):
G.append(base[each]*0.1)
3.作图
搜了半天才知道需要作的图名字叫堆积柱状图,说白了就是另外一组的数据堆在前面的数据的上面
参考博客:【Python】画个堆积柱状图(三类数据的)
特别注意:
地方就是在设置第三类数据的底部,应该是第一类加第二类作底部,同理第四类也要在第三类基础上加合。
for i in range(0, len(A)):
T[i] = T[i] + A[i]
plt.bar(x, C, align="center", bottom=T, color="#0080FF", label="C")
import matplotlib.pyplot as plt
plt.bar(x, A, align="center", color="#FFFF00", tick_label=['1', '2', '3', '4', '5', '6', '7', '8', '9', '10', '11', '12', '13', '14', '15'], label="A")
plt.bar(x, T, align="center", bottom=A, color="#00FF00", label="T")
for i in range(0, len(A)):
T[i] = T[i] + A[i]
plt.bar(x, C, align="center", bottom=T, color="#0080FF", label="C")
for i in range(0, len(A)):
C[i] = C[i] + T[i]
plt.bar(x, G, align="center", bottom=C, color="#FF0000", label="G")
plt.xlabel("gene")
plt.ylabel("number")
plt.legend()
plt.show()
可能没讲明白,可以试试把for循环的部分删去画出图,可以发现频率的和不等于1,也就是堆积柱状图的底没有选对。可以将上面的代码改成这部分再运行,就可以看到后面有bug的结果了
import matplotlib.pyplot as plt
plt.bar(x, A, align="center", color="#FFFF00", tick_label=['1', '2', '3', '4', '5', '6', '7', '8', '9', '10', '11', '12', '13', '14', '15'], label="A")
plt.bar(x, T, align="center", bottom=A, color="#00FF00", label="T")
plt.bar(x, C, align="center", bottom=T, color="#0080FF", label="C")
plt.bar(x, G, align="center", bottom=C, color="#FF0000", label="G")
plt.xlabel("gene")
plt.ylabel("number")
plt.legend()
plt.show()
错误图例
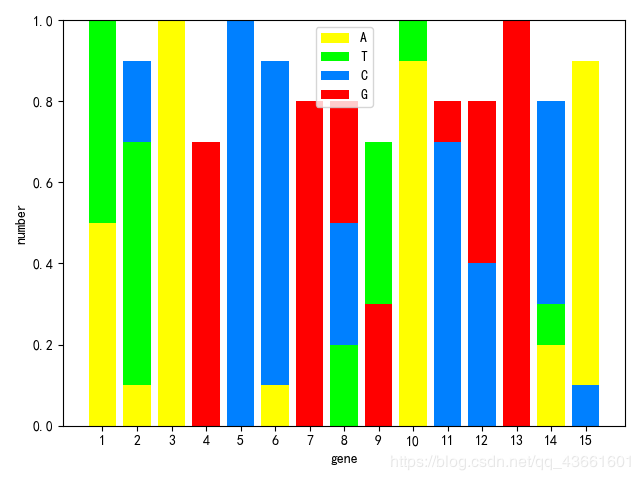
很明显,这个有些频率没有达到1,就是这个bug的问题了
完整代码
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib as mpl
mpl.rcParams["font.sans-serif"] = ["SimHei"]
mpl.rcParams["axes.unicode_minus"] = False
data = pd.read_excel('gene.xlsx')
x = []
A = []
T = []
G = []
C = []
for i in range(15):
x.append(i)
seq = data.iloc[0:10, i]
base = {'A': 0, 'T': 0, 'C': 0, 'G': 0}
for nucl in range(len(seq)):
if seq[nucl] in base:
base[seq[nucl]] += 1
for each in sorted(base.keys()):
if each == 'A':
A.append(base[each]*0.1)
elif each == 'T':
T.append(base[each]*0.1)
elif each == 'C':
C.append(base[each]*0.1)
elif each == 'G':
G.append(base[each]*0.1)
plt.bar(x, A, align="center", color="#FFFF00", tick_label=['1', '2', '3', '4', '5', '6', '7', '8', '9', '10', '11', '12', '13', '14', '15'], label="A")
plt.bar(x, T, align="center", bottom=A, color="#00FF00", label="T")
for i in range(0, len(A)):
T[i] = T[i] + A[i]
plt.bar(x, C, align="center", bottom=T, color="#0080FF", label="C")
for i in range(0, len(A)):
C[i] = C[i] + T[i]
plt.bar(x, G, align="center", bottom=C, color="#FF0000", label="G")
plt.xlabel("gene")
plt.ylabel("number")
plt.legend()
plt.show()
笔者算python小白,代码有很多部分可以优化但是不大会,算是笔者第一次写python吧,如觉着代码有问题之类的请留言,感谢!