R语言:SMOTE - Supersampling Rare Events in R:用R对非平衡数据的处理方法
SMOTE - Supersampling Rare Events in R:用R对稀有事件进行超级采样
在这个例子中将用到以下三个包
{DMwR} - Functions and data for the book “Data Mining with R” and SMOTE algorithm:SMOTE算法
{caret} - modeling wrapper, functions, commands:模型封装、函数、命令
我们利用 Thyroid Disease 数据来进行研究。
让我们清洗一些数据
# 加载数据,删除冒号和句号,并追加列名
hyper <-read.csv('http://archive.ics.uci.edu/ml/machine-learning-databases/thyroid-disease/hypothyroid.data', header=F)
names <- read.csv('http://archive.ics.uci.edu/ml/machine-learning-databases/thyroid-disease/hypothyroid.names', header=F, sep='\t')[[1]]
names <- gsub(pattern =":|[.]", replacement="", x = names)
colnames(hyper)<-names
# 我们将第一列的列名从 hypothyroid, negative改成target,并将negative变成0,其他值变成1.
colnames(hyper)[1]<-"target"
colnames(hyper)
## [1] "target" "age"
## [3] "sex" "on_thyroxine"
## [5] "query_on_thyroxine" "on_antithyroid_medication"
## [7] "thyroid_surgery" "query_hypothyroid"
## [9] "query_hyperthyroid" "pregnant"
## [11] "sick" "tumor"
## [13] "lithium" "goitre"
## [15] "TSH_measured" "TSH"
## [17] "T3_measured" "T3"
## [19] "TT4_measured" "TT4"
## [21] "T4U_measured" "T4U"
## [23] "FTI_measured" "FTI"
## [25] "TBG_measured" "TBG"
hyper$target<-ifelse(hyper$target=="negative",0,1)
# 检查下阳性和阴性的结果
table(hyper$target)
##
## 0 1
## 3012 151
prop.table(table(hyper$target))
##
## 0 1
## 0.95226 0.04774
# 可见,1仅有5%。这显然是一个扭曲的数据集,也是罕见事件。
head(hyper,2)
## target age sex on_thyroxine query_on_thyroxine on_antithyroid_medication
## 1 1 72 M f f f
## 2 1 15 F t f f
## thyroid_surgery query_hypothyroid query_hyperthyroid pregnant sick tumor
## 1 f f f f f f
## 2 f f f f f f
## lithium goitre TSH_measured TSH T3_measured T3 TT4_measured TT4
## 1 f f y 30 y 0.60 y 15
## 2 f f y 145 y 1.70 y 19
## T4U_measured T4U FTI_measured FTI TBG_measured TBG
## 1 y 1.48 y 10 n ?
## 2 y 1.13 y 17 n ?
# 这数据都是因子型变量(字符型的值),这些都需要转换成二值化的数字,以方便建模:
ind<-sapply(hyper,is.factor)
hyper[ind]<-lapply(hyper[ind],as.character)
hyper[hyper=="?"]=NA
hyper[hyper=="f"]=0
hyper[hyper=="t"]=1
hyper[hyper=="n"]=0
hyper[hyper=="y"]=1
hyper[hyper=="M"]=0
hyper[hyper=="F"]=1
hyper[ind]<-lapply(hyper[ind],as.numeric)
replaceNAWithMean<-function(x) {replace(x,is.na(x),mean(x[!is.na(x)]))}
我们利用caret包中的createDataPartition(数据分割功能)函数将数据随机分成相同的两份。
library(caret)
## Loading required package: lattice
## Loading required package: ggplot2
set.seed(1234)
splitIndex<-createDataPartition(hyper$target,time=1,p=0.5,list=FALSE)
trainSplit<-hyper[splitIndex,]
testSplit<-hyper[-splitIndex,]
prop.table(table(trainSplit$target))
##
## 0 1
## 0.95006 0.04994
prop.table(table(testSplit$target))
##
## 0 1
## 0.95446 0.04554
两者的分类结果是平衡的,因此仍然有5%左右的代表,我们仍然处于良好的水平。
我们利用caret包中的treebag模型算法,对训练集数据建立模型,并对测试集数据进行预测。
ctrl<-trainControl(method="cv",number=5)
tbmodel<-train(target~.,data=trainSplit,method="treebag",
trControl=ctrl)
## Loading required package: ipred
## Loading required package: plyr
predictors<-names(trainSplit)[names(trainSplit)!='target']
pred<-predict(tbmodel$finalModel,testSplit[,predictors])
为了评估模型,我们用pROC包的roc函数算auc得分和画图
library(pROC)
## Type 'citation("pROC")' for a citation.
##
## Attaching package: 'pROC'
##
## 下列对象被屏蔽了from 'package:stats':
##
## cov, smooth, var
auc<-roc(testSplit$target,pred)
print(auc)
##
## Call:
## roc.default(response = testSplit$target, predictor = pred)
##
## Data: pred in 1509 controls (testSplit$target 0) < 72 cases (testSplit$target 1).
## Area under the curve: 0.985
plot(auc,ylim=c(0,1),print.thres=TRUE,main=paste('AUC',round(auc$auc[[1]],2)))
##
## Call:
## roc.default(response = testSplit$target, predictor = pred)
##
## Data: pred in 1509 controls (testSplit$target 0) < 72 cases (testSplit$target 1).
## Area under the curve: 0.985
abline(h=1,col="blue",lwd=2)
abline(h=0,col="red",lwd=2)

auc得分是0.98,已经是非常不错的结果了(因为它的范围是在0.5到1之间)。
## Loading required package: grid
##
## Attaching package: 'DMwR'
##
## 下列对象被屏蔽了from 'package:plyr':
##
## join
trainSplit$target<-as.factor(trainSplit$target)
trainSplit<-SMOTE(target~.,trainSplit,perc.over=100,perc.under=200)
trainSplit$target<-as.numeric(trainSplit$target)
# 我们再次用prop.table()函数检查结果的平衡性,确定我们已经让阴性、阳性数据达到相同。
prop.table(table(trainSplit$target))
##
## 1 2
## 0.5 0.5
# 再次建立treebag模型
tbmodel<-train(target~.,data=trainSplit,method="treebag",
trControl=ctrl)
predictors<-names(trainSplit)[names(trainSplit)!='target']
pred<-predict(tbmodel$finalModel,testSplit[,predictors])
auc<-roc(testSplit$target,pred)
print(auc)
##
## Call:
## roc.default(response = testSplit$target, predictor = pred)
##
## Data: pred in 1509 controls (testSplit$target 0) < 72 cases (testSplit$target 1).
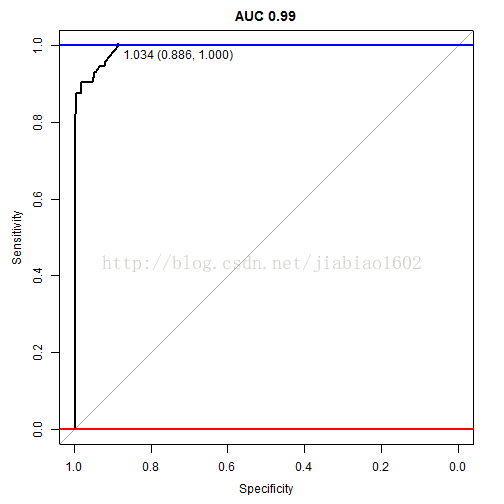
## Area under the curve: 0.99
哇,达到0.99,比之前的0.985有提高
plot(auc,ylim=c(0,1),print.thres=TRUE,main=paste('AUC',round(auc$auc[[1]],2)))
##
## Call:
## roc.default(response = testSplit$target, predictor = pred)
##
## Data: pred in 1509 controls (testSplit$target 0) < 72 cases (testSplit$target 1).
## Area under the curve: 0.99
abline(h=1,col="blue",lwd=2)
在这个例子中将用到以下三个包
{DMwR} - Functions and data for the book “Data Mining with R” and SMOTE algorithm:SMOTE算法
{caret} - modeling wrapper, functions, commands:模型封装、函数、命令
{pROC} - Area Under the Curve (AUC) functions:曲线下面积(ACU)函数
我们利用 Thyroid Disease 数据来进行研究。
让我们清洗一些数据
# 加载数据,删除冒号和句号,并追加列名
hyper <-read.csv('http://archive.ics.uci.edu/ml/machine-learning-databases/thyroid-disease/hypothyroid.data', header=F)
names <- read.csv('http://archive.ics.uci.edu/ml/machine-learning-databases/thyroid-disease/hypothyroid.names', header=F, sep='\t')[[1]]
names <- gsub(pattern =":|[.]", replacement="", x = names)
colnames(hyper)<-names
# 我们将第一列的列名从 hypothyroid, negative改成target,并将negative变成0,其他值变成1.
colnames(hyper)[1]<-"target"
colnames(hyper)
## [1] "target" "age"
## [3] "sex" "on_thyroxine"
## [5] "query_on_thyroxine" "on_antithyroid_medication"
## [7] "thyroid_surgery" "query_hypothyroid"
## [9] "query_hyperthyroid" "pregnant"
## [11] "sick" "tumor"
## [13] "lithium" "goitre"
## [15] "TSH_measured" "TSH"
## [17] "T3_measured" "T3"
## [19] "TT4_measured" "TT4"
## [21] "T4U_measured" "T4U"
## [23] "FTI_measured" "FTI"
## [25] "TBG_measured" "TBG"
hyper$target<-ifelse(hyper$target=="negative",0,1)
# 检查下阳性和阴性的结果
table(hyper$target)
##
## 0 1
## 3012 151
prop.table(table(hyper$target))
##
## 0 1
## 0.95226 0.04774
# 可见,1仅有5%。这显然是一个扭曲的数据集,也是罕见事件。
head(hyper,2)
## target age sex on_thyroxine query_on_thyroxine on_antithyroid_medication
## 1 1 72 M f f f
## 2 1 15 F t f f
## thyroid_surgery query_hypothyroid query_hyperthyroid pregnant sick tumor
## 1 f f f f f f
## 2 f f f f f f
## lithium goitre TSH_measured TSH T3_measured T3 TT4_measured TT4
## 1 f f y 30 y 0.60 y 15
## 2 f f y 145 y 1.70 y 19
## T4U_measured T4U FTI_measured FTI TBG_measured TBG
## 1 y 1.48 y 10 n ?
## 2 y 1.13 y 17 n ?
# 这数据都是因子型变量(字符型的值),这些都需要转换成二值化的数字,以方便建模:
ind<-sapply(hyper,is.factor)
hyper[ind]<-lapply(hyper[ind],as.character)
hyper[hyper=="?"]=NA
hyper[hyper=="f"]=0
hyper[hyper=="t"]=1
hyper[hyper=="n"]=0
hyper[hyper=="y"]=1
hyper[hyper=="M"]=0
hyper[hyper=="F"]=1
hyper[ind]<-lapply(hyper[ind],as.numeric)
replaceNAWithMean<-function(x) {replace(x,is.na(x),mean(x[!is.na(x)]))}
hyper<-replaceNAWithMean(hyper)
我们利用caret包中的createDataPartition(数据分割功能)函数将数据随机分成相同的两份。
library(caret)
## Loading required package: lattice
## Loading required package: ggplot2
set.seed(1234)
splitIndex<-createDataPartition(hyper$target,time=1,p=0.5,list=FALSE)
trainSplit<-hyper[splitIndex,]
testSplit<-hyper[-splitIndex,]
prop.table(table(trainSplit$target))
##
## 0 1
## 0.95006 0.04994
prop.table(table(testSplit$target))
##
## 0 1
## 0.95446 0.04554
两者的分类结果是平衡的,因此仍然有5%左右的代表,我们仍然处于良好的水平。
我们利用caret包中的treebag模型算法,对训练集数据建立模型,并对测试集数据进行预测。
ctrl<-trainControl(method="cv",number=5)
tbmodel<-train(target~.,data=trainSplit,method="treebag",
trControl=ctrl)
## Loading required package: ipred
## Loading required package: plyr
predictors<-names(trainSplit)[names(trainSplit)!='target']
pred<-predict(tbmodel$finalModel,testSplit[,predictors])
为了评估模型,我们用pROC包的roc函数算auc得分和画图
library(pROC)
## Type 'citation("pROC")' for a citation.
##
## Attaching package: 'pROC'
##
## 下列对象被屏蔽了from 'package:stats':
##
## cov, smooth, var
auc<-roc(testSplit$target,pred)
print(auc)
##
## Call:
## roc.default(response = testSplit$target, predictor = pred)
##
## Data: pred in 1509 controls (testSplit$target 0) < 72 cases (testSplit$target 1).
## Area under the curve: 0.985
plot(auc,ylim=c(0,1),print.thres=TRUE,main=paste('AUC',round(auc$auc[[1]],2)))
##
## Call:
## roc.default(response = testSplit$target, predictor = pred)
##
## Data: pred in 1509 controls (testSplit$target 0) < 72 cases (testSplit$target 1).
## Area under the curve: 0.985
abline(h=1,col="blue",lwd=2)
abline(h=0,col="red",lwd=2)

auc得分是0.98,已经是非常不错的结果了(因为它的范围是在0.5到1之间)。
很难想象SMOTE对此能再有提高了,但接下来我们利用SMOTE对数据处理后再建模,看看auc结果
在R中,SMOTE算法是DMwR软件包的一部分,主要参数有如下三个:perc.over:过采样时,生成少数类的样本个数;k:过采样中使用K近邻算法生成少数类样本时的K值,默认是5;perc.under:欠采样时,对应每个生成的少数类样本,选择原始数据多数类样本的个数。例如,perc.over=500表示对原始数据集中的每个少数样本,都将生成5个新的少数样本;perc.under=80表示从原始数据集中选择的多数类的样本是新生的数据集中少数样本的80%。
## Loading required package: grid
##
## Attaching package: 'DMwR'
##
## 下列对象被屏蔽了from 'package:plyr':
##
## join
trainSplit$target<-as.factor(trainSplit$target)
trainSplit<-SMOTE(target~.,trainSplit,perc.over=100,perc.under=200)
trainSplit$target<-as.numeric(trainSplit$target)
# 我们再次用prop.table()函数检查结果的平衡性,确定我们已经让阴性、阳性数据达到相同。
prop.table(table(trainSplit$target))
##
## 1 2
## 0.5 0.5
# 再次建立treebag模型
tbmodel<-train(target~.,data=trainSplit,method="treebag",
trControl=ctrl)
predictors<-names(trainSplit)[names(trainSplit)!='target']
pred<-predict(tbmodel$finalModel,testSplit[,predictors])
auc<-roc(testSplit$target,pred)
print(auc)
##
## Call:
## roc.default(response = testSplit$target, predictor = pred)
##
## Data: pred in 1509 controls (testSplit$target 0) < 72 cases (testSplit$target 1).
## Area under the curve: 0.99
哇,达到0.99,比之前的0.985有提高
plot(auc,ylim=c(0,1),print.thres=TRUE,main=paste('AUC',round(auc$auc[[1]],2)))
##
## Call:
## roc.default(response = testSplit$target, predictor = pred)
##
## Data: pred in 1509 controls (testSplit$target 0) < 72 cases (testSplit$target 1).
## Area under the curve: 0.99
abline(h=1,col="blue",lwd=2)
abline(h=0,col="red",lwd=2)