sklearn机器学习之特征工程
特征工程是什么
数据探索性分析Exploratory Data AnalysisEDA
数据预处理
无量纲化 数据规范化
标准化 0均值标准化Z-score standardization
区间缩放法线性函数归一化Min-Max scaling
标准化与归一化的区别 标准化规范化与归一化的区别
对定量特征二值化
对定性特征哑编码
缺失值计算
数据变换
回顾
特征选择
Filter
移除低方差的特征 Removing features with low variance
单变量特征选择 Univariate feature selection
卡方Chi2检验
Pearson相关系数 Pearson Correlation
互信息和最大信息系数 Mutual information and maximal information coefficient MIC
距离相关系数 Distance Correlation
Wrapper
递归特征消除法
Embedded
基于惩罚项的特征选择法
基于树模型的特征选择法
回顾
降维
主成分分析法PCA
线性判别分析法LDA
回顾
0 关于本文
主要内容和结构框架由@jasonfreak–使用sklearn做单机特征工程提供,其中夹杂了很多补充的例子,能够让大家更直观的感受到各个参数的意义,有一些地方我也进行自己理解层面上的纠错,目前有些细节和博主再进行讨论,修改部分我都会以删除来表示,读者可以自行斟酌,能和我一块讨论便是极好的!还是多谢原作者,我这里只是总结和补充
1 特征工程是什么?
有这么一句话在业界广泛流传:数据和特征决定了机器学习的上限,而模型和算法只是逼近这个上限而已。那特征工程到底是什么呢?顾名思义,其本质是一项工程活动,目的是最大限度地从原始数据中提取特征以供算法和模型使用。通过总结和归纳,人们认为特征工程包括以下方面:
特征选择主要有两个目的:
减少特征数量、降维,使模型泛化能力更强,减少过拟合; 增强对特征和特征值之间的理解。
特征处理是特征工程的核心部分,sklearn提供了较为完整的特征处理方法,包括数据预处理,特征选择,降维等。首次接触到sklearn,通常会被其丰富且方便的算法模型库吸引,但是这里介绍的特征处理库也十分强大!
本文中使用sklearn中的IRIS(鸢尾花)数据集来对特征处理功能进行说明。IRIS数据集由Fisher在1936年整理,包含4个特征(Sepal.Length(花萼长度)、Sepal.Width(花萼宽度)、Petal.Length(花瓣长度)、Petal.Width(花瓣宽度)),特征值都为正浮点数,单位为厘米。目标值为鸢尾花的分类(Iris Setosa(山鸢尾)、Iris Versicolour(杂色鸢尾),Iris Virginica(维吉尼亚鸢尾))。导入IRIS数据集的代码如下:
In [3]:
from sklearn.datasets import load_iris #导入IRIS数据集 iris = load_iris() #特征矩阵 iris.data
Out[3]:
array([[5.1, 3.5, 1.4, 0.2],
[4.9, 3. , 1.4, 0.2],
[4.7, 3.2, 1.3, 0.2],
[4.6, 3.1, 1.5, 0.2],
[5. , 3.6, 1.4, 0.2],
[5.4, 3.9, 1.7, 0.4],
[4.6, 3.4, 1.4, 0.3],
[5. , 3.4, 1.5, 0.2],
[4.4, 2.9, 1.4, 0.2],
[4.9, 3.1, 1.5, 0.1],
[5.4, 3.7, 1.5, 0.2],
[4.8, 3.4, 1.6, 0.2],
[4.8, 3. , 1.4, 0.1],
[4.3, 3. , 1.1, 0.1],
[5.8, 4. , 1.2, 0.2],
[5.7, 4.4, 1.5, 0.4],
[5.4, 3.9, 1.3, 0.4],
[5.1, 3.5, 1.4, 0.3],
[5.7, 3.8, 1.7, 0.3],
[5.1, 3.8, 1.5, 0.3],
[5.4, 3.4, 1.7, 0.2],
[5.1, 3.7, 1.5, 0.4],
[4.6, 3.6, 1. , 0.2],
[5.1, 3.3, 1.7, 0.5],
[4.8, 3.4, 1.9, 0.2],
[5. , 3. , 1.6, 0.2],
[5. , 3.4, 1.6, 0.4],
[5.2, 3.5, 1.5, 0.2],
[5.2, 3.4, 1.4, 0.2],
[4.7, 3.2, 1.6, 0.2],
[4.8, 3.1, 1.6, 0.2],
[5.4, 3.4, 1.5, 0.4],
[5.2, 4.1, 1.5, 0.1],
[5.5, 4.2, 1.4, 0.2],
[4.9, 3.1, 1.5, 0.2],
[5. , 3.2, 1.2, 0.2],
[5.5, 3.5, 1.3, 0.2],
[4.9, 3.6, 1.4, 0.1],
[4.4, 3. , 1.3, 0.2],
[5.1, 3.4, 1.5, 0.2],
[5. , 3.5, 1.3, 0.3],
[4.5, 2.3, 1.3, 0.3],
[4.4, 3.2, 1.3, 0.2],
[5. , 3.5, 1.6, 0.6],
[5.1, 3.8, 1.9, 0.4],
[4.8, 3. , 1.4, 0.3],
[5.1, 3.8, 1.6, 0.2],
[4.6, 3.2, 1.4, 0.2],
[5.3, 3.7, 1.5, 0.2],
[5. , 3.3, 1.4, 0.2],
[7. , 3.2, 4.7, 1.4],
[6.4, 3.2, 4.5, 1.5],
[6.9, 3.1, 4.9, 1.5],
[5.5, 2.3, 4. , 1.3],
[6.5, 2.8, 4.6, 1.5],
[5.7, 2.8, 4.5, 1.3],
[6.3, 3.3, 4.7, 1.6],
[4.9, 2.4, 3.3, 1. ],
[6.6, 2.9, 4.6, 1.3],
[5.2, 2.7, 3.9, 1.4],
[5. , 2. , 3.5, 1. ],
[5.9, 3. , 4.2, 1.5],
[6. , 2.2, 4. , 1. ],
[6.1, 2.9, 4.7, 1.4],
[5.6, 2.9, 3.6, 1.3],
[6.7, 3.1, 4.4, 1.4],
[5.6, 3. , 4.5, 1.5],
[5.8, 2.7, 4.1, 1. ],
[6.2, 2.2, 4.5, 1.5],
[5.6, 2.5, 3.9, 1.1],
[5.9, 3.2, 4.8, 1.8],
[6.1, 2.8, 4. , 1.3],
[6.3, 2.5, 4.9, 1.5],
[6.1, 2.8, 4.7, 1.2],
[6.4, 2.9, 4.3, 1.3],
[6.6, 3. , 4.4, 1.4],
[6.8, 2.8, 4.8, 1.4],
[6.7, 3. , 5. , 1.7],
[6. , 2.9, 4.5, 1.5],
[5.7, 2.6, 3.5, 1. ],
[5.5, 2.4, 3.8, 1.1],
[5.5, 2.4, 3.7, 1. ],
[5.8, 2.7, 3.9, 1.2],
[6. , 2.7, 5.1, 1.6],
[5.4, 3. , 4.5, 1.5],
[6. , 3.4, 4.5, 1.6],
[6.7, 3.1, 4.7, 1.5],
[6.3, 2.3, 4.4, 1.3],
[5.6, 3. , 4.1, 1.3],
[5.5, 2.5, 4. , 1.3],
[5.5, 2.6, 4.4, 1.2],
[6.1, 3. , 4.6, 1.4],
[5.8, 2.6, 4. , 1.2],
[5. , 2.3, 3.3, 1. ],
[5.6, 2.7, 4.2, 1.3],
[5.7, 3. , 4.2, 1.2],
[5.7, 2.9, 4.2, 1.3],
[6.2, 2.9, 4.3, 1.3],
[5.1, 2.5, 3. , 1.1],
[5.7, 2.8, 4.1, 1.3],
[6.3, 3.3, 6. , 2.5],
[5.8, 2.7, 5.1, 1.9],
[7.1, 3. , 5.9, 2.1],
[6.3, 2.9, 5.6, 1.8],
[6.5, 3. , 5.8, 2.2],
[7.6, 3. , 6.6, 2.1],
[4.9, 2.5, 4.5, 1.7],
[7.3, 2.9, 6.3, 1.8],
[6.7, 2.5, 5.8, 1.8],
[7.2, 3.6, 6.1, 2.5],
[6.5, 3.2, 5.1, 2. ],
[6.4, 2.7, 5.3, 1.9],
[6.8, 3. , 5.5, 2.1],
[5.7, 2.5, 5. , 2. ],
[5.8, 2.8, 5.1, 2.4],
[6.4, 3.2, 5.3, 2.3],
[6.5, 3. , 5.5, 1.8],
[7.7, 3.8, 6.7, 2.2],
[7.7, 2.6, 6.9, 2.3],
[6. , 2.2, 5. , 1.5],
[6.9, 3.2, 5.7, 2.3],
[5.6, 2.8, 4.9, 2. ],
[7.7, 2.8, 6.7, 2. ],
[6.3, 2.7, 4.9, 1.8],
[6.7, 3.3, 5.7, 2.1],
[7.2, 3.2, 6. , 1.8],
[6.2, 2.8, 4.8, 1.8],
[6.1, 3. , 4.9, 1.8],
[6.4, 2.8, 5.6, 2.1],
[7.2, 3. , 5.8, 1.6],
[7.4, 2.8, 6.1, 1.9],
[7.9, 3.8, 6.4, 2. ],
[6.4, 2.8, 5.6, 2.2],
[6.3, 2.8, 5.1, 1.5],
[6.1, 2.6, 5.6, 1.4],
[7.7, 3. , 6.1, 2.3],
[6.3, 3.4, 5.6, 2.4],
[6.4, 3.1, 5.5, 1.8],
[6. , 3. , 4.8, 1.8],
[6.9, 3.1, 5.4, 2.1],
[6.7, 3.1, 5.6, 2.4],
[6.9, 3.1, 5.1, 2.3],
[5.8, 2.7, 5.1, 1.9],
[6.8, 3.2, 5.9, 2.3],
[6.7, 3.3, 5.7, 2.5],
[6.7, 3. , 5.2, 2.3],
[6.3, 2.5, 5. , 1.9],
[6.5, 3. , 5.2, 2. ],
[6.2, 3.4, 5.4, 2.3],
[5.9, 3. , 5.1, 1.8]])
In [4]:
print (iris.data.shape) # (150, 4) #目标向量 iris.target print (iris.target.shape) # (150, )
(150, 4) (150,)
数据探索性分析(Exploratory Data Analysis,EDA)
探索性分析能够更好的了解数据集,检查数据集的特征和形状,验证脑海中的一些想法,对数据任务接下来的步骤有一个初步的想法。 这里我用了本地的iris数据集,方法是一样的没有区别,注意的就是需要看下一下数据的形式和加载过程中的header需不需要加载
2 数据预处理
通过特征提取,我们能得到未经处理的特征,这时的特征可能有以下问题:
不属于同一量纲:即特征的规格不一样,不能够放在一起比较。无量纲化可以解决这一问题。 信息冗余:对于某些定量特征,其包含的有效信息为区间划分,例如学习成绩,假若只关心“及格”或不“及格”,那么需要将定量的考分,转换成“1”和“0”表示及格和未及格。二值化可以解决这一问题。 定性特征不能直接使用:某些机器学习算法和模型只能接受定量特征的输入,那么需要将定性特征转换为定量特征。最简单的方式是为每一种定性值指定一个定量值,但是这种方式过于灵活,增加了调参的工作。通常使用哑编码的方式将定性特征转换为定量特征:假设有N种定性值,则将这一个特征扩展为N种特征,当原始特征值为第i种定性值时,第i个扩展特征赋值为1,其他扩展特征赋值为0。哑编码的方式相比直接指定的方式,不用增加调参的工作,对于线性模型来说,使用哑编码后的特征可达到非线性的效果。 存在缺失值:缺失值需要补充。 信息利用率低:不同的机器学习算法和模型对数据中信息的利用是不同的,之前提到在线性模型中,使用对定性特征哑编码可以达到非线性的效果。类似地,对定量变量多项式化,或者进行其他的转换,都能达到非线性的效果。
我们使用sklearn中的preproccessing库来进行数据预处理,可以覆盖以上问题的解决方案。
2.1 无量纲化 数据规范化
无量纲化使不同规格的数据转换到同一规格。常见的无量纲化方法有标准化和区间缩放法(我认为这句话是错误的)。标准化的前提是特征值服从正态分布,标准化后,其转换成标准正态分布。区间缩放法利用了边界值信息,将特征的取值区间缩放到某个特点的范围,例如[0, 1]等。
2.1.1 标准化 0均值标准化(Z-score standardization)
标准化需要计算特征的均值和标准差,公式表达为: 使用preproccessing库的StandardScaler类对数据进行标准化的代码如下:
In [ ]:
from sklearn.preprocessing import StandardScaler #标准化,返回值为标准化后的数据 StandardScaler().fit_transform(iris.data)
from sklearn.preprocessing import StandardScaler
标准化,返回值为标准化后的数据
StandardScaler().fit_transform(iris.data)
array([[ -9.00681170e-01, 1.03205722e+00, -1.34127240e+00,
-1.31297673e+00],
[ -1.14301691e+00, -1.24957601e-01, -1.34127240e+00,
-1.31297673e+00],
...
--- 例子就是如下,他是对列也就是同一特征下进行缩放,而不是对一个数据的不同特征之间(行)进行缩放 ---
from sklearn.preprocessing import StandardScaler data = np.array([[1,2],[3,4],[5,6]]).reshape(3,2) print data
[[1 2]
[3 4]
[5 6]]
print np.mean(data,axis=0) # 计算每一列均值 [ 3. 4.] print np.std(data,axis=0) # 计算每一列标准差 [ 1.63299316 1.63299316] print (data[0][0]-np.mean(data,axis=0)[0])/np.std(data,axis=0)[0] # 计算第一个元素的标准化后的值 -1.22474487139
区间缩放,返回值为缩放到[0, 1]区间的数据
StandardScaler().fit_transform(data)
array([[-1.22474487, -1.22474487],
[ 0. , 0. ],
[ 1.22474487, 1.22474487]])
2.1.2 区间缩放法(线性函数归一化(Min-Max scaling)) 区间缩放法的思路有多种,常见的一种为利用两个最值进行缩放,公式表达为:
使用preproccessing库的MinMaxScaler类对数据进行区间缩放的代码如下:
from sklearn.preprocessing import MinMaxScaler
区间缩放,返回值为缩放到[0, 1]区间的数据
MinMaxScaler().fit_transform(iris.data)
array([[ 0.22222222, 0.625 , 0.06779661, 0.04166667],
[ 0.16666667, 0.41666667, 0.06779661, 0.04166667],
[ 0.11111111, 0.5 , 0.05084746, 0.04166667],
...
--- 例子就是如下,他是对列也就是同一特征下进行缩放,而不是对一个数据的不同特征之间(行)进行缩放 ---
from sklearn.preprocessing import MinMaxScaler data = np.array([[1,2],[3,4],[5,6]]).reshape(3,2) print data
[[1 2]
[3 4]
[5 6]]
区间缩放,返回值为缩放到[0, 1]区间的数据
MinMaxScaler().fit_transform(data)
array([[ 0. , 0. ],
[ 0.5, 0.5],
[ 1. , 1. ]])
2.1.3 标准化与归一化的区别 标准化(规范化)与归一化的区别 (这是原作者的描述,我觉得有点问题) 简单来说,标准化是依照特征矩阵的列处理数据,其通过求z-score的方法,将样本的特征值转换到同一量纲下。 归一化是依照特征矩阵的行处理数据,其目的在于样本向量在点乘运算或其他核函数计算相似性时,拥有统一的标准,也就是说都转化为“单位向量”。规则为L2的归一化公式如下:
使用preproccessing库的Normalizer类对数据进行归一化的代码如下:
from sklearn.preprocessing import Normalizer
归一化,返回值为归一化后的数据
Normalizer().fit_transform(iris.data)
--- 例子就是如下,他是对一行数据的不同特征进行处理 ---
from sklearn.preprocessing import Normalizer import math data = np.array([[1,2],[3,4],[5,6]]).reshape(3,2) print data
[[1 2]
[3 4]
[5 6]]
print data[0][0]/math.sqrt((data[0][0])2 + (data[0][1])2) # 计算第一个元素L2正则化后的值 0.4472135955
规范化
Normalizer().fit_transform(data)
array([[ 0.4472136 , 0.89442719], [ 0.6 , 0.8 ], [ 0.6401844 , 0.76822128]])
这里比较搞混的一点是StandardScaler和Normalizer这个两个概念的问题(大家翻译上的误差导致信息非常混乱),其实StandardScaler就是尺寸缩放问题,即使同一特征下的数值在一定范围内浮动,如将数值所放在0-1范围内(MinMaxScaler), 或者将数据标准化成为均值为0,方差为1的数据(Z-score);而另一个就是Normalizer,将同一行数据的不同特征进行规范化,这样一个数据的不同特征具有相同的量纲或者表现力,比如说一个特征是身高,1.7m,体重为150斤,那么两个特征之间差距太大,身高这个特征变化根本无法起到决定作用(在体重这个变化特征下),毕竟大家怎么长都是一米多,但是体重差距一下子拉开20多是很正常的事
2.2 对定量特征二值化 定量特征二值化的核心在于设定一个阈值,大于阈值的赋值为1,小于等于阈值的赋值为0,公式表达如下:
使用preproccessing库的Binarizer类对数据进行二值化的代码如下:
from sklearn.preprocessing import Binarizer
二值化,阈值设置为3,返回值为二值化后的数据
Binarizer(threshold=3).fit_transform(iris.data)
2.3 对定性特征哑编码 由于IRIS数据集的特征皆为定量特征,故使用其目标值进行哑编码(实际上是不需要的)。使用preproccessing库的OneHotEncoder类对数据进行哑编码的代码如下:
from sklearn.preprocessing import OneHotEncoder
哑编码,对IRIS数据集的目标值,返回值为哑编码后的数据
OneHotEncoder().fit_transform(iris.target.reshape((-1,1)))
补充:但是从以一个角度来看,如果标签需要被量化,这个就很有用了, 如下图所示,将target这个标签转化成为数值才能进行机器学习
2.4 缺失值计算 由于IRIS数据集没有缺失值,故对数据集新增一个样本,4个特征均赋值为NaN,表示数据缺失。使用preproccessing库的Imputer类对数据进行缺失值计算的代码如下:
(原作者例子举的不好,我这里替换补充一下)
import numpy as np from sklearn.preprocessing import Imputer imp = Imputer(missing_values='NaN', strategy='mean', axis=0) # 使用特征的均值进行填充,其余还有使用众数填充等,只需要把mean改成median即可 data = np.array([np.nan, 2, 6, np.nan, 7, 6]).reshape(3,2)
print data
[[ nan 2.]
[ 6. nan]
[ 7. 6.]]
print imp.fit_transform(data)
[[ 6.5 2. ]
[ 6. 4. ]
[ 7. 6. ]]
2.5 数据变换 常见的数据变换有基于多项式的、基于指数函数的、基于对数函数的。4个特征,度为2的多项式转换公式如下:
使用preproccessing库的PolynomialFeatures类对数据进行多项式转换的代码如下:
from sklearn.preprocessing import PolynomialFeatures
多项式转换
参数degree为度,默认值为2
PolynomialFeatures().fit_transform(iris.data)
基于单变元函数的数据变换可以使用一个统一的方式完成,使用preproccessing库的FunctionTransformer对数据进行对数函数转换的代码如下:
from numpy import log1p from sklearn.preprocessing import FunctionTransformer
自定义转换函数为对数函数的数据变换
第一个参数是单变元函数
FunctionTransformer(log1p).fit_transform(iris.data)
2.6 回顾 类 功能 说明 StandardScaler 无量纲化 标准化(修正) 标准化,基于特征矩阵的列,将特征值转换至服从标准正态分布 MinMaxScaler 无量纲化 标准化(修正) 区间缩放,基于最大最小值,将特征值转换到[0, 1]区间上 Normalizer 归一化(规范化) 基于特征矩阵的行,将样本向量转换为“单位向量” Binarizer 二值化 基于给定阈值,将定量特征按阈值划分 OneHotEncoder 哑编码 将定性数据编码为定量数据 Imputer 缺失值计算 计算缺失值,缺失值可填充为均值等 PolynomialFeatures 多项式数据转换 多项式数据转换 FunctionTransformer 自定义单元数据转换 使用单变元的函数来转换数据
3 特征选择 这一段的主要是由特征选择–scikit-learn和原作者一块组合的,我负责补充
当数据预处理完成后,我们需要选择有意义的特征输入机器学习的算法和模型进行训练。通常来说,从两个方面考虑来选择特征:
特征是否发散:如果一个特征不发散,例如方差接近于0,也就是说样本在这个特征上基本上没有差异,这个特征对于样本的区分并没有什么用。 特征与目标的相关性:这点比较显见,与目标相关性高的特征,应当优选选择。除方差法外,本文介绍的其他方法均从相关性考虑。
根据特征选择的形式又可以将特征选择方法分为3种:
Filter:过滤法,按照发散性或者相关性对各个特征进行评分,设定阈值或者待选择阈值的个数,选择特征。
Wrapper:包装法,根据目标函数(通常是预测效果评分),每次选择若干特征,或者排除若干特征。 Embedded:嵌入法,先使用某些机器学习的算法和模型进行训练,得到各个特征的权值系数,根据系数从大到小选择特征。类似于Filter方法,但是是通过训练来确定特征的优劣。
我们使用sklearn中的feature_selection库来进行特征选择。
3.1 Filter 过滤法主要思想是:按照发散性或者相关性对各个特征进行对每一维的特征“打分”,即给每一维的特征赋予权重,这样的权重就代表着该维特征的重要性,设定阈值或者待选择阈值的个数,选择特征。
3.1.1 移除低方差的特征 (Removing features with low variance) 这应该是最简单的特征选择方法了:假设某特征的特征值只有0和1,并且在所有输入样本中,95%的实例的该特征取值都是1,那就可以认为这个特征作用不大。如果100%都是1,那这个特征就没意义了。当特征值都是离散型变量的时候这种方法才能用,如果是连续型变量,就需要将连续变量离散化之后才能用,而且实际当中,一般不太会有95%以上都取某个值的特征存在,所以这种方法虽然简单但是不太好用。可以把它作为特征选择的预处理,先去掉那些取值变化小的特征,然后再从接下来提到的的特征选择方法中选择合适的进行进一步的特征选择。
使用方差选择法,先要计算各个特征的方差,然后根据阈值,选择方差大于阈值的特征。使用feature_selection库的VarianceThreshold类来选择特征的代码如下:
from sklearn.feature_selection import VarianceThreshold
方差选择法,返回值为特征选择后的数据
参数threshold为方差的阈值
VarianceThreshold(threshold=3).fit_transform(iris.data)
3.1.2 单变量特征选择 (Univariate feature selection) 单变量特征选择能够对每一个特征进行测试,衡量该特征和响应变量之间的关系,根据得分扔掉不好的特征。对于回归和分类问题可以采用卡方检验等方式对特征进行测试。 方法简单,易于运行,易于理解,通常对于理解数据有较好的效果(但对特征优化、提高泛化能力来说不一定有效);这种方法有许多改进的版本、变种。
3.1.2.1 卡方(Chi2)检验 适用于:分类问题(y离散)
经典的卡方检验(原理及应用)是检验定性自变量对定性因变量的相关性。假设自变量有N种取值,因变量有M种取值,考虑自变量等于i且因变量等于j的样本频数的观察值与期望的差距,构建统计量,其中A为实际数,E为理论值:
这个统计量的含义简而言之就是自变量对因变量的相关性。用feature_selection库的SelectKBest类结合卡方检验来选择特征的代码如下:
from sklearn.feature_selection import SelectKBest from sklearn.feature_selection import chi2
选择K个最好的特征,返回选择特征后的数据
SelectKBest(chi2, k=2).fit_transform(iris.data, iris.target)
--- 例子 ---
from sklearn.datasets import load_iris from sklearn.feature_selection import SelectKBest from sklearn.feature_selection import chi2 iris = load_iris() X, y = iris.data, iris.target X.shape
(150, 4)
X_new = SelectKBest(chi2, k=2).fit_transform(X, y) X_new.shape
(150, 2) 直接砍掉了两个特征
3.1.2.2 Pearson相关系数 (Pearson Correlation) 适用于:回归问题(y连续)
尔森相关系数是一种最简单的,能帮助理解特征和响应变量之间关系的方法,该方法衡量的是变量之间的线性相关性,结果的取值区间为[-1,1],-1表示完全的负相关,+1表示完全的正相关,0表示没有线性相关。
Pearson Correlation速度快、易于计算,经常在拿到数据(经过清洗和特征提取之后的)之后第一时间就执行。Scipy的 pearsonr 方法能够同时计算 相关系数 和p-value.
import numpy as np from scipy.stats import pearsonr np.random.seed(0) size = 300 x = np.random.normal(0, 1, size)
pearsonr(x, y)的输入为特征矩阵和目标向量
np.random.normal(0, 1, 100) 创建100个均值为0,方差为1的高斯随机数
print("Lower noise", pearsonr(x, x + np.random.normal(0, 1, size))) print("Higher noise", pearsonr(x, x + np.random.normal(0, 10, size)))
输出为二元组(sorce, p-value)的数组
Lower noise (0.71824836862138386, 7.3240173129992273e-49) Higher noise (0.057964292079338148, 0.31700993885324746)
这个例子中,我们比较了变量在加入噪音之前和之后的差异。当噪音比较小的时候,相关性很强,p-value很低。至于什么p-vlaue请看喵喵的回答,个人觉得很清晰
Scikit-learn提供的 f_regrssion 方法能够批量计算特征的f_score和p-value,非常方便,参考sklearn的 pipeline
Pearson相关系数的一个明显缺陷是,作为特征排序机制,他只对线性关系敏感。如果关系是非线性的,即便两个变量具有一一对应的关系,Pearson相关性也可能会接近0。例如:
x = np.random.uniform(-1, 1, 100000) print pearsonr(x, x**2)[0] -0.00230804707612
更多类似的例子参考 sample plots 。另外,如果仅仅根据相关系数这个值来判断的话,有时候会具有很强的误导性,如 Anscombe’s quartet ,最好把数据可视化出来,以免得出错误的结论。
3.1.3 互信息和最大信息系数 (Mutual information and maximal information coefficient (MIC) 经典的互信息(互信息为随机变量X与Y之间的互信息I(X;Y)I(X;Y)为单个事件之间互信息的数学期望)也是评价定性自变量对定性因变量的相关性的,互信息计算公式如下:
互信息直接用于特征选择其实不是太方便:1、它不属于度量方式,也没有办法归一化,在不同数据及上的结果无法做比较;2、对于连续变量的计算不是很方便(X和Y都是集合,x,y都是离散的取值),通常变量需要先离散化,而互信息的结果对离散化的方式很敏感。
最大信息系数克服了这两个问题。它首先寻找一种最优的离散化方式,然后把互信息取值转换成一种度量方式,取值区间在[0,1]。 minepy 提供了MIC功能。
反过头来看 y=x^2 这个例子,MIC算出来的互信息值为1(最大的取值)。
为了处理定量数据,最大信息系数法被提出,使用feature_selection库的SelectKBest类结合最大信息系数法来选择特征的代码如下:
from sklearn.feature_selection import SelectKBest from minepy import MINE
由于MINE的设计不是函数式的,定义mic方法将其为函数式的,返回一个二元组,二元组的第2项设置成固定的P值0.5
def mic(x, y): m = MINE() m.compute_score(x, y) return (m.mic(), 0.5)
选择K个最好的特征,返回特征选择后的数据
SelectKBest(lambda X, Y: array(map(lambda x:mic(x, Y), X.T)).T, k=2).fit_transform(iris.data, iris.target)
3.1.4 距离相关系数 (Distance Correlation)
距离相关系数是为了克服Pearson相关系数的弱点而生的。在x和x^2这个例子中,即便Pearson相关系数是0,我们也不能断定这两个变量是独立的(有可能是非线性相关);但如果距离相关系数是0,那么我们就可以说这两个变量是独立的。
R的 energy 包里提供了距离相关系数的实现,另外这是 Python gist 的实现。
x = runif (1000, -1, 1) dcor(x, x**2) [1] 0.4943864
尽管有 MIC 和 距离相关系数 在了,但当变量之间的关系接近线性相关的时候,Pearson相关系数仍然是不可替代的。 第一,Pearson相关系数计算速度快,这在处理大规模数据的时候很重要。 第二,Pearson相关系数的取值区间是[-1,1],而MIC和距离相关系数都是[0,1]。这个特点使得Pearson相关系数能够表征更丰富的关系,符号表示关系的正负,绝对值能够表示强度。当然,Pearson相关性有效的前提是两个变量的变化关系是单调的。
3.2 Wrapper
包装法主要思想是:根据目标函数(通常是预测效果评分),每次选择若干特征,或者排除若干特征。也可以将特征子集的选择看作是一个搜索寻优问题,生成不同的组合,对组合进行评价,再与其他的组合进行比较。这样就将子集的选择看作是一个是一个优化问题,这里有很多的优化算法可以解决,尤其是一些启发式的优化算法,如GA,PSO,DE,ABC等,详见“优化算法—人工蜂群算法(ABC)”,“优化算法—粒子群算法(PSO)”。
3.2.1 递归特征消除法
递归消除特征法使用一个基模型来进行多轮训练,每轮训练后,消除若干权值系数的特征,再基于新的特征集进行下一轮训练。使用feature_selection库的RFE类来选择特征的代码如下:
from sklearn.feature_selection import RFE from sklearn.linear_model import LogisticRegression
递归特征消除法,返回特征选择后的数据
参数estimator为基模型
参数n_features_to_select为选择的特征个数
RFE(estimator=LogisticRegression(), n_features_to_select=2).fit_transform(iris.data, iris.target)
In [5]:
# --- 例子,使用不同的基算法对特征的评估效果不一,注意选择--- from sklearn.svm import SVC from sklearn.datasets import load_digits from sklearn.feature_selection import RFE from sklearn.linear_model import LogisticRegression iris = load_iris() x, y = iris.data, iris.target svc = SVC(kernel="linear", C=1) rfe = RFE(estimator=svc, n_features_to_select=2, step=1) rfe.fit(x, y) ranking = rfe.ranking_ print (ranking)
[3 2 1 1]
In [ ]:
# [3 2 1 1] 说明第三维度特征和第四维度特征排名前2 # 采用逻辑回归 ref2 = RFE(estimator=LogisticRegression(), n_features_to_select=2).fit(iris.data, iris.target) print (ref2.ranking_) # [3 1 2 1] 这里则选择认为第二维和第四维特征重要
3.3 Embedded
嵌入法主要思想是:使用某些机器学习的算法和模型进行训练,得到各个特征的权值系数,根据系数从大到小选择特征。类似于Filter方法,但是是通过训练来确定特征的优劣。其实是讲在确定模型的过程中,挑选出那些对模型的训练有重要意义的属性。
3.3.1 基于惩罚项的特征选择法
使用带惩罚项的基模型,除了筛选出特征外,同时也进行了降维。使用feature_selection库的SelectFromModel类结合带L1惩罚项的逻辑回归模型,来选择特征的代码如下:
from sklearn.feature_selection import SelectFromModel
from sklearn.linear_model import LogisticRegression
带L1惩罚项的逻辑回归作为基模型的特征选择
SelectFromModel(LogisticRegression(penalty="l1", C=0.1)).fit_transform(iris.data, iris.target)
In [7]:
# --- 例子,这里用支持向量机了 --- from sklearn.svm import LinearSVC from sklearn.datasets import load_iris from sklearn.feature_selection import SelectFromModel iris = load_iris() X, y = iris.data, iris.target X.shape # (150, 4) lsvc = LinearSVC(C=0.01, penalty="l1", dual=False).fit(X,y) # 这里的惩罚项是L1,别看做是11,这里L是小写 model = SelectFromModel(lsvc, prefit=True) X_new = model.transform(X) X_new[:3,:] # 选取的前三行看一下
C:\Users\ljt\Anaconda3\lib\site-packages\sklearn\svm\base.py:922: ConvergenceWarning: Liblinear failed to converge, increase the number of iterations. "the number of iterations.", ConvergenceWarning)
Out[7]:
array([[5.1, 3.5, 1.4],
[4.9, 3. , 1.4],
[4.7, 3.2, 1.3]])
In [ ]:
# L1惩罚项降维的原理在于保留多个对目标值具有同等相关性的特征中的一个,所以没选到的特征不代表不重要。故,可结合L2惩罚项来优化。具体操作为:若一个特征在L1中的权值为1,选择在L2中权值差别不大且在L1中权值为0的特征构成同类集合,将这一集合中的特征平分L1中的权值,故需要构建一个新的逻辑回归模型:
from sklearn.linear_model import LogisticRegression
class LR(LogisticRegression):
def __init__(self, threshold=0.01, dual=False, tol=1e-4, C=1.0,
fit_intercept=True, intercept_scaling=1, class_weight=None,
random_state=None, solver='liblinear', max_iter=100,
multi_class='ovr', verbose=0, warm_start=False, n_jobs=1):
#权值相近的阈值
self.threshold = threshold
LogisticRegression.__init__(self, penalty='l1', dual=dual, tol=tol, C=C,
fit_intercept=fit_intercept, intercept_scaling=intercept_scaling, class_weight=class_weight,
random_state=random_state, solver=solver, max_iter=max_iter,
multi_class=multi_class, verbose=verbose, warm_start=warm_start, n_jobs=n_jobs)
#使用同样的参数创建L2逻辑回归
self.l2 = LogisticRegression(penalty='l2', dual=dual, tol=tol, C=C, fit_intercept=fit_intercept, intercept_scaling=intercept_scaling, class_weight = class_weight, random_state=random_state, solver=solver, max_iter=max_iter, multi_class=multi_class, verbose=verbose, warm_start=warm_start, n_jobs=n_jobs)
def fit(self, X, y, sample_weight=None):
#训练L1逻辑回归
super(LR, self).fit(X, y, sample_weight=sample_weight)
self.coef_old_ = self.coef_.copy()
#训练L2逻辑回归
self.l2.fit(X, y, sample_weight=sample_weight)
cntOfRow, cntOfCol = self.coef_.shape
#权值系数矩阵的行数对应目标值的种类数目
for i in range(cntOfRow):
for j in range(cntOfCol):
coef = self.coef_[i][j]
#L1逻辑回归的权值系数不为0
if coef != 0:
idx = [j]
#对应在L2逻辑回归中的权值系数
coef1 = self.l2.coef_[i][j]
for k in range(cntOfCol):
coef2 = self.l2.coef_[i][k]
#在L2逻辑回归中,权值系数之差小于设定的阈值,且在L1中对应的权值为0
if abs(coef1-coef2) < self.threshold and j != k and self.coef_[i][k] == 0:
idx.append(k)
#计算这一类特征的权值系数均值
mean = coef / len(idx)
self.coef_[i][idx] = mean
return self
# 使用feature_selection库的SelectFromModel类结合带L1以及L2惩罚项,设定阈值来进行特征的筛选的逻辑回归模型,来选择特征的代码如下:
In [8]:
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets import load_boston
from sklearn.feature_selection import SelectFromModel
from sklearn.linear_model import LassoCV
# Load the boston dataset.
boston = load_boston()
X, y = boston.data, boston.target
# We use the base estimator LassoCV since the L1 norm promotes sparsity of features.
# 使用LassoCV来规范化使之稀疏化
clf = LassoCV()
# Set a minimum threshold of 0.25
sfm = SelectFromModel(clf, threshold=0.25)
n_features = sfm.fit_transform(X,y).shape[1] # 初始满足阈值后留下的特征
# Reset the threshold till the number of features equals two.
# Note that the attribute can be set directly instead of repeatedly
# fitting the metatransformer.
# 开始不断的进行阈值攀升,直到有特征不满足被砍掉
while n_features > 2:
sfm.threshold += 0.1
X_transform = sfm.transform(X)
n_features = X_transform.shape[1]
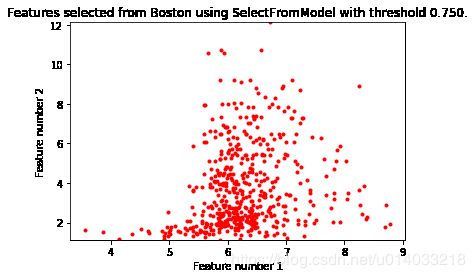
# Plot the selected two features from X.
plt.title(
"Features selected from Boston using SelectFromModel with "
"threshold %0.3f." % sfm.threshold)
feature1 = X_transform[:, 0]
feature2 = X_transform[:, 1]
plt.plot(feature1, feature2, 'r.')
plt.xlabel("Feature number 1")
plt.ylabel("Feature number 2")
plt.ylim([np.min(feature2), np.max(feature2)]) # ylim 设置y轴的范围
plt.show()
C:\Users\ljt\Anaconda3\lib\site-packages\sklearn\model_selection\_split.py:2053: FutureWarning: You should specify a value for 'cv' instead of relying on the default value. The default value will change from 3 to 5 in version 0.22. warnings.warn(CV_WARNING, FutureWarning)
3.3.2 基于树模型的特征选择法
树模型中GBDT也可用来作为基模型进行特征选择,使用feature_selection库的SelectFromModel类结合GBDT模型,来选择特征的代码如下:
In [10]:
from sklearn.feature_selection import SelectFromModel
from sklearn.ensemble import GradientBoostingClassifier
#GBDT作为基模型的特征选择
SelectFromModel(GradientBoostingClassifier()).fit_transform(iris.data, iris.target)
# --- 例子:这里用了随机森林了 ---
from sklearn.ensemble import RandomForestClassifier
from sklearn.datasets import load_iris
from sklearn.feature_selection import SelectFromModel
iris = load_iris()
X, y = iris.data, iris.target
X.shape
# (150, 4)
clf = RandomForestClassifier()
clf = clf.fit(X, y)
print (clf.feature_importances_ ) # 显示每一个特征的重要性指标,越大说明越重要,可以看出,第三第四两个特征比较重要
# [ 0.04505659 0.01056346 0.45428591 0.49009404]
model = SelectFromModel(clf, prefit=True)
X_new = model.transform(X)
X_new.shape
# (150, 2)
# 使用feature_importances_对boston数据的特征进行排序
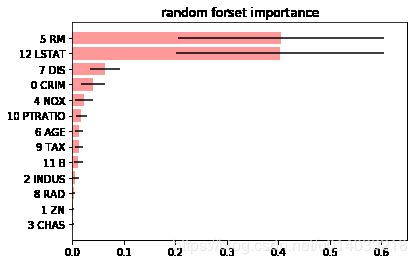
from sklearn.ensemble import RandomForestRegressor
x, y = boston.data, boston.target
feature_name = np.array(["%d %s"%(b,a) for a,b in zip(boston.feature_names,range(len(boston.feature_names)))])
rf = RandomForestRegressor(n_estimators=100, random_state=101).fit(x,y)
importance = np.mean([tree.feature_importances_ for tree in rf.estimators_], axis=0)
std = np.std([ tree.feature_importances_ for tree in rf.estimators_],axis=0)
indices = np.argsort(importance)
range_ = range(len(importance))
plt.figure()
plt.title("random forset importance")
plt.barh(range_, importance[indices],color='r',xerr=std[indices],alpha=0.4,align='center')
plt.yticks(range(len(importance)),feature_name[indices])
plt.ylim([-1,len(importance)])
plt.xlim([0.0,0.65])
plt.show()
C:\Users\ljt\Anaconda3\lib\site-packages\sklearn\ensemble\forest.py:246: FutureWarning: The default value of n_estimators will change from 10 in version 0.20 to 100 in 0.22. "10 in version 0.20 to 100 in 0.22.", FutureWarning)
[0.02108271 0.01199963 0.54213228 0.42478538]
In [ ]:
3.4 回顾 类 所属方式 说明 VarianceThreshold Filter 方差选择法 SelectKBest Filter 可选关联系数、卡方校验、最大信息系数作为得分计算的方法 RFE Wrapper 递归地训练基模型,将权值系数较小的特征从特征集合中消除 SelectFromModel Embedded 训练基模型,选择权值系数较高的特征 4 降维 当特征选择完成后,可以直接训练模型了,但是可能由于特征矩阵过大,导致计算量大,训练时间长的问题,因此降低特征矩阵维度也是必不可少的。常见的降维方法除了以上提到的基于L1惩罚项的模型以外,另外还有主成分分析法(PCA)和线性判别分析(LDA),线性判别分析本身也是一个分类模型。PCA和LDA有很多的相似点,其本质是要将原始的样本映射到维度更低的样本空间中,但是PCA和LDA的映射目标不一样:PCA是为了让映射后的样本具有最大的发散性;而LDA是为了让映射后的样本有最好的分类性能。所以说PCA是一种无监督的降维方法,而LDA是一种有监督的降维方法。 4.1 主成分分析法(PCA) 其他的一些参数的设置可以参考:scikit-learn中PCA的使用方法 使用decomposition库的PCA类选择特征的代码如下: from sklearn.decomposition import PCA #主成分分析法,返回降维后的数据 #参数n_components为主成分数目 PCA(n_components=2).fit_transform(iris.data)
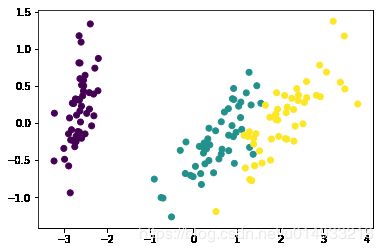
In [11]:
# --- 例子 --- from sklearn.decomposition import PCA,KernelPCA import pandas as pd import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import load_iris iris_data = load_iris() category = pd.Categorical(iris_data.target) # 将标签进行量化,就是说本来都是字符串啊,但是最后计算的时候都需要量化成1,2,3类等 pca_2c = PCA(n_components=2) # 使用PCA降到2维 #pca_2c = KernelPCA(n_components=2) x_pca_2c = pca_2c.fit_transform(iris_data.data) x_pca_2c.shape plt.scatter(x_pca_2c[:,0],x_pca_2c[:,1],c=category.codes) plt.show()
In [ ]:
4.2 线性判别分析法(LDA) 使用lda库的LDA类选择特征的代码如下: from sklearn.lda import LDA #线性判别分析法,返回降维后的数据 #参数n_components为降维后的维数 LDA(n_components=2).fit_transform(iris.data, iris.target)
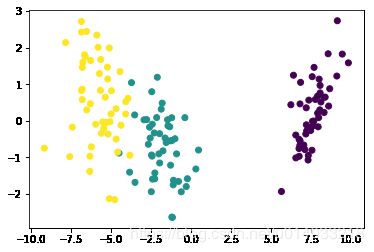
In [15]:
# --- 例子 --- # LDA相较于pca是有监督的,但不能用于回归分析 from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA import pandas as pd import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import load_iris iris_data = load_iris() category = pd.Categorical(iris_data.target) # 将标签进行量化,就是说本来都是字符串啊,但是最后计算的时候都需要量化成1,2,3类等 lda_2c = LDA(n_components=2) x_pca_2c = lda_2c.fit_transform(iris_data.data,iris_data.target) x_pca_2c.shape plt.scatter(x_pca_2c[:,0],x_pca_2c[:,1],c=category.codes) plt.show()