R语言编写决策树(rpart)CART ID3算法
决策树(decision tree)是一类常见的机器学习方法。以二分类任务为例,我们希望从给定训练数据集学得一个模型用以对新示例进行分类,这个把样本分类的任务,可看做对“当前样本属于正常吗?”这个问题的‘决策’或者‘判定’过程。顾名思义,决策树是基于树结构来进行决策的,这恰是人类在面临决策问题时一种很自然的处理机制。
常用的决策树算法:
- ID3 以信息增益作为分类标准
- CART 以基尼系数作为分类标准
算法的具体理论可以参考周志华的《机器学习》
数据预处理
loc<-"http://archive.ics.uci.edu/ml/machine-learning-databases/"
ds<-"breast-cancer-wisconsin/breast-cancer-wisconsin.data"
url<-paste(loc,ds,sep="")
data<-read.table(url,sep=",",header=F,na.strings="?")
names(data)<-c("编号","肿块厚度","肿块大小","肿块形状","边缘黏附","单个表皮细胞大小","细胞核大小","染色质","细胞核常规","有丝分裂","类别")
#print(data)
data$类别[data$类别==2]<-"良性"
data$类别[data$类别==4]<-"恶性"
#print(data)
data<-data[-1] #删除第一列元素#
#print(data)
set.seed(1234) #随机抽样设置种子
train<-sample(nrow(data),0.7*nrow(data)) #抽样函数,第一个参数为向量,nrow()返回行数 后面的是抽样参数前
tdata<-data[train,] #根据抽样参数列选择样本,都好逗号是选择行
vdata<-data[-train,] #删除抽样行采用的数据是UCI机器学习数据库里的威斯康星州乳腺癌数据集,通过对数据的分析,提取出关键特征来判断乳腺癌患病情况
tdata为训练数据集
vdata为测试数据集
建立决策树
library(rpart)
dtree<-rpart(类别~.,data=tdata,method="class", parms=list(split="information"))
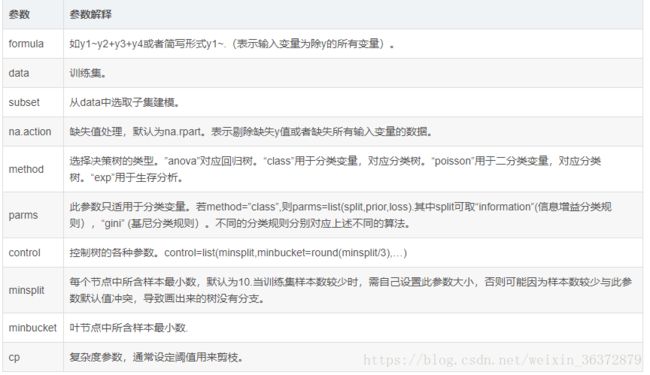
printcp(dtree)rpart()函数的格式:
rpart(formula,data,weights,subsets,na.action=na.rpart,method,parms,control…)
如果library报错,需要install数据包
使用ID3算法时候,split = “information” ,使用CART算法的时候, split = “gini”
决策树剪枝
剪枝(pruning)是决策树学习算法对付“过拟合”的主要手段,在决策树学习中,为了尽可能正确分类训练样本,结合划分过程将不断重复,有事会造成决策树分支过多,这时就可能因训练样本学得“太好了”,以致于把训练集自身的一些特点当做所有数据都具有的一般性质而导致过拟合。因此,可通过主动去掉一些分支来降低过拟合的风险。
print(dtree)可以看到,训练之后,采用了四个指标作为分支节点来建立决策树,而忽略了很多与乳腺癌不相关的特征
Variables actually used in tree construction:
[1] 细胞核大小 肿块大小 肿块厚度 肿块形状 在建立决策树之后通过printcp可以打印决策树的复杂性参数,观察树的误差等数据。
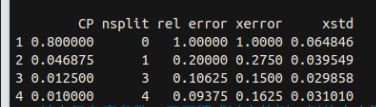
cp是参数复杂度(complexity parameter)作为控制树规模的惩罚因子,简而言之,就是cp越大,树分裂规模(nsplit)越小。输出参数(rel error)指示了当前分类模型树与空树之间的平均偏差比值。xerror为交叉验证误差,xstd为交叉验证误差的标准差。可以看到,当nsplit为3的时候,即有四个叶子结点的树,要比nsplit为4,即五个叶子结点的树的交叉误差要小。而决策树剪枝的目的就是为了得到更小交叉误差(xerror)的树。
使用prune()来剪枝,格式:prune(tree,cp,…)
从格式可以看出,按照的是cp值来进行剪枝,选择cp=0.0125来剪枝
tree<-prune(dtree,cp=0.0125)如果要写更加具有通用性的代码,可以自动选择xerror最小时候对应的cp值来剪枝
tree<-prune(dtree,cp=dtree$cptable[which.min(dtree$cptable[,"xerror"]),"CP"])
画出树图
格式:格式 rpart.plot(tree,type,fallen.leaves=T,branch,…)
| 参数 | 解释 |
|---|---|
| tree | 画图所用的树模型。 |
| type | 可取1,2,3,4.控制图形中节点的形式。 |
| fallen.leaves | fallen.leaves |
| branch | 控制图的外观。如branch=1,获得垂直树干的决策树。 |
opar<-par(no.readonly = T)
par(mfrow=c(1,2))
library(rpart.plot)
png(file = "./R/tree1.png")
rpart.plot(dtree,branch=1,type=2, fallen.leaves=T,cex=0.8, sub="剪枝前")
png(file = "./R/tree2.png")
rpart.plot(tree,branch=1, type=4,fallen.leaves=T,cex=0.8, sub="剪枝后")
par(opar)
dev.off()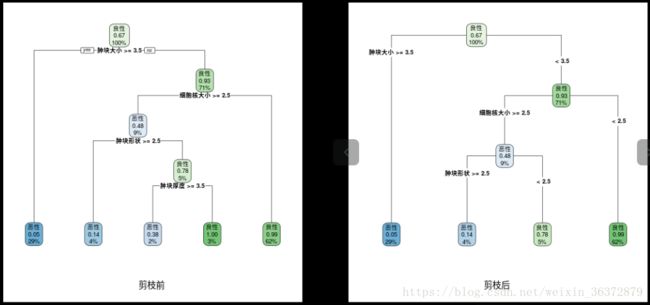
可以看到剪枝前后的对比图,剪枝前有五个叶节点(nplist = 4),剪枝之后(nplist = 3),剪枝之后,具有更小交叉验证误差。
利用测试集检测模型
格式 predict(fit,newdata,type,…)
predtree<-predict(tree,newdata=vdata,type="class") #利用预测集进行预测
table(vdata$类别,predtree,dnn=c("真实值","预测值")) #输出混淆矩阵| 真实值 | 恶性 | 良性 |
|---|---|---|
| 恶性 | 79 | 2 |
| 良性 | 7 | 122 |
从混淆矩阵可以看出此模型准确率为(79+122)/(79+2+7+122)=95.71%
使用基尼系数建立决策树的混淆矩阵:
| 真实值 | 恶性 | 良性 |
|---|---|---|
| 恶性 | 76 | 5 |
| 良性 | 7 | 122 |
准确率为:(76+122) / (81+129) = 94.29%