【转载 生信课程】03-序列比较-山东大学-生物信息学
序列比较
认识序列
FASTA 格式,第一行是一个大于号“>”开头,后面紧接注释信息
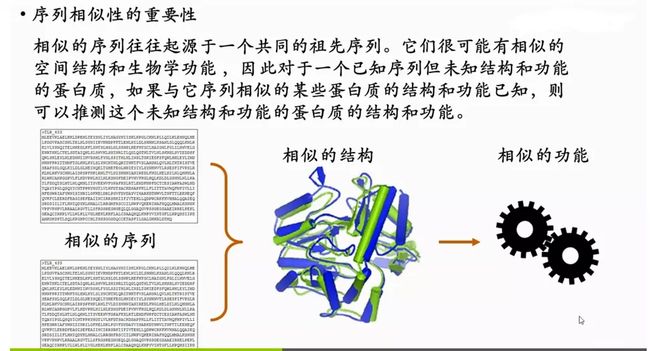
序列相似性
相似的序列》相似的结构》相似的功能
一致度
如果两个序列长度相同,那么它们的一致度可以暂时定义为它们对应位置上相同的残基数目占总长度的百分比。
例:上下相同的碱基为2,序列长度为4,则一致度为:2/4=2
相似度
如果两个序列长度相同,那么它们的相似度可以暂时定义为他们对应位置上相似的残基
与相同的残基的数目和占总长度的百分比。
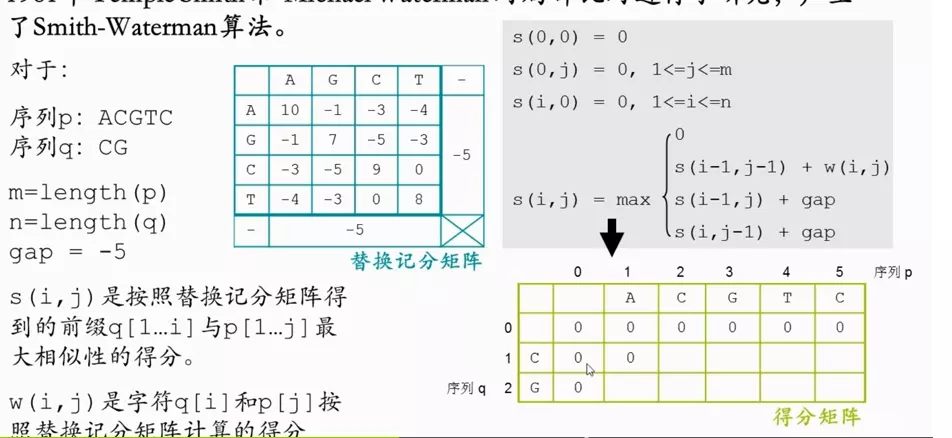
例:判断K 和 L 是否相似,需要替换记分矩阵
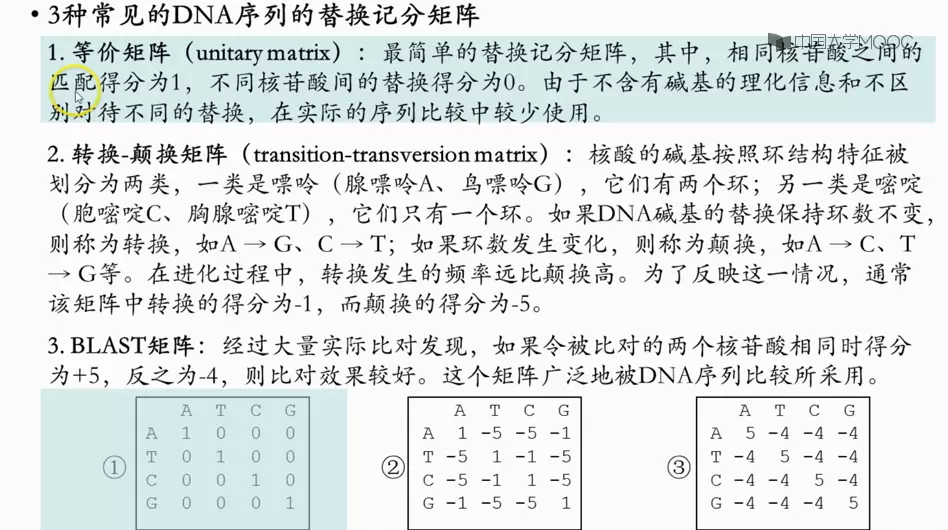
核酸序列的替换记分矩阵
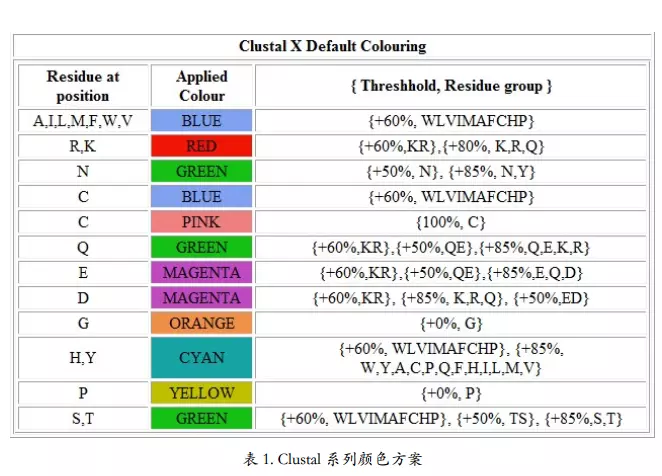
替换记分矩阵是反映残基之间相互替换率的矩阵。根据各氨基酸分值,可以知道各氨基酸的相似程度。
分类:
- DNA替换替换记分矩阵
- 蛋白质替换记分矩阵
DNA替换替换记分矩阵
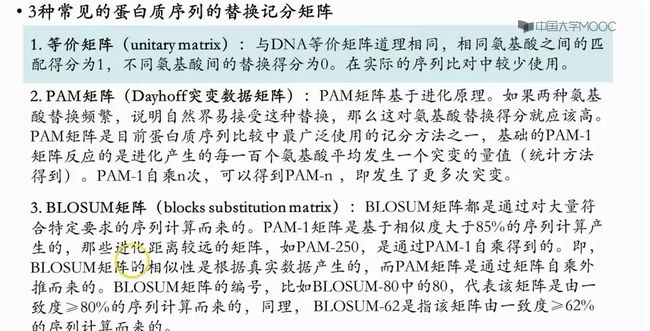
蛋白质序列的替换记分矩阵
- 等价矩阵
- PAM 矩阵
- BLOSUM 矩阵
BLOSUM 矩阵的相似性是根据真实数据产生的,而 PAM 矩阵是通过矩阵自乘外推而来的。
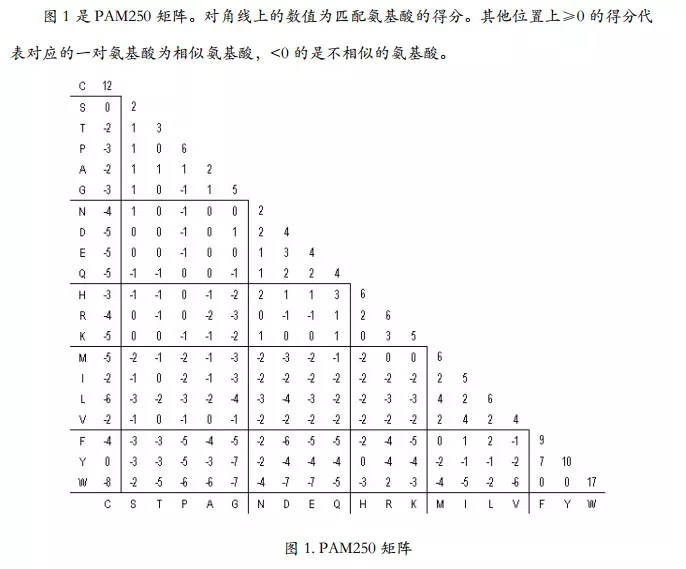
PAM 矩阵
我们需要根据要比较的序列之间的亲缘关系远近,来选择适合的 PAM 矩阵。如果序列亲缘关系远,也就是说序列间会有很多突变,那就选 PAM 后面跟一个大数字的矩阵。如果亲缘关系近,也就是突变比较少,
序列间大多数地方都是一样的,那就选 PAM 后面跟一个小数字的矩阵。
图 是 PAM250 矩阵。对角线上的数值为匹配氨基酸的得分。其他位置上≥0 的得分代
表对应的一对氨基酸为相似氨基酸,<0 的是不相似的氨基酸。
BLOSUM 矩阵
BLOSUM 后面跟一个小数字的矩阵适合用于比较相似度低的序列,也就是亲缘关系远的序列;而 BLOSUM 后面跟一个大数字的矩阵适合比较相似度高的序列,也就是亲缘关系近的序列。
图 是 BLOSUM 62 矩阵.样子和 PAM 矩阵差不多,但是里面的数值是不一样的。同样,≥0 的得分代表对应的一对氨基酸为相似氨基酸,<0 的是不相似的氨基酸。
image.png
PAM 和BLOSUM对应关系
PAM 后面的数体现的是序列的差异度,但不直接等于差异度,只是成对应关系而已;BLOSUM 后面的数体现是的序列的相似度并且直接等于相似度。所以我们看到,随着差异度的增大,适用的 PAM 矩阵后面的编号是增大的,而 BLOSUM 矩阵后面的编号是减小的。
PAM数值——对应差距程度 path 举例
BLOSUM 数值——对应相似程度 bin逼近
默认未知序列用 BLOSUM 62
image.png
image.png
蛋白质序列的替换记分矩阵
image.png
遗传密码矩阵
image.png
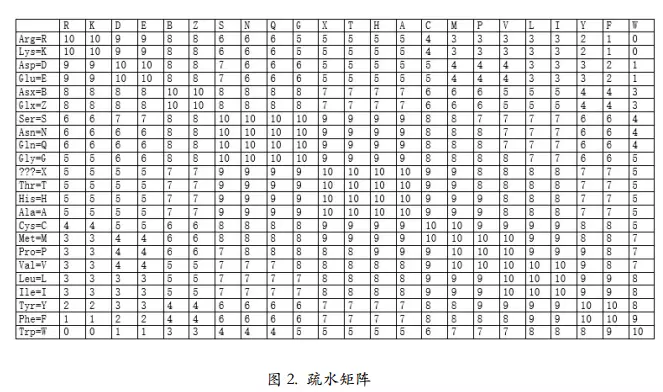
疏水矩阵
在这个矩阵里,氨基酸按照亲疏水性排列。前边是亲水的,后面是疏水的。
image.png
示例:
image.png
image.png
image.png
C=Cys
L=Leu
H=His
I=Ile
K=Lys
L和I得2分,相似
K和L得-2分,不相似
相似度=(2个相同+1个相似)/4个碱基长度=(2+1)/4=75%
如果两个序列的长度不相同怎样计算一致度与相似度?
CLHKA CIHL
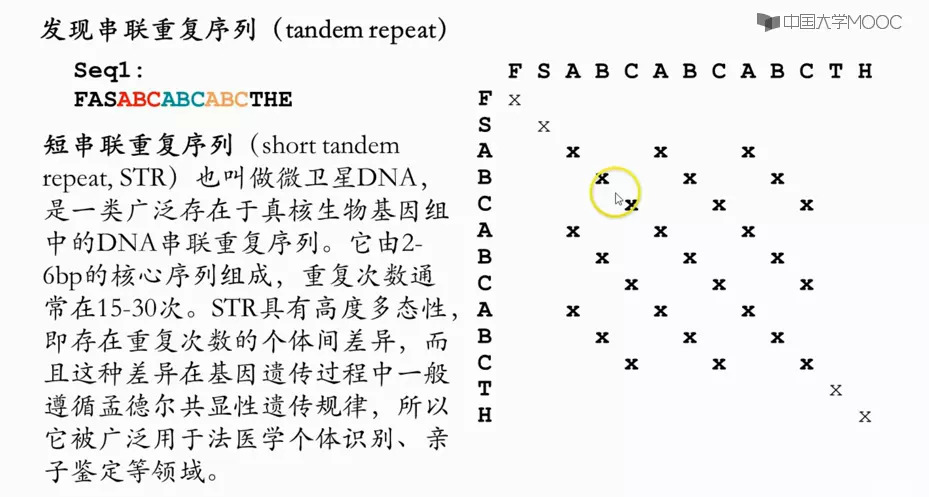
序列两两比较之打点法:打点法的用途
- 打点法——找重复出现的片段次数
image.png
相似的序列存在呈现长对角线
image.png
不相似的序列不出现对角线,呈散乱状
单序列打点
image.png
串联重复序列
image.png
序列两两比较之打点法:Dotlet 界面介绍
最常用的 Dotlet 软件
http://myhits.isb-sib.ch/cgi-bin/dotlet
示例:
- 一条序列打点
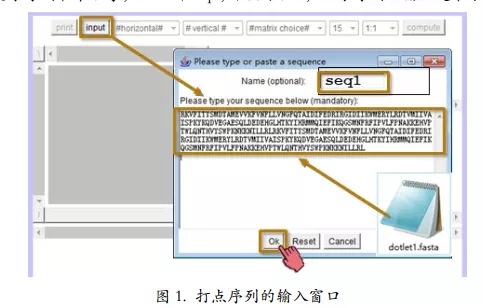
从input按钮输入蛋白质fasta序列,只能输入纯序列部分,不要输入“>标题行”。
他不能识别fasta的名字,需要手动输入英文名字
参数设置为,水平seq1 ,垂直seq1 ,只身打点。
选择BLOSUM62,
长度选择15,表示,比较前15个字母,与选择1的比较单元长度不一样而已,打点次数一次,不影响。
原始显示比例1:1
最后,提交
image.png
image.png
image.png
- 两条序列打点
水平seq2 ,垂直seq3
3,单条重复串联序列打点
水平和垂直选择seq4
5+0=-3
序列两两比较之序列比对法:什么是序列比对
序列比对就是运用特定的算法找出两个或者多个序列之间产生最大相似度
得分的空格插入和序列排列方案。
通过插入空位,让上下两行中尽可能多的一致的和相似的字符对在一起。
image.png
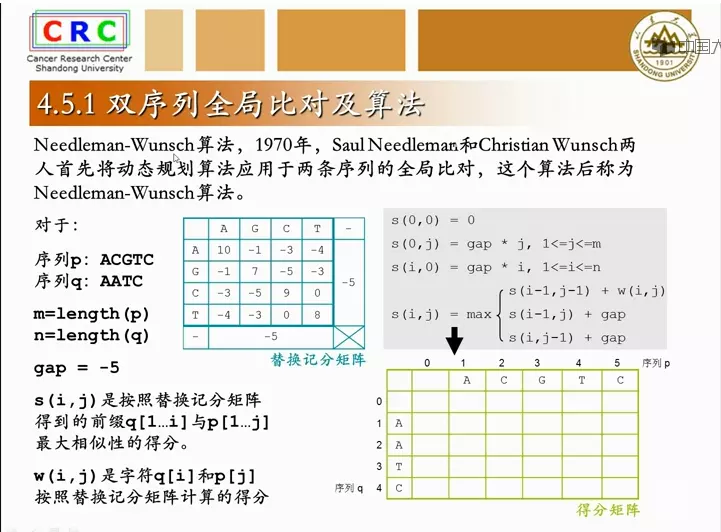
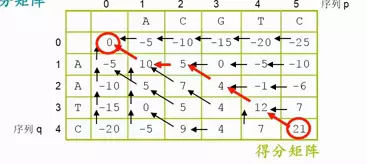
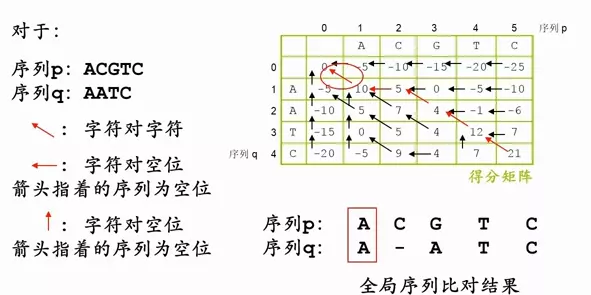
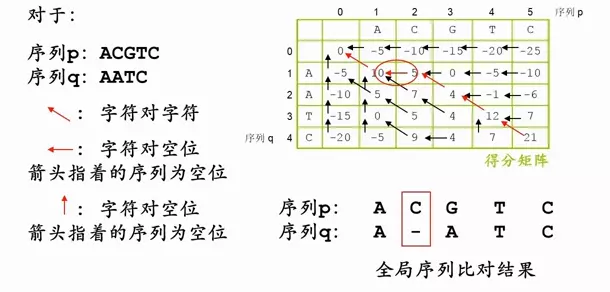
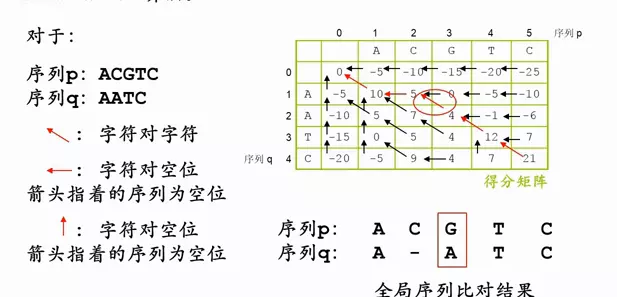
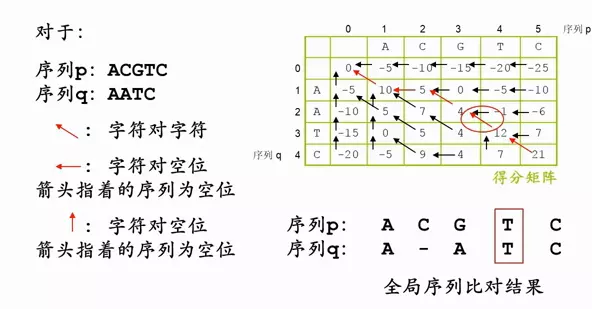
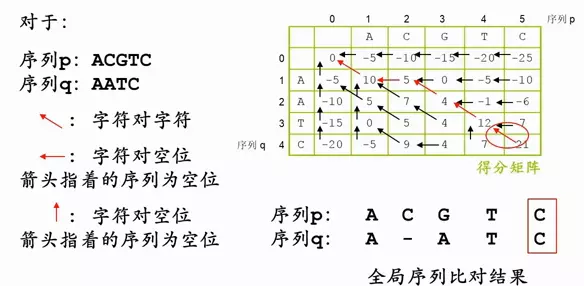
序列两两比较之序列比对法:双序列全局比对
image.png
开始赋值:
第0行,0列 ,
image.png
第0行,
考虑整行都为gap匹配的最差得分
image.png
第0列,
考虑整列都为gap匹配的最差得分
image.png
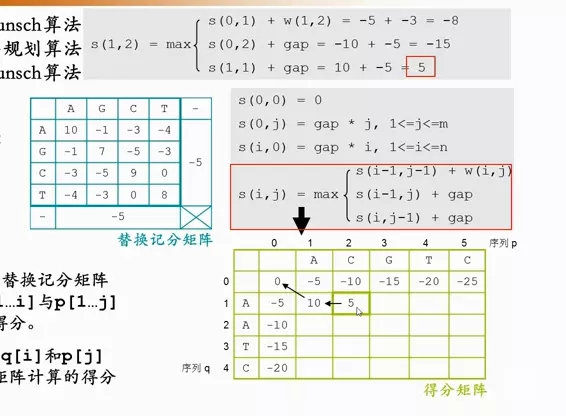
第s(1,1)
比较三个数值,找最大。
- 上方位的值s(i,j-1) + gap
- 左方位的值s(i-1,j) + gap
- 斜对角s(i-1,j-1) + 本位字母对字母的替换记分矩阵的值 w(i,j)
并用箭头记录得分的来源位置
image.png
第s(1,2)
image.png
第s(2,1)
当两个分值相同时,箭头指向两个方向
image.png
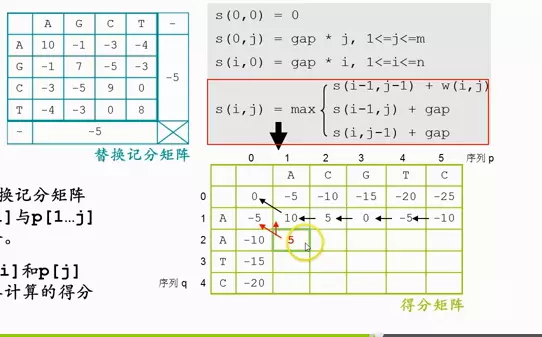
所有值的图
image.png
补充箭头,
第一行的数值来源于行的第一个,所有补上整行向左箭头,
第一列的数值来源于行的第一个,所有补上整列向上箭头,
image.png
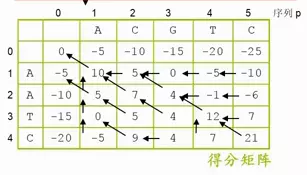
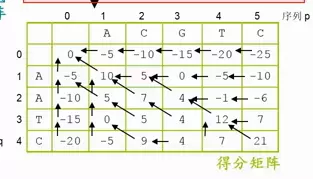
右下角的分数为全局比对的最终得分,从右下角出发,追溯指向最前的路径s(0,0),就是最优的比对序列。
image.png
根据箭头写出字符的比对结果
字符对字符
image.png
字符对空位
image.png
字符对字符
image.png
字符对字符
image.png
字符对字符
image.png
写到右下角,全局比对,结果出现,这样比分最高。每一个都是在上一步最优的情况下,得到下一步最优。
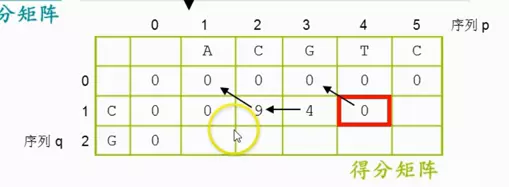
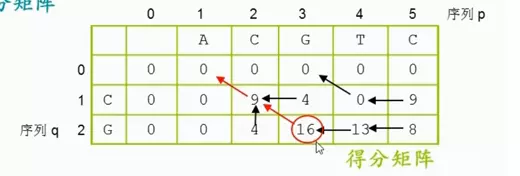
序列两两比较之序列比对法:双序列局部比对
image.png
s(1,1)
比较数值:
0
0+-3
0+-5
0+-5
最大为0分,由于0分来源于公式,不用写箭头。
image.png
0值来自于斜上角,有箭头
image.png
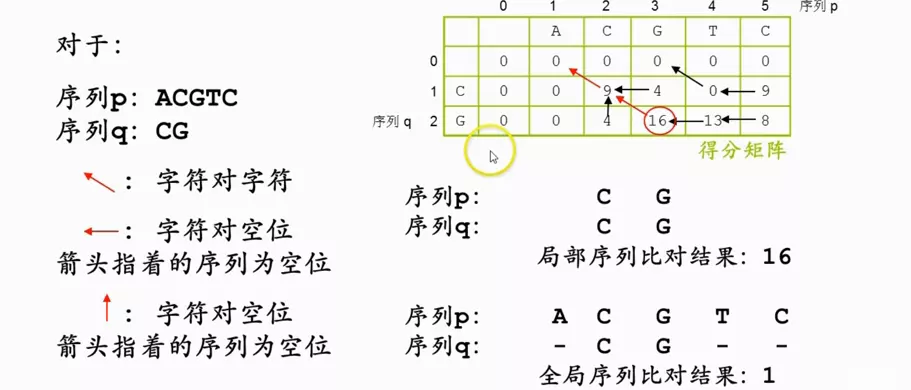
局部比对得分,最大值,在整个矩阵中找,最大值为16,从最大值开始,追溯到没有箭头为止。追溯箭头终止的位置也可以是得分矩阵中的任何一个位置。
image.png
局部比对的两端空位(没有箭头的地方)就全部忽略掉。
image.png
一致度和相似度
image.png
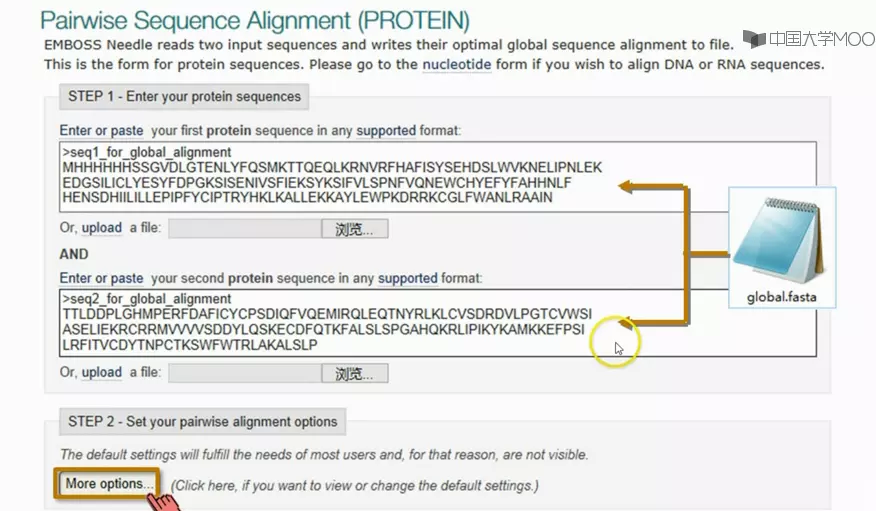
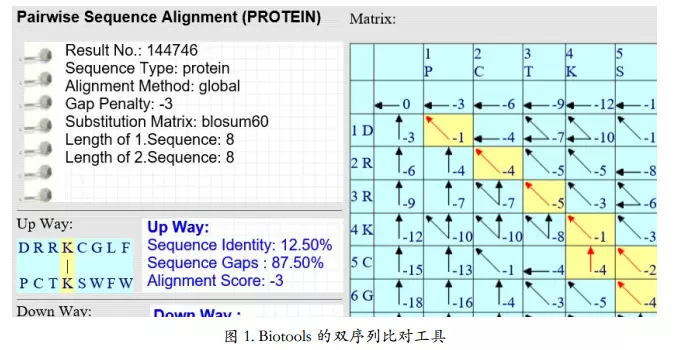
在线双序列比对工具:EMBL 全局双序列比对工具
使用率最高的是 EMBL 网站的双序列比对工具
http://www.ebi.ac.uk/Tools/psa
示例
- 全局比对》蛋白质比对工具
https://www.ebi.ac.uk/Tools/psa/emboss_needle/
image.png
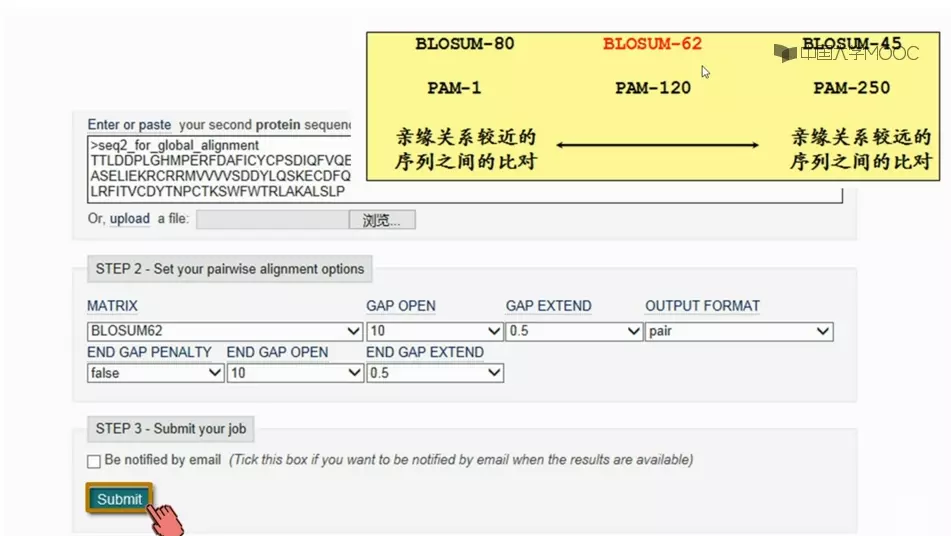
默认选择
BLOSUM-62
其他默认参数
image.png
image.png
结果页面:
# Length: 196
# Identity: 40/196 (20.4%)
# Similarity: 69/196 (35.2%)
# Gaps: 65/196 (33.2%)
# Score: 88.5
https://www.ebi.ac.uk/Tools/services/web/toolresult.ebi?jobId=emboss_needle-I20171230-070211-0300-21009296-p2m
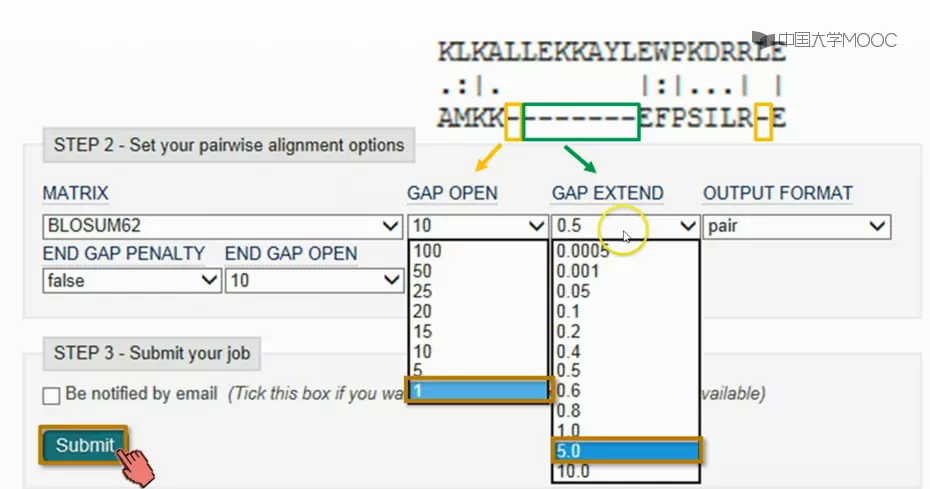
在线双序列比对工具:Gap 的类型及分值设置
gap 开头(GAP OPEN)
gap延长(GAP EXTEND)
gap 开头就是连续的一串 gap 里面打头的那一个,可以当它是队长。
gap 延长就是剩下的那些 gap,也就是队长后面跟着的小兵。
第一个gap 是 gap 开头,后面的都是 gap 延长。单独的一个 gap 按 gap 开头算。
gap 开头和 gap 延长可以分别定义不同的罚分。默认情况下,gap 开头罚分多,gap 延长罚分少。
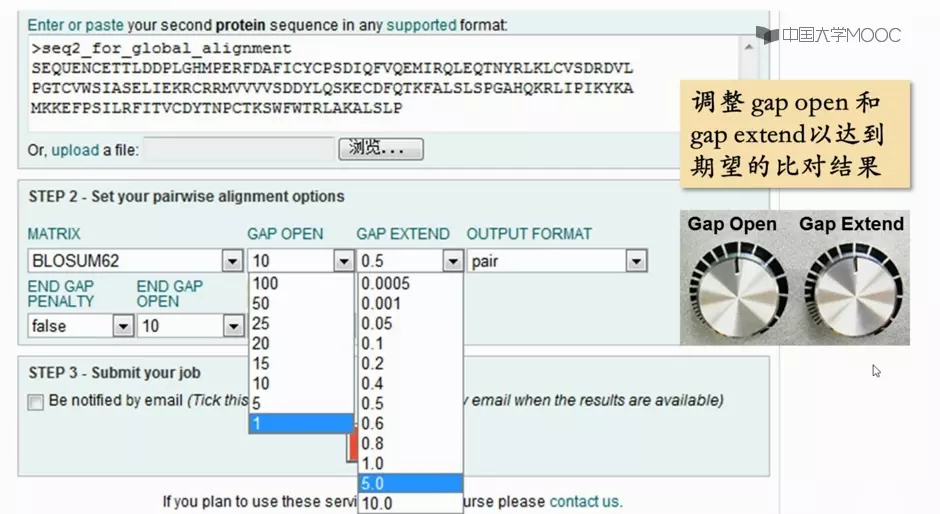
示例
这次我们反过来试试,让 gap 开头罚分少,让 gap 延长罚分多。比如 gap 开头选罚 1 分,gap 延长选罚 5 分,其他参数不变,再作一次看看结果发生了什么变化。
image.png
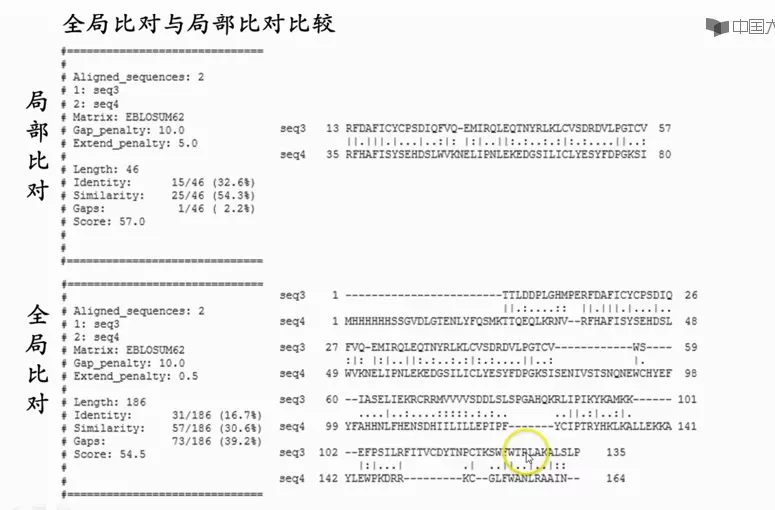
结果:
# Length: 201
# Identity: 46/201 (22.9%)
# Similarity: 78/201 (38.8%)
# Gaps: 75/201 (37.3%)
# Score: 210.0
image.png
总结:
在第一次做的结果里,也就是 gap 开头大,gap 延长小的时候,gap 很集中,有很多成
长串出现的 gap。
1)当 gap 开头大,gap 延长小的时候,说明在连
续的字母里插入一个 gap 打开一个缺口要付出很大的代价,因为 gap 开头罚分大。但是这个
缺口一旦打开了,也就是一旦有了第一个 gap,后面再接更多的 gap 就容易了,因为 gap 延
长罚分小。所以这种情况下,gap 都集中连成长串出现。
2)而反过来,当 gap 开头小,gap 延长大的时候,说明在连续的字母里插入一个 gap 打开
一个缺口很容易,并不需要付出太大代价,因为 gap 开头罚分小。但是想在第一个 gap 后面
再接一个 gap 就难了,因为 gap 延长罚分大。所以这种情况下很难有长串的 gap 出现,gap
每延长一个都要付出巨大代价。因此在第二次我们做的结果里(图 2-A)都是分散的 gap。
除了开头一段因两条序列长短不同而不得已出现的长串 gap 外,没有其他的长串 gap 了。
通过调整 gap 开头和 gap 延长,我们可以把序列比对做成我们期待的样子。
image.png
第一个例子,你知道要比对的两条序列很相似,是同源序列,所以它们的结构和功能也应该都差不多。其中一条序列的结构已知,另一条未知。你想把它们很好的比对在一起,用其中已知结构的序列做模板,来预测另一个序列的结构。
答案:选择分散的,gap开头小,延伸大。
另一例子,你知道要比对的两条序列绝大部分区域都很相似,但是其中一条序列的一个功能区在另一条序列中是缺失的。你想要通过序列比对把这个功能区找出来。这时候我们要怎么设置 gap 开头和 gap延长呢?
答案:选择长串的,gap开头大,延伸小
如果你对结果没有什么预期,那就请保持默认的参数。
除此之外,结尾的 gap 也可以划分出不同的种类并赋予不同的罚分,如果把 END GAPPENALTY 选成true,就可以设置结尾的 gap 罚分了。结尾 gap 不太常用,特别是在做亲缘关系较近的序列比对时,是否设置结尾 gap,比对结果差别不大。
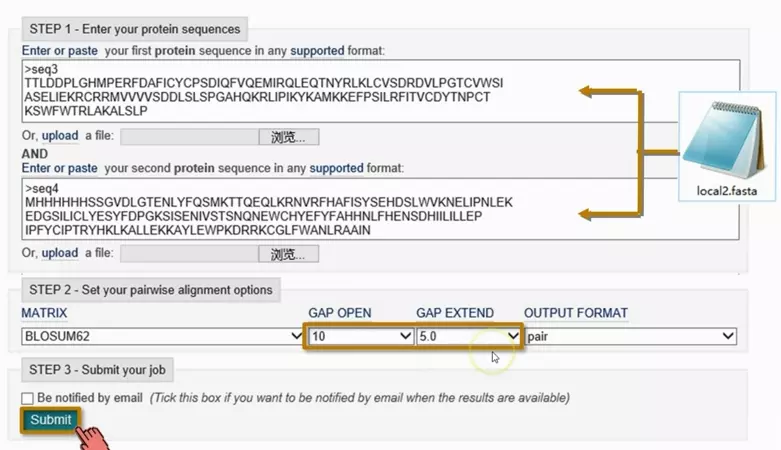
在线双序列比对工具:EMBL 局部双序列比对工具
https://www.ebi.ac.uk/Tools/psa/emboss_water/
示例
- 局部比对,默认参数
# Length: 130
# Identity: 103/130 (79.2%)
# Similarity: 103/130 (79.2%)
# Gaps: 27/130 (20.8%)
# Score: 551.0
image.png
将两条序列相近的序列,进行局部比对,gap开头和延长调到最大。
image.png
image.png
在线双序列比对工具:其他在线双序列比对工具
image.png
image.png
BLAST 搜索:BLAST 是怎么样工作的?
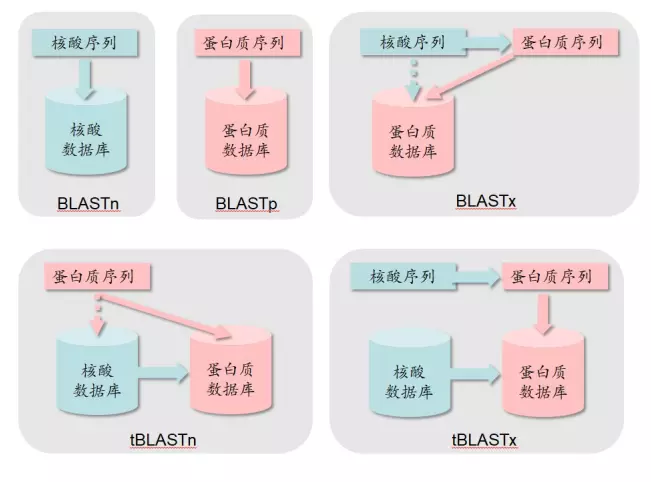
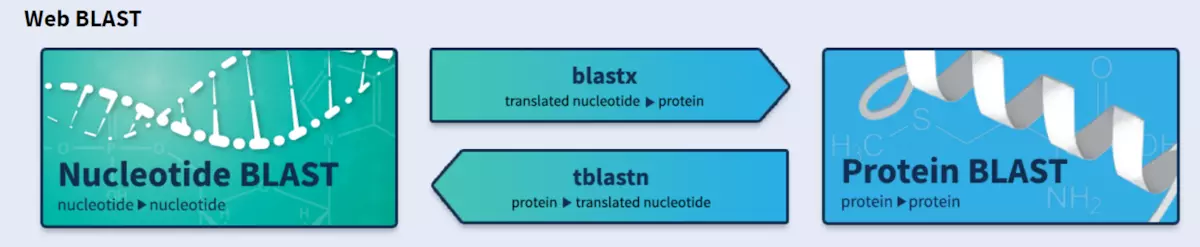
BLAST 搜索:BLAST 的种类
Blastn 核酸搜核酸
Blastp 蛋白搜蛋白
在无法得知翻译起始位点在情况下,翻译可能是从第一个碱基开始,三个三个的往后翻译,也可能是从第 2 个碱基开始,也可能从第 3 个碱基开始。另外还有可能是从这条链的互补链上开始,这样又有三个可能的开始位置,加起来一共会产生 6 条可能被翻译出来的蛋白质序列。这 6 条中有些是真实存在的,有些是不存在,但是谁真谁假我们无从知晓,所以 6 条序列都要到数据库中去搜索一下试试。
Blastx 通过核酸计算机翻译成注释或未注释的蛋白质后,搜索匹配蛋白质数据库。
tBlastn 通过蛋白质找核酸数据库,并将核酸序列计算机翻译成蛋白质后进行匹配蛋白质数据库。可以查找已注释或未注释的蛋白。
tBlastx 核酸序列计算机翻译成蛋白质后,搜索核酸序列数据库,然后再将核酸序列翻译成蛋白质后,搜索蛋白质数据库。
核酸翻译成蛋白后,找蛋白。
image.png
image.png
问答:要在核酸数据库查询一段与某DNA序列编码蛋白质最相似的序列,应选择:tBlastx
核酸翻译成蛋白后,找核酸。
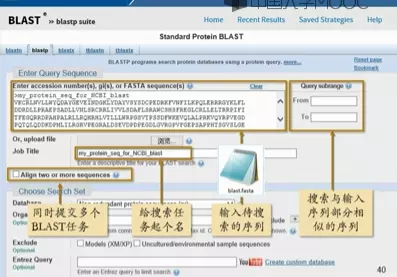
BLAST 搜索:NCBI BLASTp
Blast官网:https://blast.ncbi.nlm.nih.gov/Blast.cgi
image.png
示例
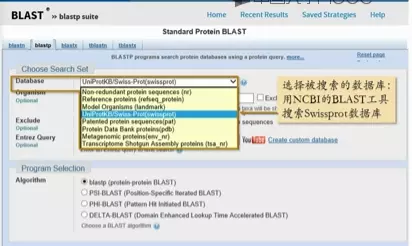
跨平台搜索Swissport数据库
image.png
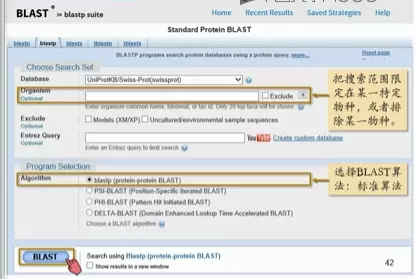
选择标准匹配
image.png
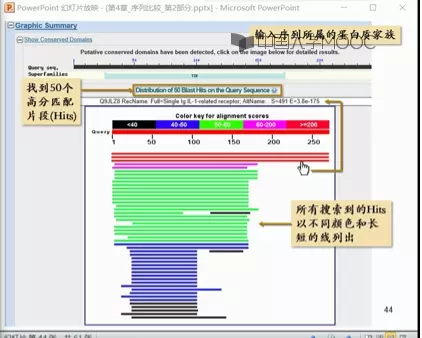
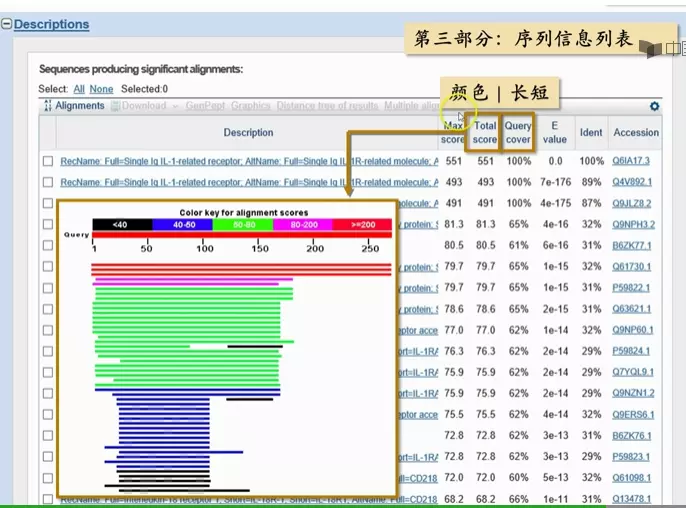
搜索结果
image.png
image.png
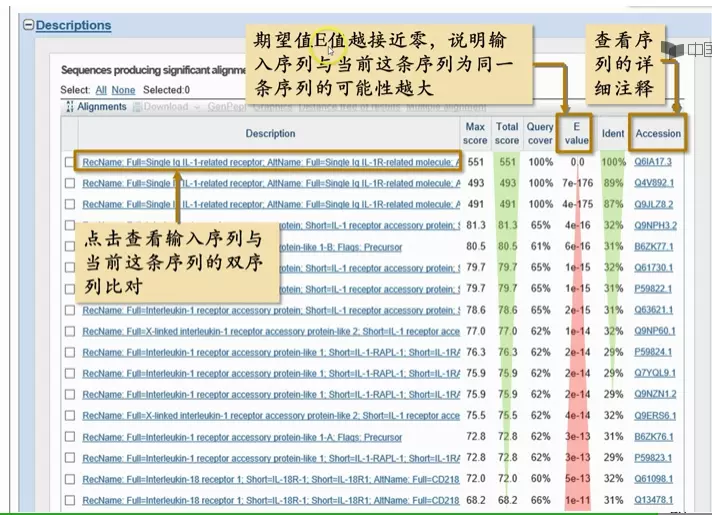
搜索序列按E值从小到达排列
image.png
BLAST 搜索:NCBI PSI-BLAST
搜索算法分类:
- Algorithm blastp (protein-protein BLAST) 标准Blast,搜索最少,最精确
- Algorithm PSI-BLAST (Position-Specific Iterated BLAST) 位点特异性迭代Blast,标准搜索不到的,可以使用此扩展搜索。
- Algorithm PHI-BLAST (Pattern Hit Initiated BLAST)
- Algorithm DELTA-BLAST (Domain Enhanced Lookup Time Accelerated BLAST
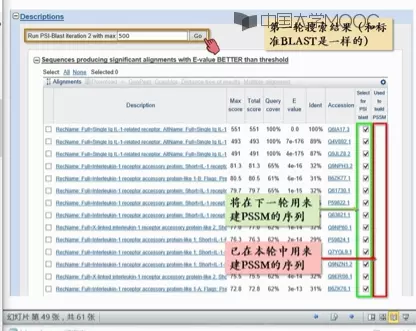
PSI-BLAST,根据上一次搜索结果,根据权重,搜索下一层结果。
image.png
第一轮搜索,的搜索结果,打勾,用于GO第二轮的搜索,第二轮新产生的用黄色标记(被标准blast漏掉的序列)。
image.png
BLAST 搜索:NCBI PHI-BLAST
PHI-BLAST 则是精准搜
索。PHI 是 Pattern-Hit Initiated 首字母缩写,中文是模式识别。PHI-BLAST 能找到与输入序
列相似的并符合某种特征模式的蛋白质序列。模式 Pattern 是对特征的描述。
image.png
模式范围
PSI-Blast 扩展匹配》标准Blast》PHI-Blast 模式匹配
image.png
BLAST 搜索:其他 BLAST
image.png
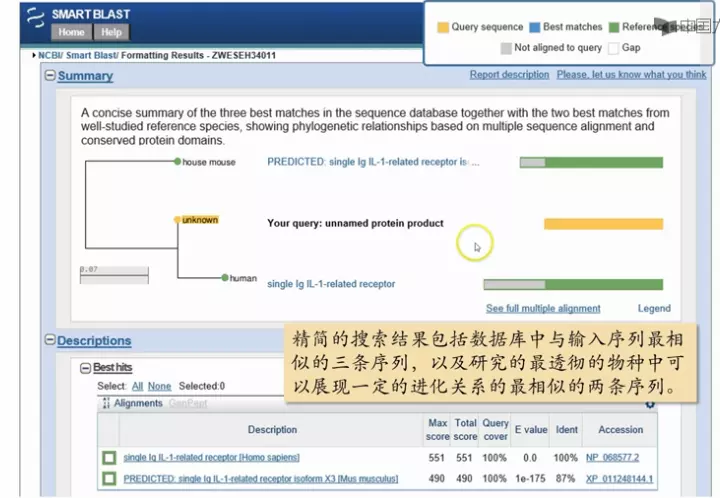
SMARTBLAST 聪明的Blast ,只需要输入序列就行。
https://blast.ncbi.nlm.nih.gov/smartblast/?LINK_LOC=BlastHomeLink
示例
image.png
在国外睡觉的时候,选择对应搜索引擎,如美国睡觉的时候,选择NCBI
image.png
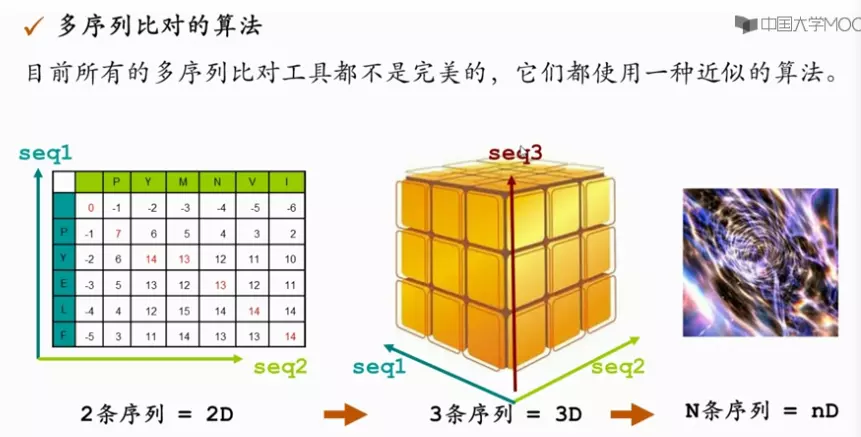
多序列比对介绍:用途和算法
多序列比对的作用
image.png
从多序列比对中看趋势
image.png
多序列比对介绍:注意事项
image.png
image.png
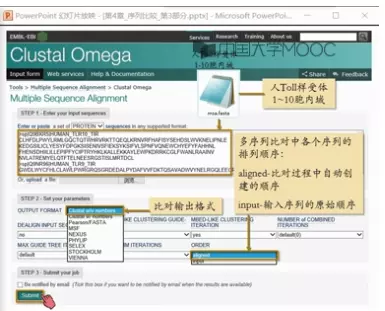
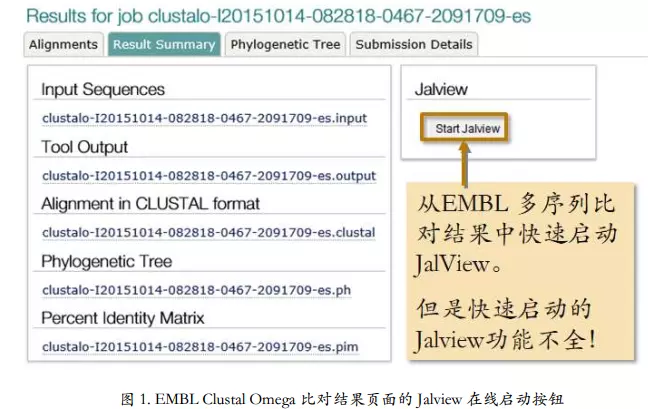
在线多序列比对工具:EMBL - Clustal Omega
image.png
EBI多序列比对网站
http://www.ebi.ac.uk/Tools/msa
示例
image.png
选择Download Aligment File进行比对
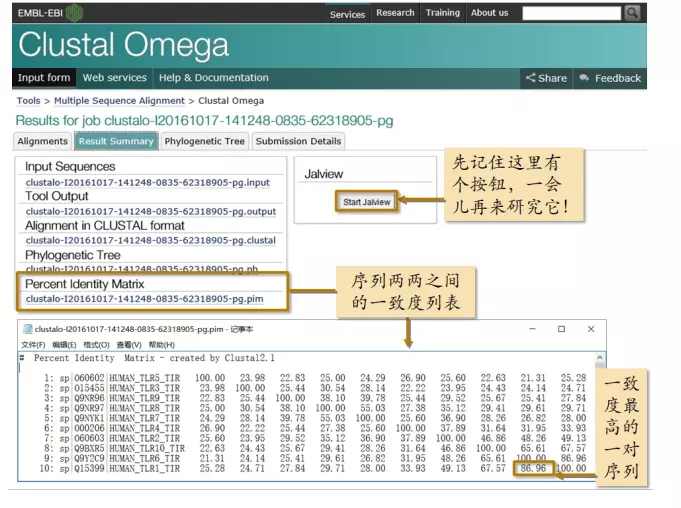
比对结果:
https://www.ebi.ac.uk/Tools/services/web/toolresult.ebi?jobId=clustalo-I20171230-133045-0811-8947437-p1m
image.png
文件下载
image.png
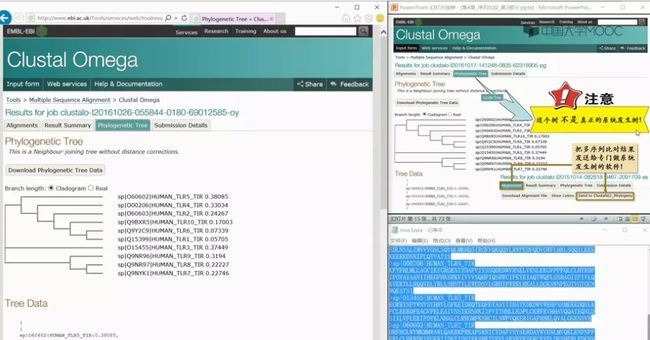
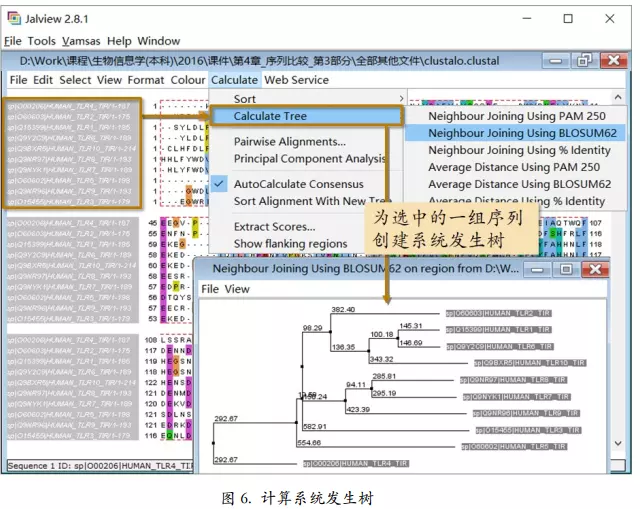
此生成的树,不能作为系统发生树,没有经过进行距离校正。
image.png
如果想要根据多序列比对结果构建系统发生树,可以在 Alignments 标签下,点击“Send toClustalW2_Phylogeny”链接,把做好的多序列比对发送给专门做系统发生树的工具。
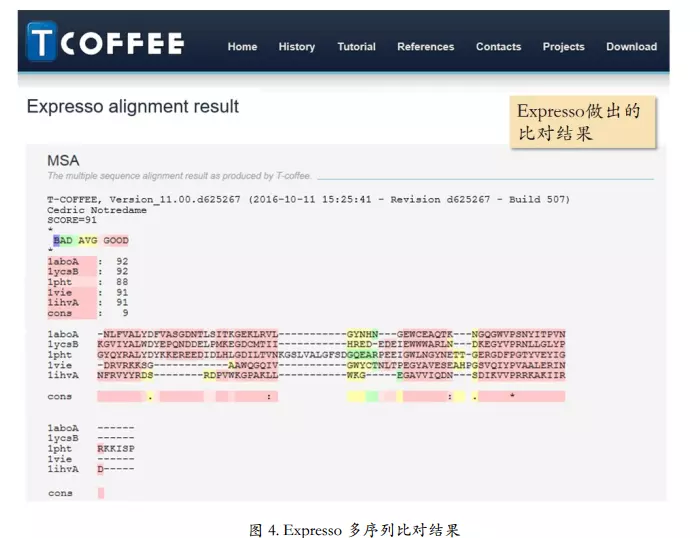
在线多序列比对工具:TCOFFEE - Expresso
官网:http://tcoffee.crg.cat
image.png
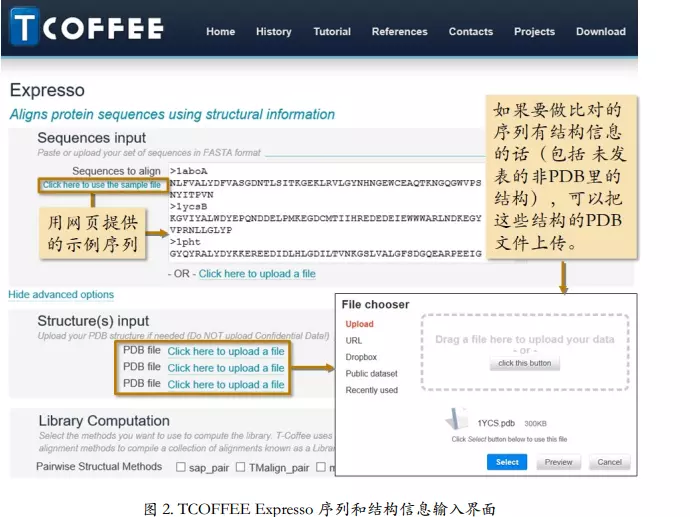
通过结构数据,辅助,提高比对结果准确度
image.png
示例
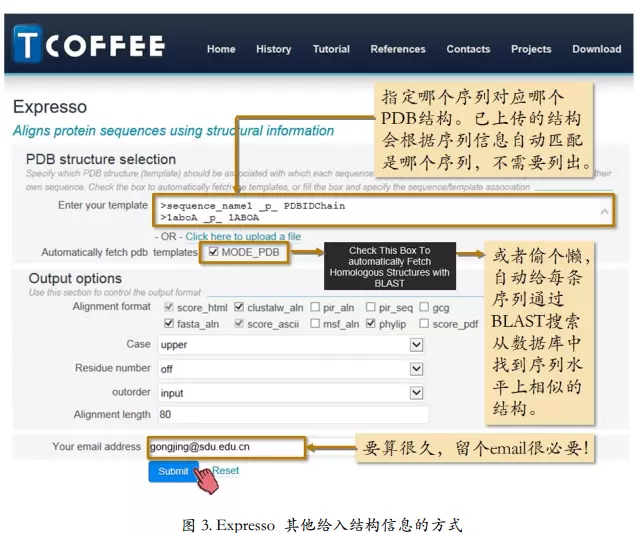
使用网站的示例序列,直接比对。
勾选自动搜索PDB数据库,填写邮箱等待结果。
Automatically fetch pdb templates MODE_PDB
image.png
image.png
TCOFFEE高质量比对结果
image.png
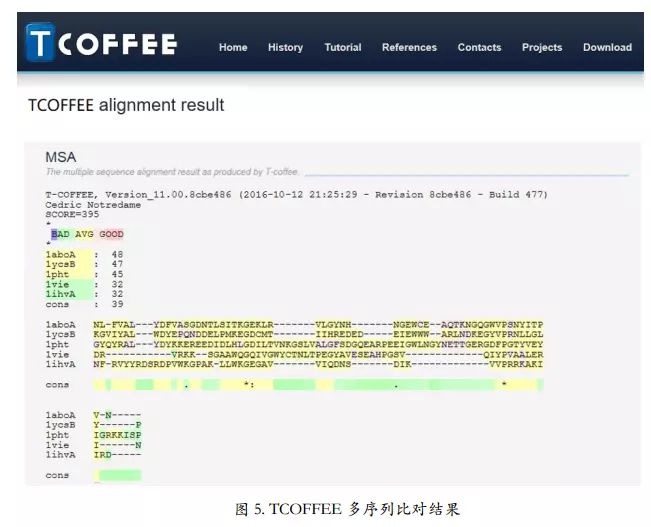
普通比对结果
image.png
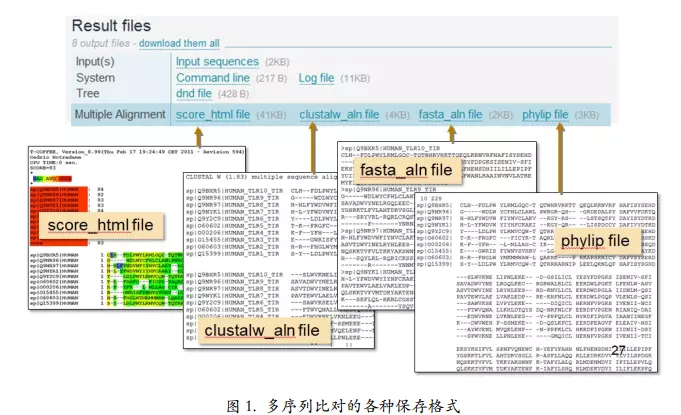
在线多序列比对工具:多序列比对的保存格式
image.png
fmtseq 工具(http://www.bioinformatics.org/JaMBW/1/2)
image.png
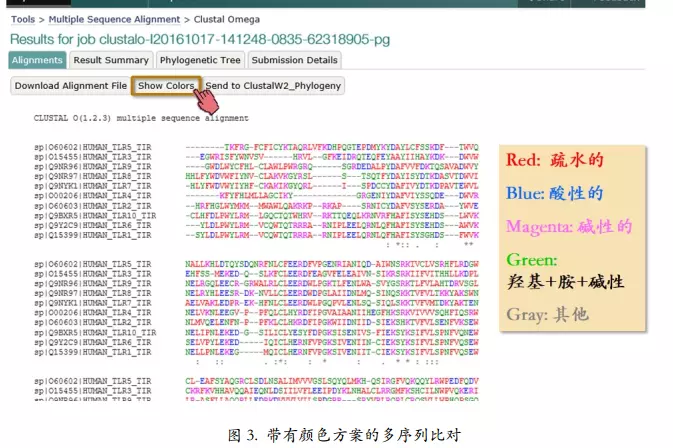
多序列比对的编辑和发布:Jalview 的介绍和操作
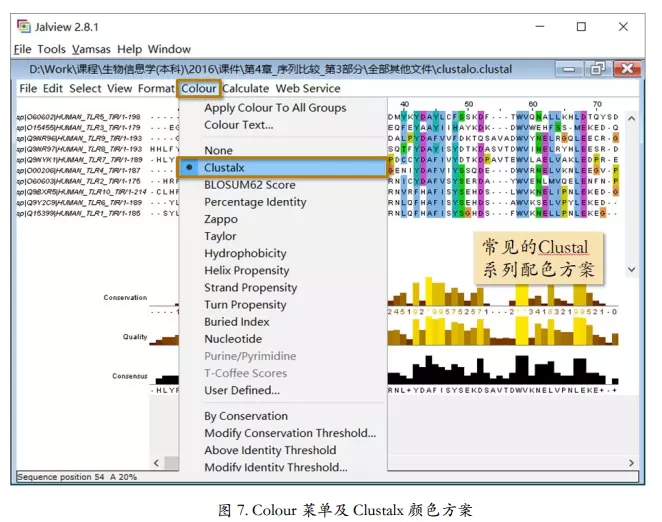
对比对结果进行美化,加颜色
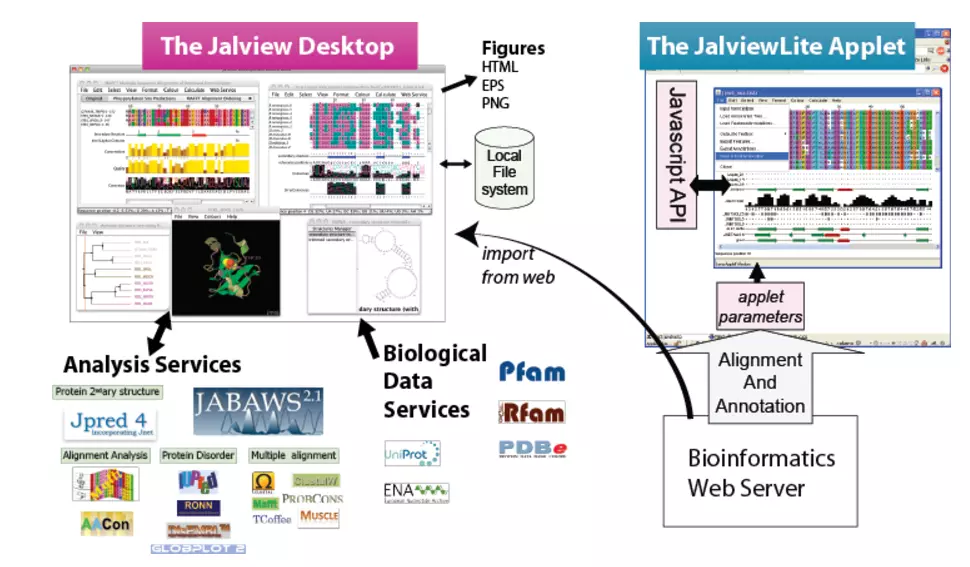
Jalview 官网(http://www.jalview.org)
image.png
全功能桌面版
image.png
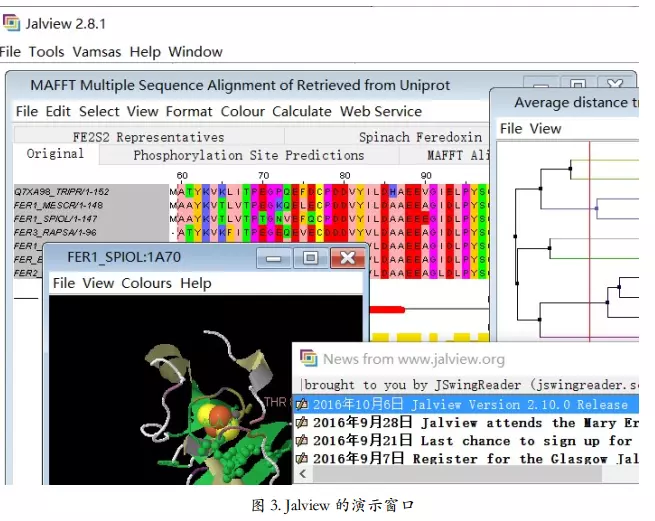
展示窗口
上传clust文件
image.png
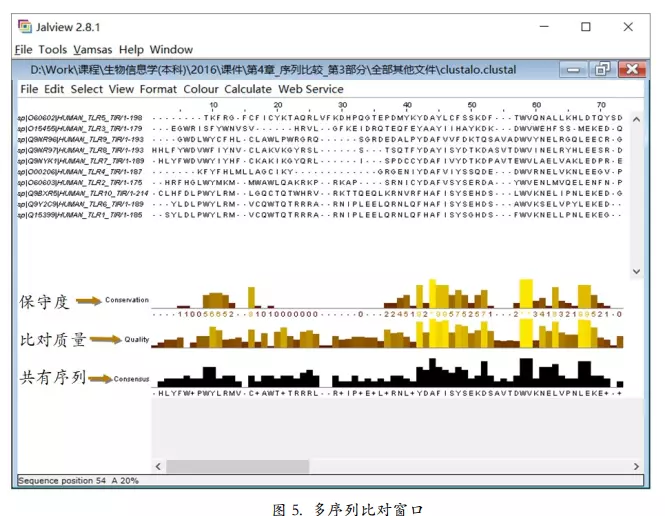
查看保守区序列
image.png
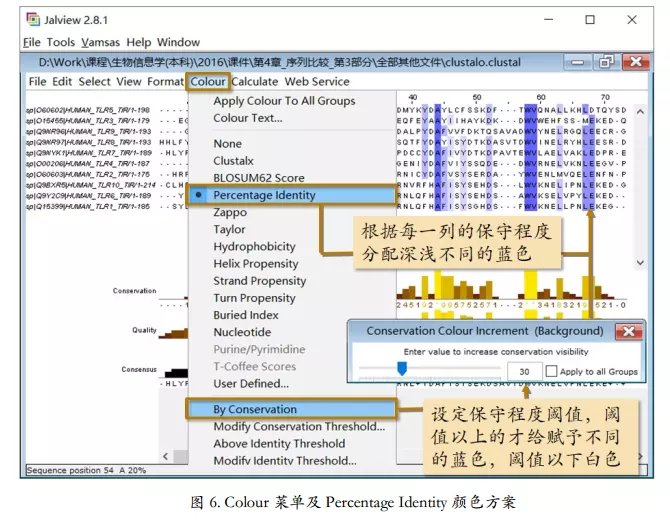
添加颜色方案
image.png
papper常用的方案
image.png
image.png
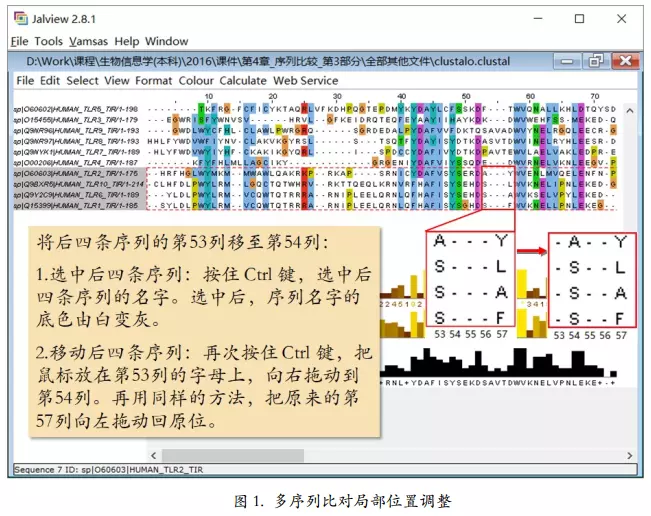
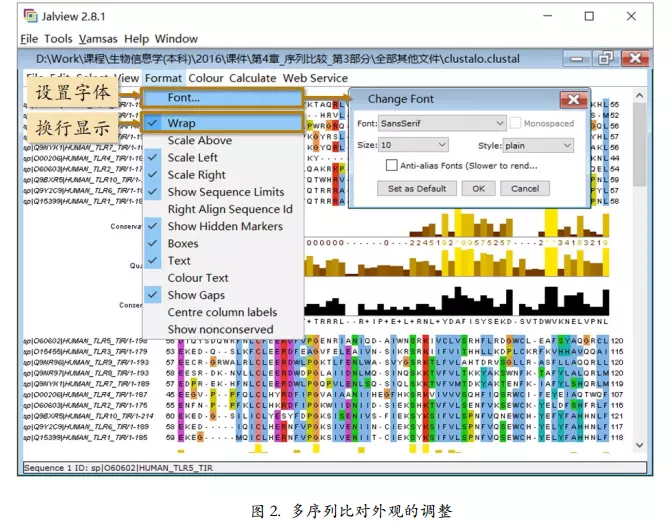
多序列比对的编辑和发布:Jalview 的编辑和发布
移动空位gap
image.png
换行
image.png
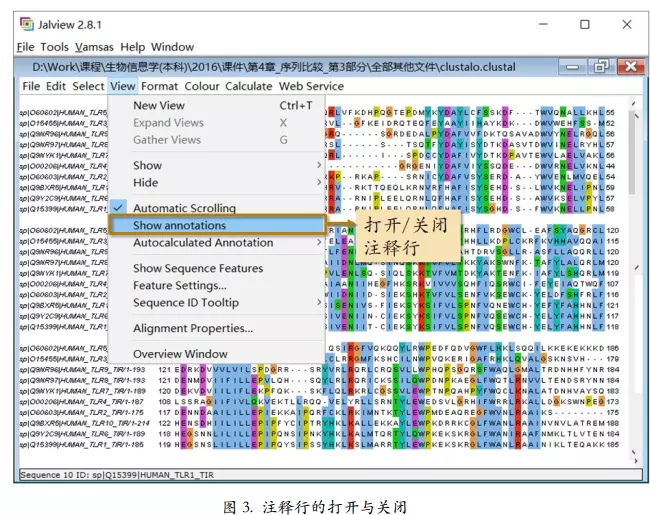
注释行的打开与关闭
image.png
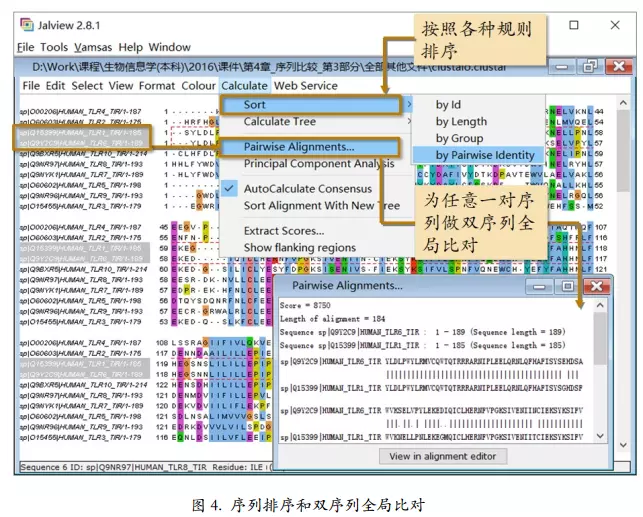
序列排序和双序列全局比对
image.png
计算系统发生树
image.png
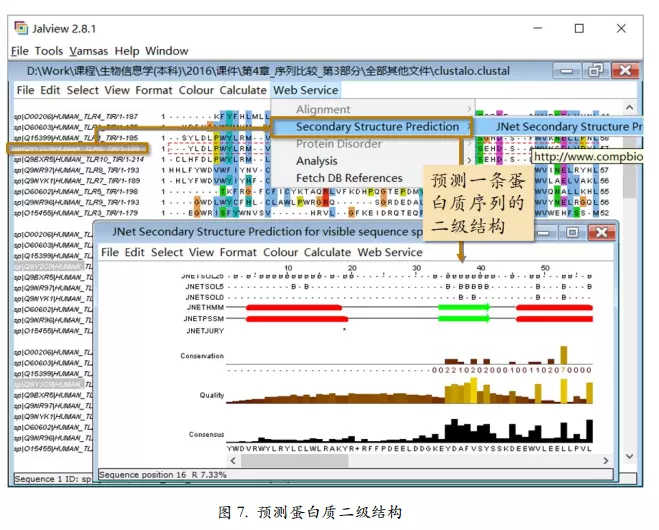
预测蛋白质二级结构
image.png
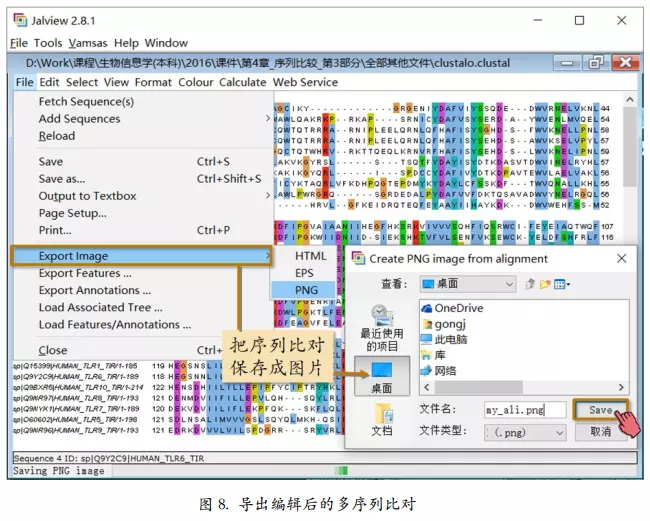
导出编辑后的多序列比对
image.png
多序列比对编辑工具
名称 网址 特点
JalView http://www.jalview.org JAVA,可嵌入网页
Boxshade http://www.ch.embnet.org/software/BOX_form.html 擅长黑白作图
ESPript http://espript.ibcp.fr/ESPript/ESPript 功能强大
MView http://bio-mview.sourceforge.net 擅长转换成 HTML 源码
发大文章彩图贵,黑白图免费
可以插入网页,可以网页打开,换行
寻找保守区域:序列标识图 WebLogo
做多序列比对,就是为了找保守区域,找* : 多的区域
“*”代表这一列残基完全相同;
“:”代表这一列残基或者相同或者相似;
“.”代表这一列残基有相似的但也有不相似的;
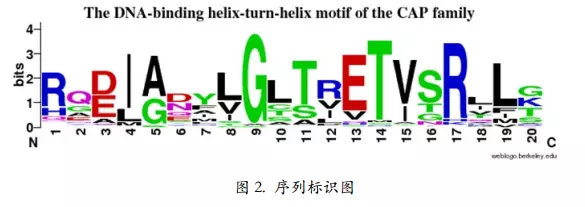
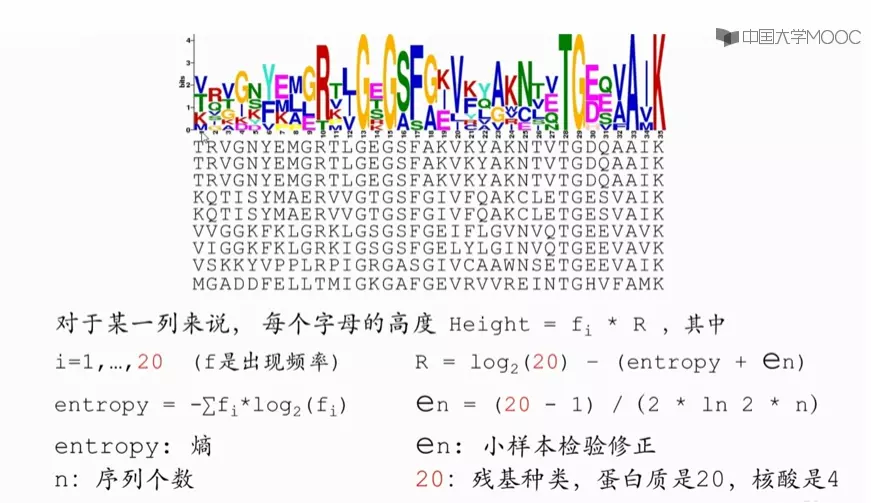
序列标识图就是序列的 logo,它是以图形的方式依次绘出序列比对中各个位置上出现的残基,每个位置上残基的累积可以反应出该位置上残基的一致性。每个残基对应图形字符的大小与残基在该位置上出现的频率成正比。 但图形字符的大小并不等于频率百分比,而是经过简单统计计算后转化的结果。
image.png
保守会看到单一高字母,和出现频率成正比。有熵值,杂变矮,
image.png
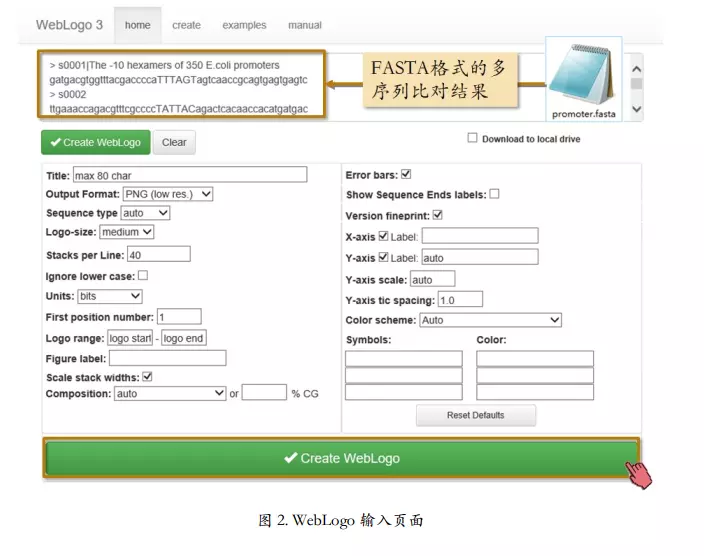
WebLogo 是一款在线创建序列标识图的软件(http://weblogo.threeplusone.com/)。
软件界面
image.png
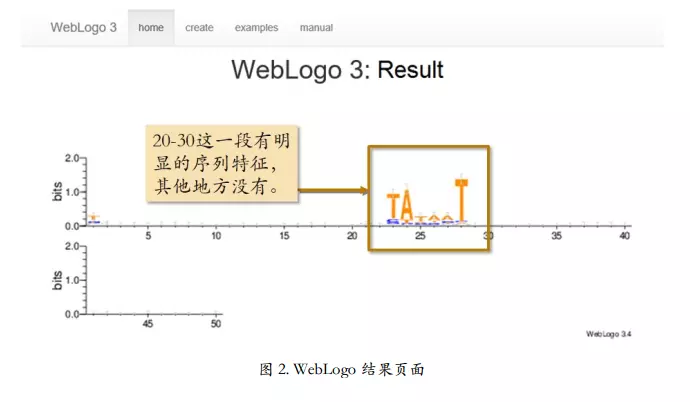
结果显示
image.png
寻找保守区域:序列基序 MEME
MEME 是 The MEME Suite 在线软件套装中的一员(http://meme-suite.org/)
image.png
进入软件
http://meme-suite.org/tools/meme
MEME HTML 结果页面
image.png
More 链接查看基序详情
image.png
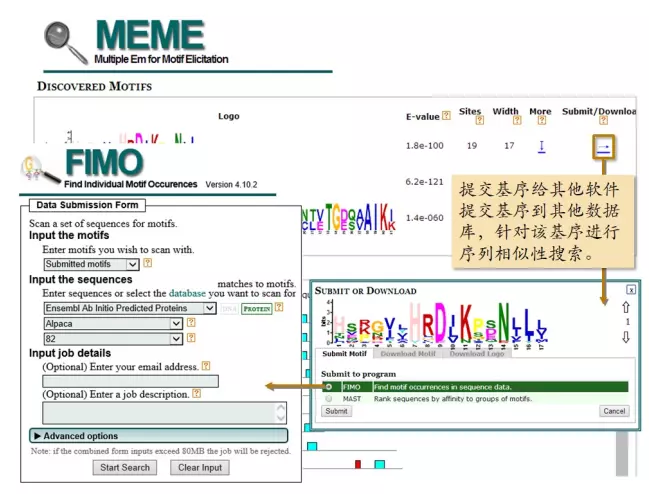
提交基序给 FIMO 进行数据库相似性搜索
![]()
image.png
image.png
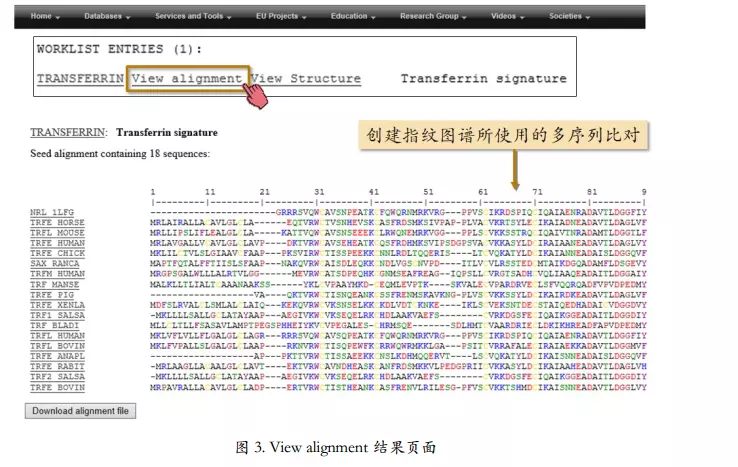
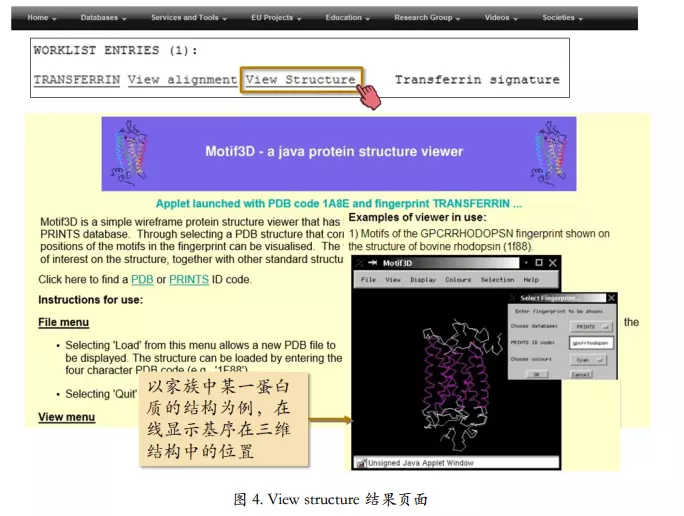
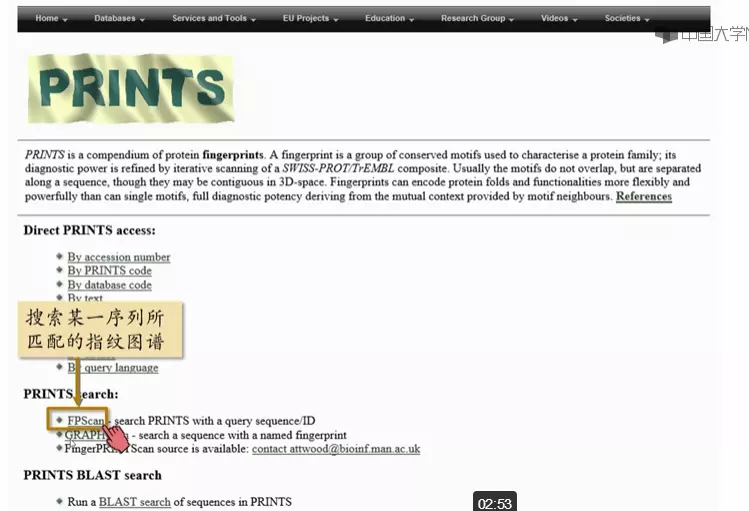
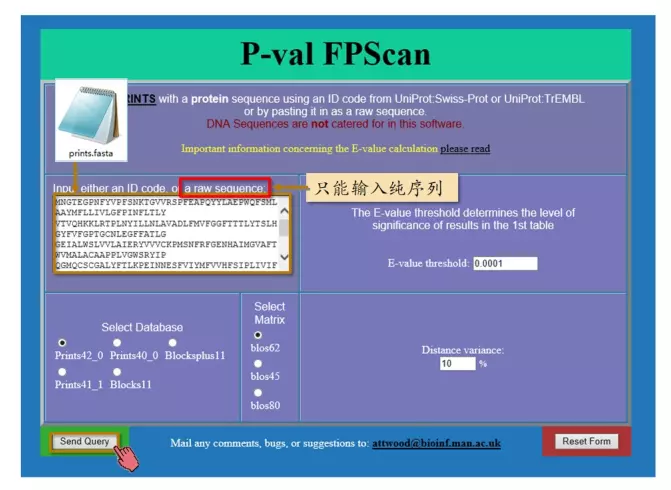
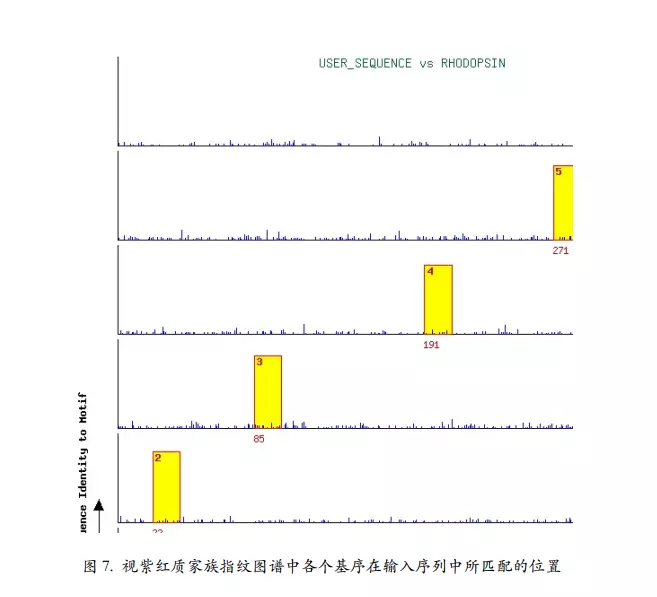
寻找保守区域:PRINTS 指纹图谱数据库
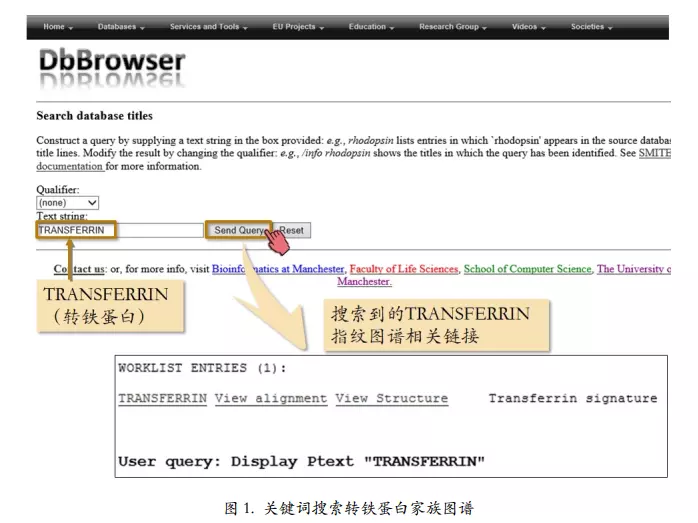
PRINTS 蛋白质序列指纹图谱数据库(http://www.bioinf.manchester.ac.uk/dbbrowser/PRINTS/)
image.png
关键词搜索转铁蛋白家族图谱
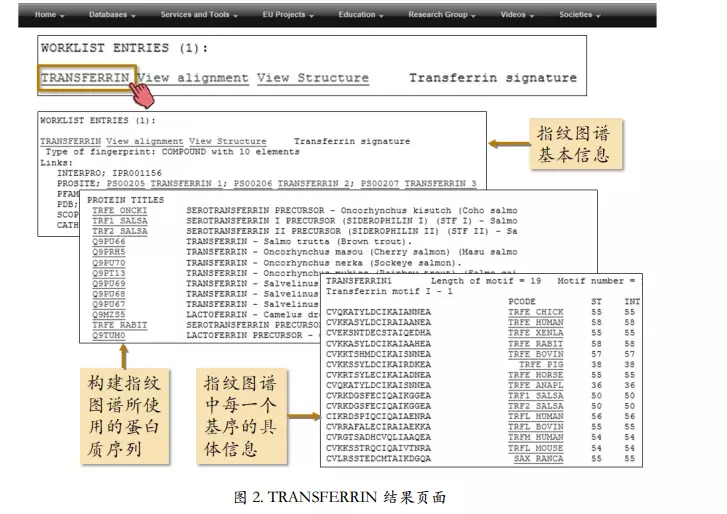
image.png
image.png
image.png
image.png
image.png
image.png
image.png
image.png
9人点赞
【生信课程】
leadingsci
拥有4钻 (约0.88元)