《利用Python进行数据分析·第2版》 附录A NumPy高级应用
第1章 准备工作
第2章 Python语法基础,IPython和Jupyter
第3章 Python的数据结构、函数和文件
第4章 NumPy基础:数组和矢量计算
第5章 pandas入门
第6章 数据加载、存储与文件格式
第7章 数据清洗和准备
第8章 数据规整:聚合、合并和重塑
第9章 绘图和可视化
第10章 数据聚合与分组运算
第11章 时间序列
第12章 pandas高级应用
第13章 Python建模库介绍
第14章 数据分析案例
附录A NumPy高级应用
附录B 更多关于IPython的内容(完)
在这篇附录中,我会深入NumPy库的数组计算。这会包括ndarray更内部的细节,和更高级的数组操作和算法。
本章包括了一些杂乱的章节,不需要仔细研究。
A.1 ndarray对象的内部机理
NumPy的ndarray提供了一种将同质数据块(可以是连续或跨越)解释为多维数组对象的方式。正如你之前所看到的那样,数据类型(dtype)决定了数据的解释方式,比如浮点数、整数、布尔值等。
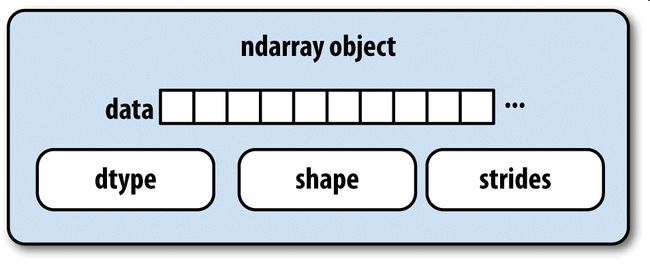
ndarray如此强大的部分原因是所有数组对象都是数据块的一个跨度视图(strided view)。你可能想知道数组视图arr[::2,::-1]不复制任何数据的原因是什么。简单地说,ndarray不只是一块内存和一个dtype,它还有跨度信息,这使得数组能以各种步幅(step size)在内存中移动。更准确地讲,ndarray内部由以下内容组成:
- 一个指向数据(内存或内存映射文件中的一块数据)的指针。
- 数据类型或dtype,描述在数组中的固定大小值的格子。
- 一个表示数组形状(shape)的元组。
- 一个跨度元组(stride),其中的整数指的是为了前进到当前维度下一个元素需要“跨过”的字节数。
图A-1简单地说明了ndarray的内部结构。
例如,一个10×5的数组,其形状为(10,5):
In [10]: np.ones((10, 5)).shape
Out[10]: (10, 5)
一个典型的(C顺序,稍后将详细讲解)3×4×5的float64(8个字节)数组,其跨度为(160,40,8) —— 知道跨度是非常有用的,通常,跨度在一个轴上越大,沿这个轴进行计算的开销就越大:
In [11]: np.ones((3, 4, 5), dtype=np.float64).strides
Out[11]: (160, 40, 8)
虽然NumPy用户很少会对数组的跨度信息感兴趣,但它们却是构建非复制式数组视图的重要因素。跨度甚至可以是负数,这样会使数组在内存中后向移动,比如在切片obj[::-1]或obj[:,::-1]中就是这样的。
NumPy数据类型体系
你可能偶尔需要检查数组中所包含的是否是整数、浮点数、字符串或Python对象。因为浮点数的种类很多(从float16到float128),判断dtype是否属于某个大类的工作非常繁琐。幸运的是,dtype都有一个超类(比如np.integer和np.floating),它们可以跟np.issubdtype函数结合使用:
In [12]: ints = np.ones(10, dtype=np.uint16)
In [13]: floats = np.ones(10, dtype=np.float32)
In [14]: np.issubdtype(ints.dtype, np.integer)
Out[14]: True
In [15]: np.issubdtype(floats.dtype, np.floating)
Out[15]: True
调用dtype的mro方法即可查看其所有的父类:
In [16]: np.float64.mro()
Out[16]:
[numpy.float64,
numpy.floating,
numpy.inexact,
numpy.number,
numpy.generic,
float,
object]
然后得到:
In [17]: np.issubdtype(ints.dtype, np.number)
Out[17]: True
大部分NumPy用户完全不需要了解这些知识,但是这些知识偶尔还是能派上用场的。图A-2说明了dtype体系以及父子类关系。
A.2 高级数组操作
除花式索引、切片、布尔条件取子集等操作之外,数组的操作方式还有很多。虽然pandas中的高级函数可以处理数据分析工作中的许多重型任务,但有时你还是需要编写一些在现有库中找不到的数据算法。
数组重塑
多数情况下,你可以无需复制任何数据,就将数组从一个形状转换为另一个形状。只需向数组的实例方法reshape传入一个表示新形状的元组即可实现该目的。例如,假设有一个一维数组,我们希望将其重新排列为一个矩阵(结果见图A-3):
In [18]: arr = np.arange(8)
In [19]: arr
Out[19]: array([0, 1, 2, 3, 4, 5, 6, 7])
In [20]: arr.reshape((4, 2))
Out[20]:
array([[0, 1],
[2, 3],
[4, 5],
[6, 7]])
多维数组也能被重塑:
In [21]: arr.reshape((4, 2)).reshape((2, 4))
Out[21]:
array([[0, 1, 2, 3],
[4, 5, 6, 7]])
作为参数的形状的其中一维可以是-1,它表示该维度的大小由数据本身推断而来:
In [22]: arr = np.arange(15)
In [23]: arr.reshape((5, -1))
Out[23]:
array([[ 0, 1, 2],
[ 3, 4, 5],
[ 6, 7, 8],
[ 9, 10, 11],
[12, 13, 14]])
与reshape将一维数组转换为多维数组的运算过程相反的运算通常称为扁平化(flattening)或散开(raveling):
In [27]: arr = np.arange(15).reshape((5, 3))
In [28]: arr
Out[28]:
array([[ 0, 1, 2],
[ 3, 4, 5],
[ 6, 7, 8],
[ 9, 10, 11],
[12, 13, 14]])
In [29]: arr.ravel()
Out[29]: array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14])
如果结果中的值与原始数组相同,ravel不会产生源数据的副本。flatten方法的行为类似于ravel,只不过它总是返回数据的副本:
In [30]: arr.flatten()
Out[30]: array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14])
数组可以被重塑或散开为别的顺序。这对NumPy新手来说是一个比较微妙的问题,所以在下一小节中我们将专门讲解这个问题。
C和Fortran顺序
NumPy允许你更为灵活地控制数据在内存中的布局。默认情况下,NumPy数组是按行优先顺序创建的。在空间方面,这就意味着,对于一个二维数组,每行中的数据项是被存放在相邻内存位置上的。另一种顺序是列优先顺序,它意味着每列中的数据项是被存放在相邻内存位置上的。
由于一些历史原因,行和列优先顺序又分别称为C和Fortran顺序。在FORTRAN 77中,矩阵全都是列优先的。
像reshape和reval这样的函数,都可以接受一个表示数组数据存放顺序的order参数。一般可以是'C'或'F'(还有'A'和'K'等不常用的选项,具体请参考NumPy的文档)。图A-3对此进行了说明:
In [31]: arr = np.arange(12).reshape((3, 4))
In [32]: arr
Out[32]:
array([[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[ 8, 9, 10, 11]])
In [33]: arr.ravel()
Out[33]: array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11])
In [34]: arr.ravel('F')
Out[34]: array([ 0, 4, 8, 1, 5, 9, 2, 6, 10, 3, 7, 11])
二维或更高维数组的重塑过程比较令人费解(见图A-3)。C和Fortran顺序的关键区别就是维度的行进顺序:
- C/行优先顺序:先经过更高的维度(例如,轴1会先于轴0被处理)。
- Fortran/列优先顺序:后经过更高的维度(例如,轴0会先于轴1被处理)。
数组的合并和拆分
numpy.concatenate可以按指定轴将一个由数组组成的序列(如元组、列表等)连接到一起:
In [35]: arr1 = np.array([[1, 2, 3], [4, 5, 6]])
In [36]: arr2 = np.array([[7, 8, 9], [10, 11, 12]])
In [37]: np.concatenate([arr1, arr2], axis=0)
Out[37]:
array([[ 1, 2, 3],
[ 4, 5, 6],
[ 7, 8, 9],
[10, 11, 12]])
In [38]: np.concatenate([arr1, arr2], axis=1)
Out[38]:
array([[ 1, 2, 3, 7, 8, 9],
[ 4, 5, 6, 10, 11, 12]])
对于常见的连接操作,NumPy提供了一些比较方便的方法(如vstack和hstack)。因此,上面的运算还可以表达为:
In [39]: np.vstack((arr1, arr2))
Out[39]:
array([[ 1, 2, 3],
[ 4, 5, 6],
[ 7, 8, 9],
[10, 11, 12]])
In [40]: np.hstack((arr1, arr2))
Out[40]:
array([[ 1, 2, 3, 7, 8, 9],
[ 4, 5, 6, 10, 11, 12]])
与此相反,split用于将一个数组沿指定轴拆分为多个数组:
In [41]: arr = np.random.randn(5, 2)
In [42]: arr
Out[42]:
array([[-0.2047, 0.4789],
[-0.5194, -0.5557],
[ 1.9658, 1.3934],
[ 0.0929, 0.2817],
[ 0.769 , 1.2464]])
In [43]: first, second, third = np.split(arr, [1, 3])
In [44]: first
Out[44]: array([[-0.2047, 0.4789]])
In [45]: second
Out[45]:
array([[-0.5194, -0.5557],
[ 1.9658, 1.3934]])
In [46]: third
Out[46]:
array([[ 0.0929, 0.2817],
[ 0.769 , 1.2464]])
传入到np.split的值[1,3]指示在哪个索引处分割数组。
表A-1中列出了所有关于数组连接和拆分的函数,其中有些是专门为了方便常见的连接运算而提供的。
堆叠辅助类:r_和c_
NumPy命名空间中有两个特殊的对象——r_和c_,它们可以使数组的堆叠操作更为简洁:
In [47]: arr = np.arange(6)
In [48]: arr1 = arr.reshape((3, 2))
In [49]: arr2 = np.random.randn(3, 2)
In [50]: np.r_[arr1, arr2]
Out[50]:
array([[ 0. , 1. ],
[ 2. , 3. ],
[ 4. , 5. ],
[ 1.0072, -1.2962],
[ 0.275 , 0.2289],
[ 1.3529, 0.8864]])
In [51]: np.c_[np.r_[arr1, arr2], arr]
Out[51]:
array([[ 0. , 1. , 0. ],
[ 2. , 3. , 1. ],
[ 4. , 5. , 2. ],
[ 1.0072, -1.2962, 3. ],
[ 0.275 , 0.2289, 4. ],
[ 1.3529, 0.8864, 5. ]])
它还可以将切片转换成数组:
In [52]: np.c_[1:6, -10:-5]
Out[52]:
array([[ 1, -10],
[ 2, -9],
[ 3, -8],
[ 4, -7],
[ 5, -6]])
r_和c_的具体功能请参考其文档。
元素的重复操作:tile和repeat
对数组进行重复以产生更大数组的工具主要是repeat和tile这两个函数。repeat会将数组中的各个元素重复一定次数,从而产生一个更大的数组:
In [53]: arr = np.arange(3)
In [54]: arr
Out[54]: array([0, 1, 2])
In [55]: arr.repeat(3)
Out[55]: array([0, 0, 0, 1, 1, 1, 2, 2, 2])
笔记:跟其他流行的数组编程语言(如MATLAB)不同,NumPy中很少需要对数组进行重复(replicate)。这主要是因为广播(broadcasting,我们将在下一节中讲解该技术)能更好地满足该需求。
默认情况下,如果传入的是一个整数,则各元素就都会重复那么多次。如果传入的是一组整数,则各元素就可以重复不同的次数:
In [56]: arr.repeat([2, 3, 4])
Out[56]: array([0, 0, 1, 1, 1, 2, 2, 2, 2])
对于多维数组,还可以让它们的元素沿指定轴重复:
In [57]: arr = np.random.randn(2, 2)
In [58]: arr
Out[58]:
array([[-2.0016, -0.3718],
[ 1.669 , -0.4386]])
In [59]: arr.repeat(2, axis=0)
Out[59]:
array([[-2.0016, -0.3718],
[-2.0016, -0.3718],
[ 1.669 , -0.4386],
[ 1.669 , -0.4386]])
注意,如果没有设置轴向,则数组会被扁平化,这可能不会是你想要的结果。同样,在对多维进行重复时,也可以传入一组整数,这样就会使各切片重复不同的次数:
In [60]: arr.repeat([2, 3], axis=0)
Out[60]:
array([[-2.0016, -0.3718],
[-2.0016, -0.3718],
[ 1.669 , -0.4386],
[ 1.669 , -0.4386],
[ 1.669 , -0.4386]])
In [61]: arr.repeat([2, 3], axis=1)
Out[61]:
array([[-2.0016, -2.0016, -0.3718, -0.3718, -0.3718],
[ 1.669 , 1.669 , -0.4386, -0.4386, -0.4386]])
tile的功能是沿指定轴向堆叠数组的副本。你可以形象地将其想象成“铺瓷砖”:
In [62]: arr
Out[62]:
array([[-2.0016, -0.3718],
[ 1.669 , -0.4386]])
In [63]: np.tile(arr, 2)
Out[63]:
array([[-2.0016, -0.3718, -2.0016, -0.3718],
[ 1.669 , -0.4386, 1.669 , -0.4386]])
第二个参数是瓷砖的数量。对于标量,瓷砖是水平铺设的,而不是垂直铺设。它可以是一个表示“铺设”布局的元组:
In [64]: arr
Out[64]:
array([[-2.0016, -0.3718],
[ 1.669 , -0.4386]])
In [65]: np.tile(arr, (2, 1))
Out[65]:
array([[-2.0016, -0.3718],
[ 1.669 , -0.4386],
[-2.0016, -0.3718],
[ 1.669 , -0.4386]])
In [66]: np.tile(arr, (3, 2))
Out[66]:
array([[-2.0016, -0.3718, -2.0016, -0.3718],
[ 1.669 , -0.4386, 1.669 , -0.4386],
[-2.0016, -0.3718, -2.0016, -0.3718],
[ 1.669 , -0.4386, 1.669 , -0.4386],
[-2.0016, -0.3718, -2.0016, -0.3718],
[ 1.669 , -0.4386, 1.669 , -0.4386]])
花式索引的等价函数:take和put
在第4章中我们讲过,获取和设置数组子集的一个办法是通过整数数组使用花式索引:
In [67]: arr = np.arange(10) * 100
In [68]: inds = [7, 1, 2, 6]
In [69]: arr[inds]
Out[69]: array([700, 100, 200, 600])
ndarray还有其它方法用于获取单个轴向上的选区:
In [70]: arr.take(inds)
Out[70]: array([700, 100, 200, 600])
In [71]: arr.put(inds, 42)
In [72]: arr
Out[72]: array([ 0, 42, 42, 300, 400, 500, 42, 42,800, 900])
In [73]: arr.put(inds, [40, 41, 42, 43])
In [74]: arr
Out[74]: array([ 0, 41, 42, 300, 400, 500, 43, 40, 800, 900])
要在其它轴上使用take,只需传入axis关键字即可:
In [75]: inds = [2, 0, 2, 1]
In [76]: arr = np.random.randn(2, 4)
In [77]: arr
Out[77]:
array([[-0.5397, 0.477 , 3.2489, -1.0212],
[-0.5771, 0.1241, 0.3026, 0.5238]])
In [78]: arr.take(inds, axis=1)
Out[78]:
array([[ 3.2489, -0.5397, 3.2489, 0.477 ],
[ 0.3026, -0.5771, 0.3026, 0.1241]])
put不接受axis参数,它只会在数组的扁平化版本(一维,C顺序)上进行索引。因此,在需要用其他轴向的索引设置元素时,最好还是使用花式索引。
A.3 广播
广播(broadcasting)指的是不同形状的数组之间的算术运算的执行方式。它是一种非常强大的功能,但也容易令人误解,即使是经验丰富的老手也是如此。将标量值跟数组合并时就会发生最简单的广播:
In [79]: arr = np.arange(5)
In [80]: arr
Out[80]: array([0, 1, 2, 3, 4])
In [81]: arr * 4
Out[81]: array([ 0, 4, 8, 12, 16])
这里我们说:在这个乘法运算中,标量值4被广播到了其他所有的元素上。
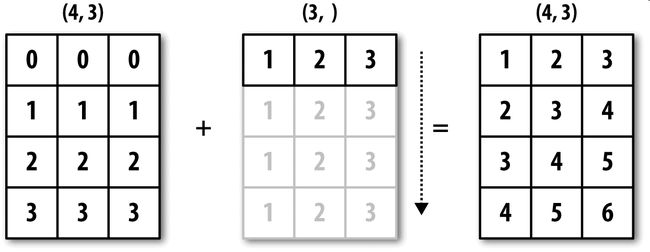
看一个例子,我们可以通过减去列平均值的方式对数组的每一列进行距平化处理。这个问题解决起来非常简单:
In [82]: arr = np.random.randn(4, 3)
In [83]: arr.mean(0)
Out[83]: array([-0.3928, -0.3824, -0.8768])
In [84]: demeaned = arr - arr.mean(0)
In [85]: demeaned
Out[85]:
array([[ 0.3937, 1.7263, 0.1633],
[-0.4384, -1.9878, -0.9839],
[-0.468 , 0.9426, -0.3891],
[ 0.5126, -0.6811, 1.2097]])
In [86]: demeaned.mean(0)
Out[86]: array([-0., 0., -0.])
图A-4形象地展示了该过程。用广播的方式对行进行距平化处理会稍微麻烦一些。幸运的是,只要遵循一定的规则,低维度的值是可以被广播到数组的任意维度的(比如对二维数组各列减去行平均值)。
于是就得到了:
虽然我是一名经验丰富的NumPy老手,但经常还是得停下来画张图并想想广播的原则。再来看一下最后那个例子,假设你希望对各行减去那个平均值。由于arr.mean(0)的长度为3,所以它可以在0轴向上进行广播:因为arr的后缘维度是3,所以它们是兼容的。根据该原则,要在1轴向上做减法(即各行减去行平均值),较小的那个数组的形状必须是(4,1):
In [87]: arr
Out[87]:
array([[ 0.0009, 1.3438, -0.7135],
[-0.8312, -2.3702, -1.8608],
[-0.8608, 0.5601, -1.2659],
[ 0.1198, -1.0635, 0.3329]])
In [88]: row_means = arr.mean(1)
In [89]: row_means.shape
Out[89]: (4,)
In [90]: row_means.reshape((4, 1))
Out[90]:
array([[ 0.2104],
[-1.6874],
[-0.5222],
[-0.2036]])
In [91]: demeaned = arr - row_means.reshape((4, 1))
In [92]: demeaned.mean(1)
Out[92]: array([ 0., -0., 0., 0.])
图A-5说明了该运算的过程。
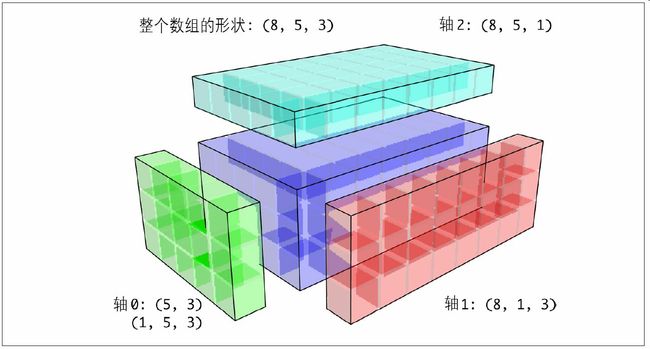
图A-6展示了另外一种情况,这次是在一个三维数组上沿0轴向加上一个二维数组。
沿其它轴向广播
高维度数组的广播似乎更难以理解,而实际上它也是遵循广播原则的。如果不然,你就会得到下面这样一个错误:
In [93]: arr - arr.mean(1)
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
in ()
----> 1 arr - arr.mean(1)
ValueError: operands could not be broadcast together with shapes (4,3) (4,)
人们经常需要通过算术运算过程将较低维度的数组在除0轴以外的其他轴向上广播。根据广播的原则,较小数组的“广播维”必须为1。在上面那个行距平化的例子中,这就意味着要将行平均值的形状变成(4,1)而不是(4,):
In [94]: arr - arr.mean(1).reshape((4, 1))
Out[94]:
array([[-0.2095, 1.1334, -0.9239],
[ 0.8562, -0.6828, -0.1734],
[-0.3386, 1.0823, -0.7438],
[ 0.3234, -0.8599, 0.5365]])
对于三维的情况,在三维中的任何一维上广播其实也就是将数据重塑为兼容的形状而已。图A-7说明了要在三维数组各维度上广播的形状需求。
于是就有了一个非常普遍的问题(尤其是在通用算法中),即专门为了广播而添加一个长度为1的新轴。虽然reshape是一个办法,但插入轴需要构造一个表示新形状的元组。这是一个很郁闷的过程。因此,NumPy数组提供了一种通过索引机制插入轴的特殊语法。下面这段代码通过特殊的np.newaxis属性以及“全”切片来插入新轴:
In [95]: arr = np.zeros((4, 4))
In [96]: arr_3d = arr[:, np.newaxis, :]
In [97]: arr_3d.shape
Out[97]: (4, 1, 4)
In [98]: arr_1d = np.random.normal(size=3)
In [99]: arr_1d[:, np.newaxis]
Out[99]:
array([[-2.3594],
[-0.1995],
[-1.542 ]])
In [100]: arr_1d[np.newaxis, :]
Out[100]: array([[-2.3594, -0.1995, -1.542 ]])
因此,如果我们有一个三维数组,并希望对轴2进行距平化,那么只需要编写下面这样的代码就可以了:
In [101]: arr = np.random.randn(3, 4, 5)
In [102]: depth_means = arr.mean(2)
In [103]: depth_means
Out[103]:
array([[-0.4735, 0.3971, -0.0228, 0.2001],
[-0.3521, -0.281 , -0.071 , -0.1586],
[ 0.6245, 0.6047, 0.4396, -0.2846]])
In [104]: depth_means.shape
Out[104]: (3, 4)
In [105]: demeaned = arr - depth_means[:, :, np.newaxis]
In [106]: demeaned.mean(2)
Out[106]:
array([[ 0., 0., -0., -0.],
[ 0., 0., -0., 0.],
[ 0., 0., -0., -0.]])
有些读者可能会想,在对指定轴进行距平化时,有没有一种既通用又不牺牲性能的方法呢?实际上是有的,但需要一些索引方面的技巧:
def demean_axis(arr, axis=0):
means = arr.mean(axis)
# This generalizes things like [:, :, np.newaxis] to N dimensions
indexer = [slice(None)] * arr.ndim
indexer[axis] = np.newaxis
return arr - means[indexer]
通过广播设置数组的值
算术运算所遵循的广播原则同样也适用于通过索引机制设置数组值的操作。对于最简单的情况,我们可以这样做:
In [107]: arr = np.zeros((4, 3))
In [108]: arr[:] = 5
In [109]: arr
Out[109]:
array([[ 5., 5., 5.],
[ 5., 5., 5.],
[ 5., 5., 5.],
[ 5., 5., 5.]])
但是,假设我们想要用一个一维数组来设置目标数组的各列,只要保证形状兼容就可以了:
In [110]: col = np.array([1.28, -0.42, 0.44, 1.6])
In [111]: arr[:] = col[:, np.newaxis]
In [112]: arr
Out[112]:
array([[ 1.28, 1.28, 1.28],
[-0.42, -0.42, -0.42],
[ 0.44, 0.44, 0.44],
[ 1.6 , 1.6 , 1.6 ]])
In [113]: arr[:2] = [[-1.37], [0.509]]
In [114]: arr
Out[114]:
array([[-1.37 , -1.37 , -1.37 ],
[ 0.509, 0.509, 0.509],
[ 0.44 , 0.44 , 0.44 ],
[ 1.6 , 1.6 , 1.6 ]])
A.4 ufunc高级应用
虽然许多NumPy用户只会用到通用函数所提供的快速的元素级运算,但通用函数实际上还有一些高级用法能使我们丢开循环而编写出更为简洁的代码。
ufunc实例方法
NumPy的各个二元ufunc都有一些用于执行特定矢量化运算的特殊方法。表A-2汇总了这些方法,下面我将通过几个具体的例子对它们进行说明。
reduce接受一个数组参数,并通过一系列的二元运算对其值进行聚合(可指明轴向)。例如,我们可以用np.add.reduce对数组中各个元素进行求和:
In [115]: arr = np.arange(10)
In [116]: np.add.reduce(arr)
Out[116]: 45
In [117]: arr.sum()
Out[117]: 45
起始值取决于ufunc(对于add的情况,就是0)。如果设置了轴号,约简运算就会沿该轴向执行。这就使你能用一种比较简洁的方式得到某些问题的答案。在下面这个例子中,我们用np.logical_and检查数组各行中的值是否是有序的:
In [118]: np.random.seed(12346) # for reproducibility
In [119]: arr = np.random.randn(5, 5)
In [120]: arr[::2].sort(1) # sort a few rows
In [121]: arr[:, :-1] < arr[:, 1:]
Out[121]:
array([[ True, True, True, True],
[False, True, False, False],
[ True, True, True, True],
[ True, False, True, True],
[ True, True, True, True]], dtype=bool)
In [122]: np.logical_and.reduce(arr[:, :-1] < arr[:, 1:], axis=1)
Out[122]: array([ True, False, True, False, True], dtype=bool)
注意,logical_and.reduce跟all方法是等价的。
ccumulate跟reduce的关系就像cumsum跟sum的关系那样。它产生一个跟原数组大小相同的中间“累计”值数组:
In [123]: arr = np.arange(15).reshape((3, 5))
In [124]: np.add.accumulate(arr, axis=1)
Out[124]:
array([[ 0, 1, 3, 6, 10],
[ 5, 11, 18, 26, 35],
[10, 21, 33, 46, 60]])
outer用于计算两个数组的叉积:
In [125]: arr = np.arange(3).repeat([1, 2, 2])
In [126]: arr
Out[126]: array([0, 1, 1, 2, 2])
In [127]: np.multiply.outer(arr, np.arange(5))
Out[127]:
array([[0, 0, 0, 0, 0],
[0, 1, 2, 3, 4],
[0, 1, 2, 3, 4],
[0, 2, 4, 6, 8],
[0, 2, 4, 6, 8]])
outer输出结果的维度是两个输入数据的维度之和:
In [128]: x, y = np.random.randn(3, 4), np.random.randn(5)
In [129]: result = np.subtract.outer(x, y)
In [130]: result.shape
Out[130]: (3, 4, 5)
最后一个方法reduceat用于计算“局部约简”,其实就是一个对数据各切片进行聚合的groupby运算。它接受一组用于指示如何对值进行拆分和聚合的“面元边界”:
In [131]: arr = np.arange(10)
In [132]: np.add.reduceat(arr, [0, 5, 8])
Out[132]: array([10, 18, 17])
最终结果是在arr[0:5]、arr[5:8]以及arr[8:]上执行的约简。跟其他方法一样,这里也可以传入一个axis参数:
In [133]: arr = np.multiply.outer(np.arange(4), np.arange(5))
In [134]: arr
Out[134]:
array([[ 0, 0, 0, 0, 0],
[ 0, 1, 2, 3, 4],
[ 0, 2, 4, 6, 8],
[ 0, 3, 6, 9, 12]])
In [135]: np.add.reduceat(arr, [0, 2, 4], axis=1)
Out[135]:
array([[ 0, 0, 0],
[ 1, 5, 4],
[ 2, 10, 8],
[ 3, 15, 12]])
表A-2总结了部分的ufunc方法。
编写新的ufunc
有多种方法可以让你编写自己的NumPy ufuncs。最常见的是使用NumPy C API,但它超越了本书的范围。在本节,我们讲纯粹的Python ufunc。
numpy.frompyfunc接受一个Python函数以及两个分别表示输入输出参数数量的参数。例如,下面是一个能够实现元素级加法的简单函数:
In [136]: def add_elements(x, y):
.....: return x + y
In [137]: add_them = np.frompyfunc(add_elements, 2, 1)
In [138]: add_them(np.arange(8), np.arange(8))
Out[138]: array([0, 2, 4, 6, 8, 10, 12, 14], dtype=object)
用frompyfunc创建的函数总是返回Python对象数组,这一点很不方便。幸运的是,还有另一个办法,即numpy.vectorize。虽然没有frompyfunc那么强大,但可以让你指定输出类型:
In [139]: add_them = np.vectorize(add_elements, otypes=[np.float64])
In [140]: add_them(np.arange(8), np.arange(8))
Out[140]: array([ 0., 2., 4., 6., 8., 10., 12., 14.])
虽然这两个函数提供了一种创建ufunc型函数的手段,但它们非常慢,因为它们在计算每个元素时都要执行一次Python函数调用,这就会比NumPy自带的基于C的ufunc慢很多:
In [141]: arr = np.random.randn(10000)
In [142]: %timeit add_them(arr, arr)
4.12 ms +- 182 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [143]: %timeit np.add(arr, arr)
6.89 us +- 504 ns per loop (mean +- std. dev. of 7 runs, 100000 loops each)
本章的后面,我会介绍使用Numba(http://numba.pydata.org/),创建快速Python ufuncs。
A.5 结构化和记录式数组
你可能已经注意到了,到目前为止我们所讨论的ndarray都是一种同质数据容器,也就是说,在它所表示的内存块中,各元素占用的字节数相同(具体根据dtype而定)。从表面上看,它似乎不能用于表示异质或表格型的数据。结构化数组是一种特殊的ndarray,其中的各个元素可以被看做C语言中的结构体(struct,这就是“结构化”的由来)或SQL表中带有多个命名字段的行:
In [144]: dtype = [('x', np.float64), ('y', np.int32)]
In [145]: sarr = np.array([(1.5, 6), (np.pi, -2)], dtype=dtype)
In [146]: sarr
Out[146]:
array([( 1.5 , 6), ( 3.1416, -2)],
dtype=[('x', '定义结构化dtype(请参考NumPy的在线文档)的方式有很多。最典型的办法是元组列表,各元组的格式为(field_name,field_data_type)。这样,数组的元素就成了元组式的对象,该对象中各个元素可以像字典那样进行访问:
In [147]: sarr[0]
Out[147]: ( 1.5, 6)
In [148]: sarr[0]['y']
Out[148]: 6
字段名保存在dtype.names属性中。在访问结构化数组的某个字段时,返回的是该数据的视图,所以不会发生数据复制:
In [149]: sarr['x']
Out[149]: array([ 1.5 , 3.1416])
嵌套dtype和多维字段
在定义结构化dtype时,你可以再设置一个形状(可以是一个整数,也可以是一个元组):
In [150]: dtype = [('x', np.int64, 3), ('y', np.int32)]
In [151]: arr = np.zeros(4, dtype=dtype)
In [152]: arr
Out[152]:
array([([0, 0, 0], 0), ([0, 0, 0], 0), ([0, 0, 0], 0), ([0, 0, 0], 0)],
dtype=[('x', '在这种情况下,各个记录的x字段所表示的是一个长度为3的数组:
In [153]: arr[0]['x']
Out[153]: array([0, 0, 0])
这样,访问arr['x']即可得到一个二维数组,而不是前面那个例子中的一维数组:
In [154]: arr['x']
Out[154]:
array([[0, 0, 0],
[0, 0, 0],
[0, 0, 0],
[0, 0, 0]])
这就使你能用单个数组的内存块存放复杂的嵌套结构。你还可以嵌套dtype,作出更复杂的结构。下面是一个简单的例子:
In [155]: dtype = [('x', [('a', 'f8'), ('b', 'f4')]), ('y', np.int32)]
In [156]: data = np.array([((1, 2), 5), ((3, 4), 6)], dtype=dtype)
In [157]: data['x']
Out[157]:
array([( 1., 2.), ( 3., 4.)],
dtype=[('a', 'pandas的DataFrame并不直接支持该功能,但它的分层索引机制跟这个差不多。
为什么要用结构化数组
跟pandas的DataFrame相比,NumPy的结构化数组是一种相对较低级的工具。它可以将单个内存块解释为带有任意复杂嵌套列的表格型结构。由于数组中的每个元素在内存中都被表示为固定的字节数,所以结构化数组能够提供非常快速高效的磁盘数据读写(包括内存映像)、网络传输等功能。
结构化数组的另一个常见用法是,将数据文件写成定长记录字节流,这是C和C++代码中常见的数据序列化手段(业界许多历史系统中都能找得到)。只要知道文件的格式(记录的大小、元素的顺序、字节数以及数据类型等),就可以用np.fromfile将数据读入内存。这种用法超出了本书的范围,知道这点就可以了。
A.6 更多有关排序的话题
跟Python内置的列表一样,ndarray的sort实例方法也是就地排序。也就是说,数组内容的重新排列是不会产生新数组的:
In [160]: arr = np.random.randn(6)
In [161]: arr.sort()
In [162]: arr
Out[162]: array([-1.082 , 0.3759, 0.8014, 1.1397, 1.2888, 1.8413])
在对数组进行就地排序时要注意一点,如果目标数组只是一个视图,则原始数组将会被修改:
In [163]: arr = np.random.randn(3, 5)
In [164]: arr
Out[164]:
array([[-0.3318, -1.4711, 0.8705, -0.0847, -1.1329],
[-1.0111, -0.3436, 2.1714, 0.1234, -0.0189],
[ 0.1773, 0.7424, 0.8548, 1.038 , -0.329 ]])
In [165]: arr[:, 0].sort() # Sort first column values in-place
In [166]: arr
Out[166]:
array([[-1.0111, -1.4711, 0.8705, -0.0847, -1.1329],
[-0.3318, -0.3436, 2.1714, 0.1234, -0.0189],
[ 0.1773, 0.7424, 0.8548, 1.038 , -0.329 ]])
相反,numpy.sort会为原数组创建一个已排序副本。另外,它所接受的参数(比如kind)跟ndarray.sort一样:
In [167]: arr = np.random.randn(5)
In [168]: arr
Out[168]: array([-1.1181, -0.2415, -2.0051, 0.7379, -1.0614])
In [169]: np.sort(arr)
Out[169]: array([-2.0051, -1.1181, -1.0614, -0.2415, 0.7379])
In [170]: arr
Out[170]: array([-1.1181, -0.2415, -2.0051, 0.7379, -1.0614])
这两个排序方法都可以接受一个axis参数,以便沿指定轴向对各块数据进行单独排序:
In [171]: arr = np.random.randn(3, 5)
In [172]: arr
Out[172]:
array([[ 0.5955, -0.2682, 1.3389, -0.1872, 0.9111],
[-0.3215, 1.0054, -0.5168, 1.1925, -0.1989],
[ 0.3969, -1.7638, 0.6071, -0.2222, -0.2171]])
In [173]: arr.sort(axis=1)
In [174]: arr
Out[174]:
array([[-0.2682, -0.1872, 0.5955, 0.9111, 1.3389],
[-0.5168, -0.3215, -0.1989, 1.0054, 1.1925],
[-1.7638, -0.2222, -0.2171, 0.3969, 0.6071]])
你可能注意到了,这两个排序方法都不可以被设置为降序。其实这也无所谓,因为数组切片会产生视图(也就是说,不会产生副本,也不需要任何其他的计算工作)。许多Python用户都很熟悉一个有关列表的小技巧:values[::-1]可以返回一个反序的列表。对ndarray也是如此:
In [175]: arr[:, ::-1]
Out[175]:
array([[ 1.3389, 0.9111, 0.5955, -0.1872, -0.2682],
[ 1.1925, 1.0054, -0.1989, -0.3215, -0.5168],
[ 0.6071, 0.3969, -0.2171, -0.2222, -1.7638]])
间接排序:argsort和lexsort
在数据分析工作中,常常需要根据一个或多个键对数据集进行排序。例如,一个有关学生信息的数据表可能需要以姓和名进行排序(先姓后名)。这就是间接排序的一个例子,如果你阅读过有关pandas的章节,那就已经见过不少高级例子了。给定一个或多个键,你就可以得到一个由整数组成的索引数组(我亲切地称之为索引器),其中的索引值说明了数据在新顺序下的位置。argsort和numpy.lexsort就是实现该功能的两个主要方法。下面是一个简单的例子:
In [176]: values = np.array([5, 0, 1, 3, 2])
In [177]: indexer = values.argsort()
In [178]: indexer
Out[178]: array([1, 2, 4, 3, 0])
In [179]: values[indexer]
Out[179]: array([0, 1, 2, 3, 5])
一个更复杂的例子,下面这段代码根据数组的第一行对其进行排序:
In [180]: arr = np.random.randn(3, 5)
In [181]: arr[0] = values
In [182]: arr
Out[182]:
array([[ 5. , 0. , 1. , 3. , 2. ],
[-0.3636, -0.1378, 2.1777, -0.4728, 0.8356],
[-0.2089, 0.2316, 0.728 , -1.3918, 1.9956]])
In [183]: arr[:, arr[0].argsort()]
Out[183]:
array([[ 0. , 1. , 2. , 3. , 5. ],
[-0.1378, 2.1777, 0.8356, -0.4728, -0.3636],
[ 0.2316, 0.728 , 1.9956, -1.3918, -0.2089]])
lexsort跟argsort差不多,只不过它可以一次性对多个键数组执行间接排序(字典序)。假设我们想对一些以姓和名标识的数据进行排序:
In [184]: first_name = np.array(['Bob', 'Jane', 'Steve', 'Bill', 'Barbara'])
In [185]: last_name = np.array(['Jones', 'Arnold', 'Arnold', 'Jones', 'Walters'])
In [186]: sorter = np.lexsort((first_name, last_name))
In [187]: sorter
Out[187]: array([1, 2, 3, 0, 4])
In [188]: zip(last_name[sorter], first_name[sorter])
Out[188]:
刚开始使用lexsort的时候可能会比较容易头晕,这是因为键的应用顺序是从最后一个传入的算起的。不难看出,last_name是先于first_name被应用的。
笔记:Series和DataFrame的sort_index以及Series的order方法就是通过这些函数的变体(它们还必须考虑缺失值)实现的。
其他排序算法
稳定的(stable)排序算法会保持等价元素的相对位置。对于相对位置具有实际意义的那些间接排序而言,这一点非常重要:
In [189]: values = np.array(['2:first', '2:second', '1:first', '1:second',
.....: '1:third'])
In [190]: key = np.array([2, 2, 1, 1, 1])
In [191]: indexer = key.argsort(kind='mergesort')
In [192]: indexer
Out[192]: array([2, 3, 4, 0, 1])
In [193]: values.take(indexer)
Out[193]:
array(['1:first', '1:second', '1:third', '2:first', '2:second'],
dtype='mergesort(合并排序)是唯一的稳定排序,它保证有O(n log n)的性能(空间复杂度),但是其平均性能比默认的quicksort(快速排序)要差。表A-3列出了可用的排序算法及其相关的性能指标。大部分用户完全不需要知道这些东西,但了解一下总是好的。
部分排序数组
排序的目的之一可能是确定数组中最大或最小的元素。NumPy有两个优化方法,numpy.partition和np.argpartition,可以在第k个最小元素划分的数组:
In [194]: np.random.seed(12345)
In [195]: arr = np.random.randn(20)
In [196]: arr
Out[196]:
array([-0.2047, 0.4789, -0.5194, -0.5557, 1.9658, 1.3934, 0.0929,
0.2817, 0.769 , 1.2464, 1.0072, -1.2962, 0.275 , 0.2289,
1.3529, 0.8864, -2.0016, -0.3718, 1.669 , -0.4386])
In [197]: np.partition(arr, 3)
Out[197]:
array([-2.0016, -1.2962, -0.5557, -0.5194, -0.3718, -0.4386, -0.2047,
0.2817, 0.769 , 0.4789, 1.0072, 0.0929, 0.275 , 0.2289,
1.3529, 0.8864, 1.3934, 1.9658, 1.669 , 1.2464])
当你调用partition(arr, 3),结果中的头三个元素是最小的三个,没有特定的顺序。numpy.argpartition与numpy.argsort相似,会返回索引,重排数据为等价的顺序:
In [198]: indices = np.argpartition(arr, 3)
In [199]: indices
Out[199]:
array([16, 11, 3, 2, 17, 19, 0, 7, 8, 1, 10, 6, 12, 13, 14, 15, 5,
4, 18, 9])
In [200]: arr.take(indices)
Out[200]:
array([-2.0016, -1.2962, -0.5557, -0.5194, -0.3718, -0.4386, -0.2047,
0.2817, 0.769 , 0.4789, 1.0072, 0.0929, 0.275 , 0.2289,
1.3529, 0.8864, 1.3934, 1.9658, 1.669 , 1.2464])
numpy.searchsorted:在有序数组中查找元素
searchsorted是一个在有序数组上执行二分查找的数组方法,只要将值插入到它返回的那个位置就能维持数组的有序性:
In [201]: arr = np.array([0, 1, 7, 12, 15])
In [202]: arr.searchsorted(9)
Out[202]: 3
你可以传入一组值就能得到一组索引:
In [203]: arr.searchsorted([0, 8, 11, 16])
Out[203]: array([0, 3, 3, 5])
从上面的结果中可以看出,对于元素0,searchsorted会返回0。这是因为其默认行为是返回相等值组的左侧索引:
In [204]: arr = np.array([0, 0, 0, 1, 1, 1, 1])
In [205]: arr.searchsorted([0, 1])
Out[205]: array([0, 3])
In [206]: arr.searchsorted([0, 1], side='right')
Out[206]: array([3, 7])
再来看searchsorted的另一个用法,假设我们有一个数据数组(其中的值在0到10000之间),还有一个表示“面元边界”的数组,我们希望用它将数据数组拆分开:
In [207]: data = np.floor(np.random.uniform(0, 10000, size=50))
In [208]: bins = np.array([0, 100, 1000, 5000, 10000])
In [209]: data
Out[209]:
array([ 9940., 6768., 7908., 1709., 268., 8003., 9037., 246.,
4917., 5262., 5963., 519., 8950., 7282., 8183., 5002.,
8101., 959., 2189., 2587., 4681., 4593., 7095., 1780.,
5314., 1677., 7688., 9281., 6094., 1501., 4896., 3773.,
8486., 9110., 3838., 3154., 5683., 1878., 1258., 6875.,
7996., 5735., 9732., 6340., 8884., 4954., 3516., 7142.,
5039., 2256.])
然后,为了得到各数据点所属区间的编号(其中1表示面元[0,100)),我们可以直接使用searchsorted:
In [210]: labels = bins.searchsorted(data)
In [211]: labels
Out[211]:
array([4, 4, 4, 3, 2, 4, 4, 2, 3, 4, 4, 2, 4, 4, 4, 4, 4, 2, 3, 3, 3, 3, 4,
3, 4, 3, 4, 4, 4, 3, 3, 3, 4, 4, 3, 3, 4, 3, 3, 4, 4, 4, 4, 4, 4, 3,
3, 4, 4, 3])
通过pandas的groupby使用该结果即可非常轻松地对原数据集进行拆分:
In [212]: pd.Series(data).groupby(labels).mean()
Out[212]:
2 498.000000
3 3064.277778
4 7389.035714
dtype: float64
A.7 用Numba编写快速NumPy函数
Numba是一个开源项目,它可以利用CPUs、GPUs或其它硬件为类似NumPy的数据创建快速函数。它使用了LLVM项目(http://llvm.org/),将Python代码转换为机器代码。
为了介绍Numba,来考虑一个纯粹的Python函数,它使用for循环计算表达式(x - y).mean():
import numpy as np
def mean_distance(x, y):
nx = len(x)
result = 0.0
count = 0
for i in range(nx):
result += x[i] - y[i]
count += 1
return result / count
这个函数很慢:
In [209]: x = np.random.randn(10000000)
In [210]: y = np.random.randn(10000000)
In [211]: %timeit mean_distance(x, y)
1 loop, best of 3: 2 s per loop
In [212]: %timeit (x - y).mean()
100 loops, best of 3: 14.7 ms per loop
NumPy的版本要比它快过100倍。我们可以转换这个函数为编译的Numba函数,使用numba.jit函数:
In [213]: import numba as nb
In [214]: numba_mean_distance = nb.jit(mean_distance)
也可以写成装饰器:
@nb.jit
def mean_distance(x, y):
nx = len(x)
result = 0.0
count = 0
for i in range(nx):
result += x[i] - y[i]
count += 1
return result / count
它要比矢量化的NumPy快:
In [215]: %timeit numba_mean_distance(x, y)
100 loops, best of 3: 10.3 ms per loop
Numba不能编译Python代码,但它支持纯Python写的一个部分,可以编写数值算法。
Numba是一个深厚的库,支持多种硬件、编译模式和用户插件。它还可以编译NumPy Python API的一部分,而不用for循环。Numba也可以识别可以便以为机器编码的结构体,但是若调用CPython API,它就不知道如何编译。Numba的jit函数有一个选项,nopython=True,它限制了可以被转换为Python代码的代码,这些代码可以编译为LLVM,但没有任何Python C API调用。jit(nopython=True)有一个简短的别名numba.njit。
前面的例子,我们还可以这样写:
from numba import float64, njit
@njit(float64(float64[:], float64[:]))
def mean_distance(x, y):
return (x - y).mean()
我建议你学习Numba的线上文档(http://numba.pydata.org/)。下一节介绍一个创建自定义Numpy ufunc对象的例子。
用Numba创建自定义numpy.ufunc对象
numba.vectorize创建了一个编译的NumPy ufunc,它与内置的ufunc很像。考虑一个numpy.add的Python例子:
from numba import vectorize
@vectorize
def nb_add(x, y):
return x + y
现在有:
In [13]: x = np.arange(10)
In [14]: nb_add(x, x)
Out[14]: array([ 0., 2., 4., 6., 8., 10., 12., 14., 16., 18.])
In [15]: nb_add.accumulate(x, 0)
Out[15]: array([ 0., 1., 3., 6., 10., 15., 21., 28., 36., 45.])
A.8 高级数组输入输出
我在第4章中讲过,np.save和np.load可用于读写磁盘上以二进制格式存储的数组。其实还有一些工具可用于更为复杂的场景。尤其是内存映像(memory map),它使你能处理在内存中放不下的数据集。
内存映像文件
内存映像文件是一种将磁盘上的非常大的二进制数据文件当做内存中的数组进行处理的方式。NumPy实现了一个类似于ndarray的memmap对象,它允许将大文件分成小段进行读写,而不是一次性将整个数组读入内存。另外,memmap也拥有跟普通数组一样的方法,因此,基本上只要是能用于ndarray的算法就也能用于memmap。
要创建一个内存映像,可以使用函数np.memmap并传入一个文件路径、数据类型、形状以及文件模式:
In [214]: mmap = np.memmap('mymmap', dtype='float64', mode='w+',
.....: shape=(10000, 10000))
In [215]: mmap
Out[215]:
memmap([[ 0., 0., 0., ..., 0., 0., 0.],
[ 0., 0., 0., ..., 0., 0., 0.],
[ 0., 0., 0., ..., 0., 0., 0.],
...,
[ 0., 0., 0., ..., 0., 0., 0.],
[ 0., 0., 0., ..., 0., 0., 0.],
[ 0., 0., 0., ..., 0., 0., 0.]])
对memmap切片将会返回磁盘上的数据的视图:
In [216]: section = mmap[:5]
如果将数据赋值给这些视图:数据会先被缓存在内存中(就像是Python的文件对象),调用flush即可将其写入磁盘:
In [217]: section[:] = np.random.randn(5, 10000)
In [218]: mmap.flush()
In [219]: mmap
Out[219]:
memmap([[ 0.7584, -0.6605, 0.8626, ..., 0.6046, -0.6212, 2.0542],
[-1.2113, -1.0375, 0.7093, ..., -1.4117, -0.1719, -0.8957],
[-0.1419, -0.3375, 0.4329, ..., 1.2914, -0.752 , -0.44 ],
...,
[ 0. , 0. , 0. , ..., 0. , 0. , 0. ],
[ 0. , 0. , 0. , ..., 0. , 0. , 0. ],
[ 0. , 0. , 0. , ..., 0. , 0. , 0. ]])
In [220]: del mmap
只要某个内存映像超出了作用域,它就会被垃圾回收器回收,之前对其所做的任何修改都会被写入磁盘。当打开一个已经存在的内存映像时,仍然需要指明数据类型和形状,因为磁盘上的那个文件只是一块二进制数据而已,没有任何元数据:
In [221]: mmap = np.memmap('mymmap', dtype='float64', shape=(10000, 10000))
In [222]: mmap
Out[222]:
memmap([[ 0.7584, -0.6605, 0.8626, ..., 0.6046, -0.6212, 2.0542],
[-1.2113, -1.0375, 0.7093, ..., -1.4117, -0.1719, -0.8957],
[-0.1419, -0.3375, 0.4329, ..., 1.2914, -0.752 , -0.44 ],
...,
[ 0. , 0. , 0. , ..., 0. , 0. , 0. ],
[ 0. , 0. , 0. , ..., 0. , 0. , 0. ],
[ 0. , 0. , 0. , ..., 0. , 0. , 0. ]])
内存映像可以使用前面介绍的结构化或嵌套dtype。
HDF5及其他数组存储方式
PyTables和h5py这两个Python项目可以将NumPy的数组数据存储为高效且可压缩的HDF5格式(HDF意思是“层次化数据格式”)。你可以安全地将好几百GB甚至TB的数据存储为HDF5格式。要学习Python使用HDF5,请参考pandas线上文档。
A.9 性能建议
使用NumPy的代码的性能一般都很不错,因为数组运算一般都比纯Python循环快得多。下面大致列出了一些需要注意的事项:
- 将Python循环和条件逻辑转换为数组运算和布尔数组运算。
- 尽量使用广播。
- 避免复制数据,尽量使用数组视图(即切片)。
- 利用ufunc及其各种方法。
如果单用NumPy无论如何都达不到所需的性能指标,就可以考虑一下用C、Fortran或Cython(等下会稍微介绍一下)来编写代码。我自己在工作中经常会用到Cython(http://cython.org),因为它不用花费我太多精力就能得到C语言那样的性能。
连续内存的重要性
虽然这个话题有点超出本书的范围,但还是要提一下,因为在某些应用场景中,数组的内存布局可以对计算速度造成极大的影响。这是因为性能差别在一定程度上跟CPU的高速缓存(cache)体系有关。运算过程中访问连续内存块(例如,对以C顺序存储的数组的行求和)一般是最快的,因为内存子系统会将适当的内存块缓存到超高速的L1或L2CPU Cache中。此外,NumPy的C语言基础代码(某些)对连续存储的情况进行了优化处理,这样就能避免一些跨越式的内存访问。
一个数组的内存布局是连续的,就是说元素是以它们在数组中出现的顺序(即Fortran型(列优先)或C型(行优先))存储在内存中的。默认情况下,NumPy数组是以C型连续的方式创建的。列优先的数组(比如C型连续数组的转置)也被称为Fortran型连续。通过ndarray的flags属性即可查看这些信息:
In [225]: arr_c = np.ones((1000, 1000), order='C')
In [226]: arr_f = np.ones((1000, 1000), order='F')
In [227]: arr_c.flags
Out[227]:
C_CONTIGUOUS : True
F_CONTIGUOUS : False
OWNDATA : True
WRITEABLE : True
ALIGNED : True
UPDATEIFCOPY : False
In [228]: arr_f.flags
Out[228]:
C_CONTIGUOUS : False
F_CONTIGUOUS : True
OWNDATA : True
WRITEABLE : True
ALIGNED : True
UPDATEIFCOPY : False
In [229]: arr_f.flags.f_contiguous
Out[229]: True
在这个例子中,对两个数组的行进行求和计算,理论上说,arr_c会比arr_f快,因为arr_c的行在内存中是连续的。我们可以在IPython中用%timeit来确认一下:
In [230]: %timeit arr_c.sum(1)
784 us +- 10.4 us per loop (mean +- std. dev. of 7 runs, 1000 loops each)
In [231]: %timeit arr_f.sum(1)
934 us +- 29 us per loop (mean +- std. dev. of 7 runs, 1000 loops each)
如果想从NumPy中提升性能,这里就应该是下手的地方。如果数组的内存顺序不符合你的要求,使用copy并传入'C'或'F'即可解决该问题:
In [232]: arr_f.copy('C').flags
Out[232]:
C_CONTIGUOUS : True
F_CONTIGUOUS : False
OWNDATA : True
WRITEABLE : True
ALIGNED : True
UPDATEIFCOPY : False
注意,在构造数组的视图时,其结果不一定是连续的:
In [233]: arr_c[:50].flags.contiguous
Out[233]: True
In [234]: arr_c[:, :50].flags
Out[234]:
C_CONTIGUOUS : False
F_CONTIGUOUS : False
OWNDATA : False
WRITEABLE : True
ALIGNED : True
UPDATEIFCOPY : False
第1章 准备工作
第2章 Python语法基础,IPython和Jupyter
第3章 Python的数据结构、函数和文件
第4章 NumPy基础:数组和矢量计算
第5章 pandas入门
第6章 数据加载、存储与文件格式
第7章 数据清洗和准备
第8章 数据规整:聚合、合并和重塑
第9章 绘图和可视化
第10章 数据聚合与分组运算
第11章 时间序列
第12章 pandas高级应用
第13章 Python建模库介绍
第14章 数据分析案例
附录A NumPy高级应用
附录B 更多关于IPython的内容(完)