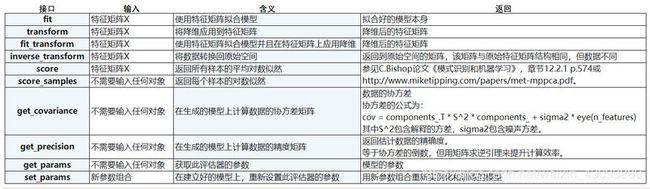
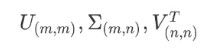sklearn(5)—— 降维算法
1 概述
1.1 从什么叫“维度”说开来
我们不断提到一些语言,比如说:随机森林是通过随机抽取特征来建树,以避免高维计算;再比如说,sklearn中导入特征矩阵,必须是至少二维;在特征工程中,还特地提到了,特征选择的目的是通过降维来降低算法的计算成本……这些语言都很正常地被我用来使用,直到有一天,一个小伙伴问了我,”维度“到底是什么?
对于数组和Series来说,维度就是功能shape返回的结果,shape中返回了几个数字,就是几维。索引以外的数据,不分行列的叫一维(此时shape返回唯一的维度上的数据个数),有行列之分叫二维(shape返回行x列),也称为表。一张表最多二维,复数的表构成了更高的维度。当一个数组中存在2张3行4列的表时,shape返回的是(更高维,行,列)。当数组中存在2组2张3行4列的表时,数据就是4维,shape返回(2,2,3,4)。

数组中的每一张表,都可以是一个特征矩阵或一个DataFrame,这些结构永远只有一张表,所以一定有行列,其中行是样本,列是特征。针对每一张表,维度指的是样本的数量或特征的数量,一般无特别说明,指的都是特征的数量。除了索引之外,一个特征是一维,两个特征是二维,n个特征是n维。

对图像来说,维度就是图像中特征向量的数量。特征向量可以理解为是坐标轴,一个特征向量定义一条直线,是一维,两个相互垂直的特征向量定义一个平面,即一个直角坐标系,就是二维,三个相互垂直的特征向量定义一个空间,即一个立体直角坐标系,就是三维。三个以上的特征向量相互垂直,定义人眼无法看见,也无法想象的高维空间。

降维算法中的”降维“,指的是降低特征矩阵中特征的数量。上周的课中我们说过,降维的目的是为了让算法运算更快,效果更好,但其实还有另一种需求:数据可视化。从上面的图我们其实可以看得出,图像和特征矩阵的维度是可以相互对应的,即一个特征对应一个特征向量,对应一条坐标轴。所以,三维及以下的特征矩阵,是可以被可视化的,这可以帮助我们很快地理解数据的分布,而三维以上特征矩阵的则不能被可视化,数据的性质也就比较难理解。
1.2 sklearn中的降维算法
sklearn中降维算法都被包括在模块decomposition中,这个模块本质是一个矩阵分解模块。在过去的十年中,如果要讨论算法进步的先锋,矩阵分解可以说是独树一帜。矩阵分解可以用在降维,深度学习,聚类分析,数据预处理,低纬度特征学习,推荐系统,大数据分析等领域。在2006年,Netflix曾经举办了一个奖金为100万美元的推荐系统算法比赛,最后的获奖者就使用了矩阵分解中的明星:奇异值分解SVD。
| 类 | 说明 |
| 主成分分析 | |
| decomposition.PCA | 主成分分析(PCA) |
| decomposition. IncrementalPCA | 增量主成分分析(IPCA) |
| decomposition. MiniBatchSparsePCA | 核主成分分析(KPCA) |
| decomposition. MiniBatchSparsePCA | 小批量稀疏主成分分析 |
| decomposition. SparsePCA | 稀疏主成分分析( SparsePCA) |
| decomposition. TruncatedSVD | 截断的SVD( aka LSA) |
| 因子分析 | |
| decomposition.FactorAnalysis | 因子分析(FA) |
| 独立成分分析 | |
| decomposition.FastlCA | 独立成分分析的快速算法 |
| 字典学习 | |
| decomposition. DictionaryLearning | 字典学习 |
| decomposition.MiniBatchDictionaryLearning | 小批量字典学习 |
| decomposition.dict_learning | 字典学习用于矩阵分解 |
| decomposition.dict_learning_online | 在线字典学习用于矩阵分解 |
| 高级矩阵分解 | |
| decomposition. LatentDirichletAllocation | 具有在线变分贝叶斯算法的隐含狄利克雷分布 |
| decomposition.NMF | 非负矩阵分解(NMF) |
| 其他矩阵分解 | |
| decomposition.SparseCoder | 稀疏编码 |
SVD和主成分分析PCA都属于矩阵分解算法中的入门算法,都是通过分解特征矩阵来进行降维,它们也是我们今天要讲解的重点。虽然是入门算法,却不代表PCA和SVD简单:下面两张图是我在一篇SVD的论文中随意截取的两页,可以看到满满的数学公式(基本是线性代数)。

2 PCA与SVD
在降维过程中,我们会减少特征的数量,这意味着删除数据,数据量变少则表示模型可以获取的信息会变少,模型的表现可能会因此受影响。同时,在高维数据中,必然有一些特征是不带有有效的信息的(比如噪音),或者有一些特征带有的信息和其他一些特征是重复的(比如一些特征可能会线性相关)。我们希望能够找出一种办法来帮助我们衡量特征上所带的信息量,让我们在降维的过程中,能够即减少特征的数量,又保留大部分有效信息——将那些带有重复信息的特征合并,并删除那些带无效信息的特征等等——逐渐创造出能够代表原特征矩阵大部分信息的,特征更少的,新特征矩阵。
特征工程中,我们提到过一种重要的特征选择方法:方差过滤。如果一个特征的方差很小,则意味着这个特征上很可能有大量取值都相同(比如90%都是1,只有10%是0,甚至100%是1),那这一个特征的取值对样本而言就没有区分度,这种特征就不带有有效信息。从方差的这种应用就可以推断出,如果一个特征的方差很大,则说明这个特征上带有大量的信息。因此,在降维中,PCA使用的信息量衡量指标,就是样本方差,又称可解释性方差,方差越大,特征所带的信息量越多。
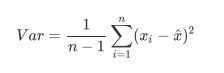
Var代表一个特征的方差,n代表样本量,xi代表一个特征中的每个样本取值,xhat代表这一列样本的均值。
| 面试高危问题 |
|---|
| 方差计算公式中为什么除数是n-1? 这是为了得到样本方差的无偏估计,更多大家可以自己去探索 |
2.1 降维究竟是怎样实现?
class sklearn.decomposition.PCA (n_components=None, copy=True, whiten=False, svd_solver=’auto’, tol=0.0,iterated_power=’auto’, random_state=None)
PCA作为矩阵分解算法的核心算法,其实没有太多参数,但不幸的是每个参数的意义和运用都很难,因为几乎每个参数都涉及到高深的数学原理。为了参数的运用和意义变得明朗,我们来看一组简单的二维数据的降维。
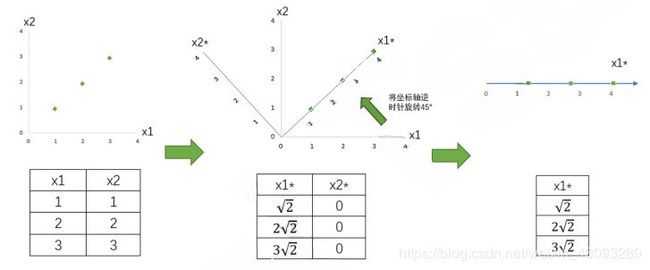
我们现在有一组简单的数据,有特征x1和x2,三个样本数据的坐标点分别为(1,1),(2,2),(3,3)。我们可以让x1和x2分别作为两个特征向量,很轻松地用一个二维平面来描述这组数据。这组数据现在每个特征的均值都为2,方差则等于:
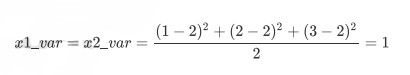
每个特征的数据一模一样,因此方差也都为1,数据的方差总和是2。
现在我们的目标是:只用一个特征向量来描述这组数据,即将二维数据降为一维数据,并且尽可能地保留信息量,即让数据的总方差尽量靠近2。于是,我们将原本的直角坐标系逆时针旋转45°,形成了新的特征向量x1和x2组成的新平面,在这个新平面中,三个样本数据的坐标点可以表示为 ( 2 , 0 ) , ( 2 2 , 0 ) , ( 3 2 , 0 ) (\sqrt{2},0),(2\sqrt{2},0),(3\sqrt{2},0) (2,0),(22,0),(32,0)。可以注意到,x2上的数值此时都变成了0,因此x2明显不带有任何有效信息了(此时x2的方差也为0了)。此时,x2特征上的数据均值是 2 2 2\sqrt{2} 22,而方差则可表示成:

x1*上的数据均值为0,方差也为0。
此时,我们根据信息含量的排序,取信息含量最大的一个特征,因为我们想要的是一维数据。所以我们可以将x2删除,同时也删除图中的x2特征向量,剩下的x1*就代表了曾经需要两个特征来代表的三个样本点。通过旋转原有特征向量组成的坐标轴来找到新特征向量和新坐标平面,我们将三个样本点的信息压缩到了一条直线上,实现了二维变一维,并且尽量保留原始数据的信息。一个成功的降维,就实现了。

不难注意到,在这个降维过程中,有几个重要的步骤:
| 过程 | 二维特征矩阵 | n维特征矩阵 |
|---|---|---|
| 1 | 输入原数据,结构为(3,2) 找出原本的2个特征对应的直角坐标系,本质是找出这2个特征构成的2维平面 |
输入原数据,结构为(m,n) 找出原本的n个特征向量构成的n维空间 |
| 2 | 决定降维后的特征数量:1 | 决定降维后的特征数量:k |
| 3 | 旋转,找出一个新坐标系 本质是找出2个新的特征向量,以及它们构成的新2维平面 新特征向量让数据能够被压缩到少数特征上,并且总信息量不损失太多 |
通过某种变化,找出n个新的特征向量,以及它们构成的新n维空间 |
| 4 | 找出数据点在新坐标系上,2个新坐标轴上的坐标 | 找出原始数据在新特征空间∨中的n个新特征向量上对应的值,即“将数据映射到新空间中” |
| 5 | 选取第1个方差最大的特征向量,删掉没有被选中的特征,成功将2维平面降为1维 | 选取前k个信息量最大的特征,删掉没有被选中的特征,成功将n维空间降为k维 |
在步骤3当中,我们用来找出n个新特征向量,让数据能够被压缩到少数特征上并且总信息量不损失太多的技术就是矩阵分解。PCA和SVD是两种不同的降维算法,但他们都遵从上面的过程来实现降维,只是两种算法中矩阵分解的方法不同,信息量的衡量指标不同罢了。PCA使用方差作为信息量的衡量指标,并且特征值分解来找出空间V。降维时,它会通过一系列数学的神秘操作(比如说,产生协方差矩阵 1 n X X T \frac{1}{n}XX^{T} n1XXT)将特征矩阵X分解为以下三个矩阵,其中 Q Q Q和 Q − 1 Q^{-1} Q−1是辅助的矩阵,Σ是一个对角矩阵(即除了对角线上有值,其他位置都是0的矩阵),其对角线上的元素就是方差。降维完成之后,PCA找到的每个新特征向量就叫做“主成分”,而被丢弃的特征向量被认为信息量很少,这些信息很可能就是噪音。
![]() 而SVD使用奇异值分解来找出空间V,其中Σ也是一个对角矩阵,不过它对角线上的元素是奇异值,这也是SVD中用
而SVD使用奇异值分解来找出空间V,其中Σ也是一个对角矩阵,不过它对角线上的元素是奇异值,这也是SVD中用
来衡量特征上的信息量的指标。U和V^{T}分别是左奇异矩阵和右奇异矩阵,也都是辅助矩阵。
![]() 在数学原理中,无论是PCA和SVD都需要遍历所有的特征和样本来计算信息量指标。并且在矩阵分解的过程之中,会产生比原来的特征矩阵更大的矩阵,比如原数据的结构是(m,n),在矩阵分解中为了找出最佳新特征空间V,可能需要产生(n,n),(m,m)大小的矩阵,还需要产生协方差矩阵去计算更多的信息。而现在无论是Python还是R,或者其他的任何语言,在大型矩阵运算上都不是特别擅长,无论代码如何简化,我们不可避免地要等待计算机去完成这个非常庞大的数学计算过程。因此,降维算法的计算量很大,运行比较缓慢,但无论如何,它们的功能无可替代,它们依然是机器学习领域的宠儿。
在数学原理中,无论是PCA和SVD都需要遍历所有的特征和样本来计算信息量指标。并且在矩阵分解的过程之中,会产生比原来的特征矩阵更大的矩阵,比如原数据的结构是(m,n),在矩阵分解中为了找出最佳新特征空间V,可能需要产生(n,n),(m,m)大小的矩阵,还需要产生协方差矩阵去计算更多的信息。而现在无论是Python还是R,或者其他的任何语言,在大型矩阵运算上都不是特别擅长,无论代码如何简化,我们不可避免地要等待计算机去完成这个非常庞大的数学计算过程。因此,降维算法的计算量很大,运行比较缓慢,但无论如何,它们的功能无可替代,它们依然是机器学习领域的宠儿。
| 思考:降维和特征选择都是特征工程技术,它们有什么不同? |
|---|
| 特征工程中有三种方式:特征提取,特征创造和特征选择。仔细观察上面的降维例子和上周我们讲解过的特征选择,你发现有什么不同了吗? 特征选择是从已存在的特征中选取携带信息最多的,选完之后的特征依然具有可解释性,我们依然知道这个特征在原数据的哪个位置,代表着原数据上的什么含义。 而降维算法,是将已存在的特征进行压缩,降维完毕后的特征不是原本的特征矩阵中的任何一个特征,而是通过某些方式组合起来的新特征。通常来说,在新的特征矩阵生成之前,我们无法知晓降维算法们都建立了怎样的新特征向量,新特征矩阵生成之后也不具有可读性,我们无法判断新特征矩阵的特征是从原数据中的什么特征组合而来,新特征虽然带有原始数据的信息,却已经不是原数据上代表着的含义了。降维算法因此是特征创造(feature creation,或feature construction)的一种。 可以想见,PCA一般不适用于探索特征和标签之间的关系的模型(如线性回归),因为无法解释的新特征和标签之间的关系不具有意义。在线性回归模型中,我们使用特征选择。 |
2.2 重要参数n_components
n_components是我们降维后需要的维度,即降维后需要保留的特征数量,降维流程中第二步里需要确认的k值,一般输入[0, min(X.shape)]范围中的整数。一说到K,大家可能都会想到,类似于KNN中的K和随机森林中的n_estimators,这是一个需要我们人为去确认的超参数,并且我们设定的数字会影响到模型的表现。如果留下的特
征太多,就达不到降维的效果,如果留下的特征太少,那新特征向量可能无法容纳原始数据集中的大部分信息,因此,n_components既不能太大也不能太小。那怎么办呢?
可以先从我们的降维目标说起:如果我们希望可视化一组数据来观察数据分布,我们往往将数据降到三维以下,很多时候是二维,即n_components的取值为2。
2.2.1 迷你案例:高维数据的可视化
- 调用库和模块
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn.decomposition import PCA
- 提取数据集
iris = load_iris()
y = iris.target
X = iris.data
X
'''
array([[5.1, 3.5, 1.4, 0.2],
[4.9, 3. , 1.4, 0.2],
[4.7, 3.2, 1.3, 0.2],
[4.6, 3.1, 1.5, 0.2],
[5. , 3.6, 1.4, 0.2],
[5.4, 3.9, 1.7, 0.4],
[4.6, 3.4, 1.4, 0.3],
[5. , 3.4, 1.5, 0.2],
[4.4, 2.9, 1.4, 0.2],
...
[5.9, 3. , 5.1, 1.8]])
'''
#作为数组,X是几维?
X.shape # (150, 4)
#作为数据表或特征矩阵,X是几维?
import pandas as pd
pd.DataFrame(X) # 如下图
y
'''
array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2])
'''
#调用PCA
pca = PCA(n_components=2) #实例化
pca = pca.fit(X) #拟合模型
X_dr = pca.transform(X) #获取新矩阵
X_dr
'''
array([[-2.68412563, 0.31939725],
[-2.71414169, -0.17700123],
[-2.88899057, -0.14494943],
[-2.74534286, -0.31829898],
[-2.72871654, 0.32675451],
[-2.28085963, 0.74133045],
[-2.82053775, -0.08946138],
...
[ 1.39018886, -0.28266094]])
'''
X_dr.shape # (150, 2)
#也可以fit_transform一步到位
#X_dr = PCA(2).fit_transform(X)
- 可视化
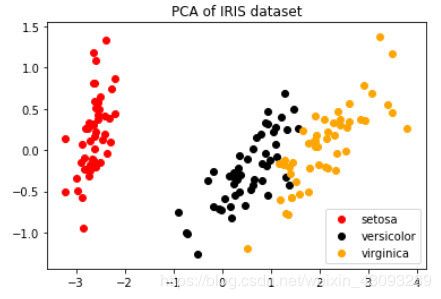
#要将三种鸢尾花的数据分布显示在二维平面坐标系中,对应的两个坐标(两个特征向量)应该是三种鸢尾花降维后的x1和x2,怎样才能取出三种鸢尾花下不同的x1和x2呢?
X_dr[y == 0, 0] #这里是布尔索引,看出来了么?
#要展示三中分类的分布,需要对三种鸢尾花分别绘图
#可以写成三行代码,也可以写成for循环
'''
array([-2.68412563, -2.71414169, -2.88899057, -2.74534286, -2.72871654,
-2.28085963, -2.82053775, -2.62614497, -2.88638273, -2.6727558 ,
-2.50694709, -2.61275523, -2.78610927, -3.22380374, -2.64475039,
-2.38603903, -2.62352788, -2.64829671, -2.19982032, -2.5879864 ,
-2.31025622, -2.54370523, -3.21593942, -2.30273318, -2.35575405,
-2.50666891, -2.46882007, -2.56231991, -2.63953472, -2.63198939,
-2.58739848, -2.4099325 , -2.64886233, -2.59873675, -2.63692688,
-2.86624165, -2.62523805, -2.80068412, -2.98050204, -2.59000631,
-2.77010243, -2.84936871, -2.99740655, -2.40561449, -2.20948924,
-2.71445143, -2.53814826, -2.83946217, -2.54308575, -2.70335978])
'''
iris.target_names # array(['setosa', 'versicolor', 'virginica'], dtype='
plt.figure()
plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0])
plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1])
plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2])
plt.legend() #图例
plt.title('PCA of IRIS dataset')
plt.show()
colors = ['red', 'black', 'orange']
iris.target_names
plt.figure()
for i in [0, 1, 2]:
plt.scatter(X_dr[y == i, 0]
,X_dr[y == i, 1]
,alpha=.7 # 图像透明度(点的透明度0.7)
,c=colors[i]
,label=iris.target_names[i]
)
plt.legend()
plt.title('PCA of IRIS dataset')
plt.show()
鸢尾花的分布被展现在我们眼前了,明显这是一个分簇的分布,并且每个簇之间的分布相对比较明显,也许versicolor和virginia这两种花之间会有一些分类错误,但setosa肯定不会被分错。这样的数据很容易分类,可以遇见,KNN,随机森林,神经网络,朴素贝叶斯,Adaboost这些分类器在鸢尾花数据集上,未调整的时候都可以有
95%上下的准确率。
- 探索降维后的数据
#属性explained_variance,查看降维后每个新特征向量上所带的信息量大小(可解释性方差的大小)
pca.explained_variance_ # array([4.22824171, 0.24267075])
#属性explained_variance_ratio,查看降维后每个新特征向量所占的信息量占原始数据总信息量的百分比
#又叫做可解释方差贡献率
pca.explained_variance_ratio_ # array([0.92461872, 0.05306648])
#大部分信息都被有效地集中在了第一个特征上
pca.explained_variance_ratio_.sum() # 0.9776852063187949
pca_line = PCA().fit(X)
pca_line.explained_variance_ratio_ # array([0.92461872, 0.05306648, 0.01710261, 0.00521218])
import numpy as np
np.cumsum(pca_line.explained_variance_ratio_) # 数据累加
'''
array([0.92461872, 0.97768521, 0.99478782, 1. ])
'''
- 选择最好的n_components:累积可解释方差贡献率曲线
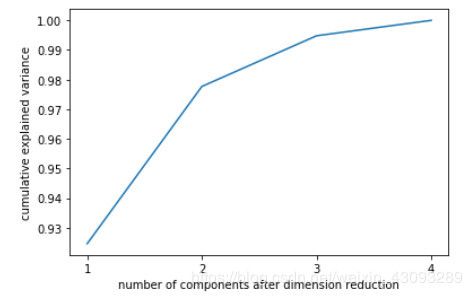
当参数components中不填写任何值,则默认返回min(X.shape)个特征,一般来说,样本量都会大于特征数目,所以什么都不填就相当于转换了新特征空间,但没有减少特征的个数。一般来说,不会使用这种输入方式。但我们却可以使用这种输入方式来画出累计可解释方差贡献率曲线,以此选择最好的n_components的整数取值。
累积可解释方差贡献率曲线是一条以降维后保留的特征个数为横坐标,降维后新特征矩阵捕捉到的可解释方差贡献率为纵坐标的曲线,能够帮助我们决定n_components最好的取值。
import numpy as np
pca_line = PCA().fit(X)
plt.plot([1,2,3,4],np.cumsum(pca_line.explained_variance_ratio_))
plt.xticks([1,2,3,4]) #这是为了限制坐标轴显示为整数
plt.xlabel("number of components after dimension reduction")
plt.ylabel("cumulative explained variance ratio")
plt.show() # 如下图
2.2.2 最大似然估计自选超参数
除了输入整数,n_components还有哪些选择呢?之前我们提到过,矩阵分解的理论发展在业界独树一帜,勤奋智慧的数学大神Minka, T.P.在麻省理工学院媒体实验室做研究时找出了让PCA用最大似然估计(maximum likelihoodestimation)自选超参数的方法,输入“mle”作为n_components的参数输入,就可以调用这种方法。
pca_mle = PCA(n_components="mle")
pca_mle = pca_mle.fit(X)
X_mle = pca_mle.transform(X)
X_mle
'''
array([[-2.68412563, 0.31939725, -0.02791483],
[-2.71414169, -0.17700123, -0.21046427],
[-2.88899057, -0.14494943, 0.01790026],
[-2.74534286, -0.31829898, 0.03155937],
[-2.72871654, 0.32675451, 0.09007924],
[-2.28085963, 0.74133045, 0.16867766],
[-2.82053775, -0.08946138, 0.25789216],
[-2.62614497, 0.16338496, -0.02187932],
[-2.88638273, -0.57831175, 0.02075957],
...
[ 1.39018886, -0.28266094, 0.36290965]])
'''
#可以发现,mle为我们自动选择了3个特征
pca_mle.explained_variance_ratio_.sum() # 0.9947878161267246
#得到了比设定2个特征时更高的信息含量,对于鸢尾花这个很小的数据集来说,3个特征对应这么高的信息含量,并不需要去纠结于只保留2个特征,毕竟三个特征也可以可视化
2.2.3 按信息量占比选超参数
输入[0,1]之间的浮点数,并且让参数svd_solver ==‘full’,表示希望降维后的总解释性方差占比大于n_components指定的百分比,即是说,希望保留百分之多少的信息量。比如说,如果我们希望保留97%的信息量,就可以输入n_components = 0.97,PCA会自动选出能够让保留的信息量超过97%的特征数量。
pca_f = PCA(n_components=0.97,svd_solver="full")
pca_f = pca_f.fit(X)
X_f = pca_f.transform(X)
pca_f.explained_variance_ratio_ #array([0.92461872, 0.05306648])
pca_f.explained_variance_ratio_.sum() #0.9776852063187949
2.3 重要参数svd_solver
2.3.1 PCA中的SVD哪里来?
svd_solver是奇异值分解器的意思,为什么PCA算法下面会有有关奇异值分解的参数?不是两种算法么?我们之前曾经提到过,PCA和SVD涉及了大量的矩阵计算,两者都是运算量很大的模型,但其实,SVD有一种惊人的数学性质,即是它可以跳过数学神秘的宇宙,不计算协方差矩阵,直接找出一个新特征向量组成的n维空间,而这个n维空间就是奇异值分解后的右矩阵 V T V^{T} VT(所以一开始在讲解降维过程时,我们说”生成新特征向量组成的空间 “V”,并非巧合,而是特指奇异值分解中的矩阵 V T V^{T} VT)。
传统 SVD: X → 数学神秘的宇宙 → U Σ V T UΣV^{T} UΣVT
开挂的 SVD:X → 一个简化非常多的数学过程 → V T V^{T} VT
右奇异矩阵 V T V^{T} VT有着如下性质:
![]()
k就是n_components,是我们降维后希望得到的维度。若X为(m,n)的特征矩阵, V T V^{T} VT就是结构为(n,n)的矩阵,取这个矩阵的前k行(进行切片),即将V转换为结构为(k,n)的矩阵。而V_{(k,n)}与原特征矩阵X相乘,即可得到降维后的特征矩阵X_dr。这是说,奇异值分解可以不计算协方差矩阵等等结构复杂计算冗长的矩阵,就直接求出新特征空间和降维后的特征矩阵。
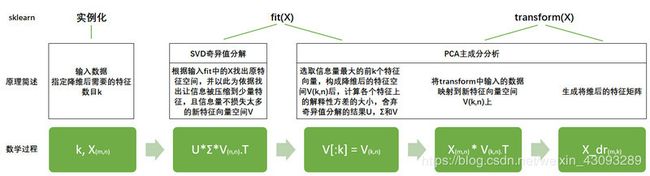
简而言之,SVD在矩阵分解中的过程比PCA简单快速,虽然两个算法都走一样的分解流程,但SVD可以作弊耍赖。但是遗憾的是,SVD的信息量衡量指标比较复杂,要理解”奇异值“远不如理解”方差“来得容易,因此,sklearn将降维流程拆成了两部分:一部分是计算特征空间V,由奇异值分解完成,另一部分是映射数据和求解新特征矩阵,由主成分分析完成,实现了用SVD的性质减少计算量,却让信息量的评估指标是方差,具体流程如下图:
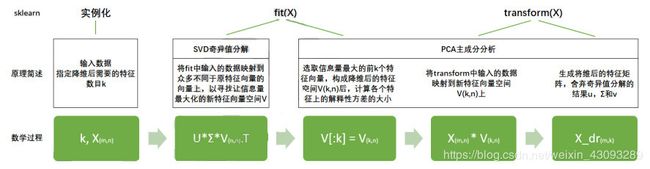
讲到这里,相信大家就能够理解,为什么PCA的类里会包含控制SVD分解器的参数了。通过SVD和PCA的合作, sklearn实现了一种计算更快更简单,但效果却很好的“合作降维”。很多人理解SVD,是把SVD当作PCA的一种求解方法,其实指的就是在矩阵分解时不使用PCA本身的特征值分解,而使用奇异值分解来减少计算量。这种方法确实存在,但在 sklearn中,矩阵U和Σ虽然会被计算出来(同样也是一种比起PCA来说简化非常多的数学过程,不产生协方差矩阵),但完全不会被用到,也无法调取查看或者使用,因此我们可以认为,U和Σ在fit过后就被遗弃了。奇异值分解追求的仅仅是V,只要有了V,就可以计算岀降维后的特征矩阵。在 transform过程之后,fit中奇异值分解的结果除了V(k,n)以外,就会被舍弃,而V(k,n)会被保存在属性 components_当中,可以调用查看。
PCA(2).fit(X).components_
'''
PCA(2).fit(X).components_
array([[ 0.36138659, -0.08452251, 0.85667061, 0.3582892 ],
[ 0.65658877, 0.73016143, -0.17337266, -0.07548102]])
'''
PCA(2).fit(X).components_.shape # V(k,n)
# (2, 4)
2.3.2 重要参数 svd_solver 与 random_state
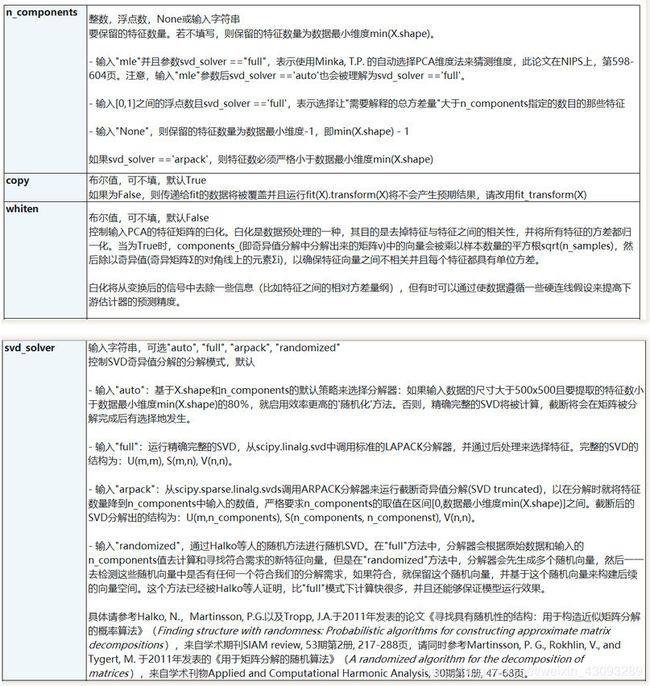
参数 svd_solver 是在降维过程中,用来控制矩阵分解的一些细节的参数。有四种模式可选:“auto”, “full”, “arpack”,“randomized”,默认”auto"。
-
"auto":基于 X.shape 和 n_components 的默认策略来选择分解器:如果输入数据的尺寸大于 500x500 且要提取的特征数小于数据最小维度 min(X.shape)的 80%,就启用效率更高的”randomized“方法。否则,精确完整 的 SVD 将被计算,截断将会在矩阵被分解完成后有选择地发生。
-
"full":从 scipy.linalg.svd 中调用标准的 LAPACK 分解器来生成精确完整的 SVD,**适合数据量比较适中,计算时 间充足的情况,**生成的精确完整的 SVD 的结构为:
- "arpack":从 scipy.sparse.linalg.svds 调用 ARPACK 分解器来运行截断奇异值分解(SVD truncated),分解时就将特征数量降到 n_components中输入的数值k,**可以加快运算速度,适合特征矩阵很大的时候,**但一般用于 特征矩阵为稀疏矩阵的情况,此过程包含一定的随机性。截断后的SVD分解出的结构为:
- "randomized",通过 Halko 等人的随机方法进行随机 SVD。在"full"方法中,分解器会根据原始数据和输入的 n_components 值去计算和寻找符合需求的新特征向量,但是在"randomized"方法中,分解器会先生成多个 随机向量,然后一一去检测这些随机向量中是否有任何一个符合我们的分解需求,如果符合,就保留这个随机向量,并基于这个随机向量来构建后续的向量空间。这个方法已经被 Halko 等人证明,比"full"模式下计算快 很多,并且还能够保证模型运行效果。适合特征矩阵巨大,计算量庞大的情况。
而参数 random_state 在参数 svd_solver 的值为"arpack" or "randomized"的时候生效,可以控制这两种 SVD 模式中的随机模式。通常我们就选用”auto“,不必对这个参数纠结太多。
2.3.3 重要属性 components_
现在我们了解了,V(k,n)是新特征空间,是我们要将原始数据进行映射的那些新特征向量组成的矩阵。我们用它来 计算新的特征矩阵,但我们希望获取的毕竟是 X_dr,为什么我们要把 V(k,n)这个矩阵保存在 n_components 这个属 性当中来让大家调取查看呢?
我们之前谈到过 PCA 与特征选择的区别,即特征选择后的特征矩阵是可解读的,而 PCA 降维后的特征矩阵式不可解读的:PCA 是将已存在的特征进行压缩,降维完毕后的特征不是原本的特征矩阵中的任何一个特征,而是通过某些方式组合起来的新特征。通常来说,**在新的特征矩阵生成之前,我们无法知晓 PCA 都建立了怎样的新特征向量,新特征矩阵生成之后也不具有可读性,**我们无法判断新特征矩阵的特征是从原数据中的什么特征组合而来,新特征虽然带有原始数据的信息,却已经不是原数据上代表着的含义了。
但是其实,在矩阵分解时,PCA 是有目标的:在原有特征的基础上,找出能够让信息尽量聚集的新特征向量。在 sklearn 使用的 PCA 和 SVD 联合的降维方法中,这些新特征向量组成的新特征空间其实就是 V(k,n)。当 V(k,n)是数字 时,我们无法判断 V(k,n)和原有的特征究竟有着怎样千丝万缕的数学联系。但是,如果原特征矩阵是图像,V(k,n)这 个空间矩阵也可以被可视化的话,我们就可以通过两张图来比较,就可以看出新特征空间究竟从原始数据里提取了什么重要的信息。
让我们来看一个,人脸识别中属性 components_的运用。
- 导入需要的库和模块
from sklearn.datasets import fetch_lfw_people # 人脸数据集(7个人脸,1000多张张片组成)
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import numpy as np
- 实例化数据集,探索数据
faces = fetch_lfw_people(min_faces_per_person=60) # 数据实例化(第一次实例化需要点时间)
# 换成特征矩阵之后,这个矩阵是什么样?
faces.data.shape # (1348, 2914)
# 行是样本
# 列是样本相关的所有特征
# 怎样理解这个数据的维度?
faces.images.shape # (1348, 62, 47)
# 1348 是矩阵中图像的个数
# 62 是每个图像的特征矩阵的行
# 47 是每个图像的特征矩阵的列
X = faces.data # 用于画图的矩阵
备注: 实例化的时候报错 出现ImportError: The Python Imaging Library (PIL) is required to load data from jpeg files
解决办法:
conda install Pillow
- 看看图像什么样?将原特征矩阵进行可视化
# 数据本身是图像,和数据本身只是数字,使用的可视化方法不同
# 创建画布和子图对象
fig, axes = plt.subplots(4,5
,figsize=(8,4) # 画布的尺寸和比例
,subplot_kw = {"xticks":[],"yticks":[]} # 不要显示坐标轴
) # 结果如下图
fig # 生成一张纸
axes # 生成对象的数组
'''
array([[,
,
,
,
],
[,
,
,
,
],
[,
,
,
,
],
[,
,
,
,
]],
dtype=object)
'''
axes.shape #(4, 5)
axes[0][0].imshow(faces.images[0,:,:]) # 取出image中除了1348 外的另外所有的像素 即 62,47
# 输出 ),
(1, ),
(2, ),
(3, ),
(4, ),
(5, ),
(6, ),
(7, ),
(8, ),
(9, ),
(10, ),
(11, ),
(12, ),
(13, ),
(14, ),
(15, ),
(16, ),
(17, ),
(18, ),
(19, )]
'''
# 填充图像
for i,ax in enumerate(axes.flat):
ax.imshow(faces.images[i,:,:]
,cmap="gray" # 选择色彩模式
)
# 运行后不会又返回结果,需要给画布才能出结果
# 创建画布和子图对象
fig, axes = plt.subplots(4,5
,figsize=(8,4) # 画布的尺寸和比例
,subplot_kw = {"xticks":[],"yticks":[]} # 不要显示坐标轴
)
# 填充图像
for i,ax in enumerate(axes.flat):
ax.imshow(faces.images[i,:,:]
,cmap="gray" # 选择色彩模式
)# 结果如下图所示
# 创建画布和子图对象
fig, axes = plt.subplots(3,8
,figsize=(8,4) # 画布的尺寸和比例
,subplot_kw = {"xticks":[],"yticks":[]} # 不要显示坐标轴
)
# 填充图像
for i,ax in enumerate(axes.flat):
ax.imshow(faces.images[i,:,:]
,cmap="gray" # 选择色彩模式
)# 结果如下图所示
'''
https://matplotlib.org/tutorials/colors/colormaps.html 提供的色彩
参考案例:
cmaps['Perceptually Uniform Sequential'] = [
'viridis', 'plasma', 'inferno', 'magma', 'cividis']
cmaps['Sequential'] = [
'Greys', 'Purples', 'Blues', 'Greens', 'Oranges', 'Reds',
'YlOrBr', 'YlOrRd', 'OrRd', 'PuRd', 'RdPu', 'BuPu',
'GnBu', 'PuBu', 'YlGnBu', 'PuBuGn', 'BuGn', 'YlGn']
'''
- 建模降维,提取新特征空间矩阵
#原本有2900维,我们现在来降到150维
pca = PCA(150).fit(X)
# V只有空间特征,没有数据(只有骨头没有肉)
V = pca.components_
V.shape # (150, 2914)
- 将新特征空间矩阵可视化
V[0].shape #(2914,)
V[0].reshape(62,47).shape # 将2914维的数据转化为62行47列的二维数组
# (62,47)
fig, axes = plt.subplots(3
,8
,figsize=(8,4)
,subplot_kw = {"xticks":[],"yticks":[]}
)
for i, ax in enumerate(axes.flat):
ax.imshow(V[i,:].reshape(62,47),cmap="gray") # 结果如下图所示
这张图稍稍有一些恐怖,但可以看出,比起降维前的数据,新特征空间可视化后的人脸非常模糊,这是因为原始数据还没有被映射到特征空间中。但是可以看出,整体比较亮的图片,获取的信息较多,整体比较暗的图片,却只能看见黑漆漆的一块。在比较亮的图片中,眼睛,鼻子,嘴巴,都相对清晰,脸的轮廓,头发之类的比较模糊。
这说明,新特征空间里的特征向量们,大部分是"五官"和"亮度"相关的向量,所以新特征向量上的信息肯定大部分是由原数据中和"五官"和"亮度"相关的特征中提取出来的。到这里,我们通过可视化新特征空间 V,解释了一部分降维后的特征:虽然显示出来的数字看着不知所云,但画出来的图表示,这些特征是和”五官“以及”亮度“有关的。这也再次证明了,PCA 能够将原始数据集中重要的数据进行聚集。
2.4 重要接口 inverse_transform
在上周的特征工程课中,我们学到了神奇的接口 inverse_transform,可以将我们归一化,标准化,甚至做过哑变 量的特征矩阵还原回原始数据中的特征矩阵,这几乎在向我们暗示,任何有 inverse_transform 这个接口的过程都 是可逆的。PCA 应该也是如此。在 sklearn 中,我们通过让原特征矩阵 X 右乘新特征空间矩阵 V(k,n)来生成新特征矩 阵 X_dr,那理论上来说,让新特征矩阵 X_dr 右乘V(k,n)的逆矩阵 V ( k , n ) − 1 V^{-1}_{(k,n)} V(k,n)−1 ,就可以将新特征矩阵 X_dr 还原为 X。那 sklearn 是否这样做了呢?让我们来看看下面的案例。
2.4.1 迷你案例:用人脸识别看 PCA 降维后的信息保存量
人脸识别是最容易的,用来探索 inverse_transform 功能的数据。我们先调用一组人脸数据 X(m,n),对人脸图像进行绘制,然后我们对人脸数据进行降维得到 X_dr,之后再使用inverse_transform(X_dr)返回一个 X_inverse(m,n),并对这个新矩阵中的人脸图像也进行绘制。如果 PCA 的降维过程是可逆的,我们应当期待 X(m,n)和 X_inverse(m,n) 返回一模一样的图像,即携带一模一样的信息。
- 导入需要的库和模块(与2.3.3 步骤一致)
from sklearn.datasets import fetch_lfw_people # 人脸数据集(7个人脸,1000多张张片组成)
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import numpy as np
- 实例化数据集,探索数据
faces = fetch_lfw_people(min_faces_per_person=60) # 数据实例化(第一次实例化需要点时间)
# 换成特征矩阵之后,这个矩阵是什么样?
faces.data.shape # (1348, 2914)
# 怎样理解这个数据的维度?
faces.images.shape # (1348, 62, 47)
X = faces.data # 用于画图的矩阵
- 建模降维,获取降维后的特征矩阵 X_dr
pca = PCA(150)
X_dr = pca.fit_transform(X) # 拟合+提取结果
X_dr.shape # (1348, 150)
- 将降维后矩阵用 inverse_transform 返回原空间
X_inverse = pca.inverse_transform(X_dr)
X_inverse.shape # (1348, 2914)
- 将特征矩阵 X 和 X_inverse 可视化
fig, ax = plt.subplots(2,10
,figsize=(10,2.5)
,subplot_kw = {"xticks":[],"yticks":[]}
)
#和2.3.3节中的案例一样,我们需要对子图对象进行遍历的循环,来将图像填入子图中
#那在这里,我们使用怎样的循环?
#现在我们的ax中是2行10列,第一行是原数据,第二行是inverse_transform后返回的数据
#所以我们需要同时循环两份数据,即一次循环画一列上的两张图,而不是把ax拉平
for i in range(10):
ax[0,i].imshow(faces.images[i,:,:], cmap="binary_r")
ax[1,i].imshow(X_inverse[i].reshape(62,47),cmap="binary_r")
# 结果如下图所示
可以明显看出,这两组数据可视化后,由降维后再通过 inverse_transform 转换回原维度的数据画出的图像和原数 据画的图像大致相似,但原数据的图像明显更加清晰。这说明,inverse_transform 并没有实现数据的完全逆转。 这是因为,在降维的时候,部分信息已经被舍弃了,X_dr 中往往不会包含原数据 100%的信息,所以在逆转的时 候,即便维度升高,原数据中已经被舍弃的信息也不可能再回来了。所以,降维不是完全可逆的。
Inverse_transform 的功能,是基于 X_dr 中的数据进行升维,将数据重新映射到原数据所在的特征空间中,而并非 恢复所有原有的数据。但同时,我们也可以看出,降维到 300 以后的数据,的确保留了原数据的大部分信息,所以图像看起来,才会和原数据高度相似,只是稍稍模糊罢了。
2.4.2 迷你案例:用 PCA 做噪音过滤
降维的目的之一就是希望抛弃掉对模型带来负面影响的特征,而我们相信,带有效信息的特征的方差应该是远大于噪音的,所以相比噪音,有效的特征所带的信息应该不会在 PCA 过程中被大量抛弃。inverse_transform 能够在不 恢复原始数据的情况下,将降维后的数据返回到原本的高维空间,即是说能够实现”保证维度,但去掉方差很小特征所带的信息“。利用 inverse_transform 的这个性质,我们能够实现噪音过滤。
- 导入所需要的库和模块
from sklearn.datasets import load_digits
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import numpy as np
- 导入数据,探索数据
digits = load_digits() # 手写数字数据集
digits.data.shape # (1797, 64)
set(digits.target.tolist()) # 去重显示标签
'''
{0, 1, 2, 3, 4, 5, 6, 7, 8, 9}
'''
digits.images.shape # (1797, 8, 8)
- 定义画图函数
def plot_digits(data):
# data的结构必须是(m,n),并n要能够被分成(8,8)这样的结构
fig,axes = plt.subplots(4,10
,figsize=(10,4)
,subplot_kw = {"xticks":[],"yticks":[]}
)
for i, ax in enumerate(axes.flat):
ax.imshow(data[i].reshape(8,8),cmap="binary")
plot_digits(digits.data) # 结果如下图所示
import numpy as np
rng = np.random.RandomState(42)
#在指定的数据集中,随机抽取服从正态分布的数据
#两个参数,分别是指定的数据集,和抽取出来的正太分布的方差
noisy = rng.normal(digits.data,2)
noisy.shape # (1797, 64)
plot_digits(noisy) # 结果如下图所示
pca = PCA(0.5,svd_solver="full").fit(noisy) #实例化 + 拟合 取出降维后的矩阵特征占有原矩阵的50%
X_dr = pca.transform(noisy)
X_dr.shape # (1797, 6)
- 逆转降维结果,实现降噪
without_noise = pca.inverse_transform(X_dr) # 转为原始空间才可以可视化
plot_digits(without_noise)# 结果如下图所示
2.5 重要接口,参数和属性总结
到现在,我们已经完成了对 PCA 的讲解。我们讲解了重要参数参数 n_components,svd_solver,random_state, 讲解了三个重要属性:components_, explained_variance_以及 explained_variance_ratio_,无数次用到了接口 fit,transform,fit_transform,还讲解了与众不同的重要接口 inverse_transform。所有的这些内容都可以被总结在这张图中:
3 案例:PCA 对手写数字数据集的降维
还记得我们使用的手写数字的数据集吗?数据集结构为(42000, 784),用 KNN 跑一次半小时,得到准确率在 96.6%上下,用随机森林跑一次 12 秒,准确率在 93.8%,虽然 KNN 效果好,但由于数据量太大,KNN 计算太缓慢,所以我们不得不选用随机森林。我们使用了各种技术对手写数据集进行特征选择,最后使用嵌入法 SelectFromModel 选出了 324 个特征,将随机森林的效果也调到了 96%以上。但是,因为数据量依然巨大,还是 有 300 多个特征。今天,我们就来试着用 PCA 处理一下这个数据,看看效果如何。
- 导入需要的模块和库
from sklearn.decomposition import PCA
from sklearn.ensemble import RandomForestClassifier as RFC
from sklearn.model_selection import cross_val_score
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
- 导入数据,探索数据
data = pd.read_csv(r"C:\Users\ASUS\sklearn\digit_recognizor.csv")
X = data.iloc[:,1:] # 取出所有行,取出第一列之后的所有列(从索引为1的列开始都取出来)
y = data.iloc[:,0]# 所有的行,索引为0的那一列都取出来
X.shape # (42000, 784)
**备注:**数据参考digit_recognizor.csv
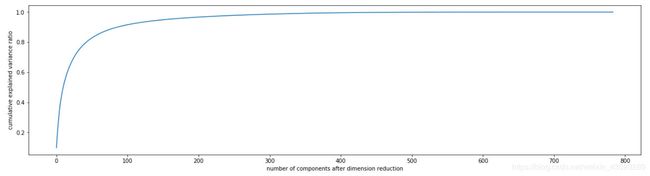
- 画累计方差贡献率曲线,找最佳降维后维度的范围
'''
PCA(): 表示取最小值 min(X.shape) 即:784
'''
pca_line = PCA().fit(X)
plt.figure(figsize=[20,5])
plt.plot(np.cumsum(pca_line.explained_variance_ratio_))
# x轴:降维后的特征数目
plt.xlabel("number of components after dimension reduction")
# y轴:累计可解释方差比例之和
plt.ylabel("cumulative explained variance ratio")
plt.show() # 结果如下图所示
'''
解析:
'''
pca_line.explained_variance_ratio_.shape # 返回784个特征的累计贡献率
# (784,)
#======【TIME WARNING:2mins 30s】======#
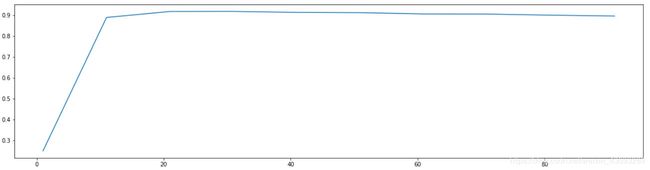
score = []
for i in range(1,101,10):
X_dr = PCA(i).fit_transform(X)
once = cross_val_score(RFC(n_estimators=10,random_state=0) ,X_dr,y,cv=5).mean()
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(1,101,10),score)
plt.show()
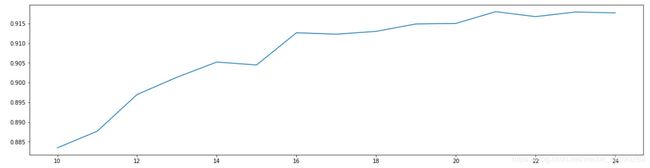
- 细化学习曲线,找出降维后的最佳维度
#======【TIME WARNING:2mins 30s】======#
score = []
for i in range(10,25):
X_dr = PCA(i).fit_transform(X)
once = cross_val_score(RFC(n_estimators=10,random_state=0),X_dr,y,cv=5).mean()
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(10,25),score)
plt.show()
X_dr = PCA(21).fit_transform(X)
#======【TIME WARNING:1mins 30s】======#
cross_val_score(RFC(n_estimators=10,random_state=0),X_dr,y,cv=5).mean() # 0.917619750840417
cross_val_score(RFC(n_estimators=100,random_state=0),X_dr,y,cv=5).mean() # 0.9437386158483362
模型效果还好,跑出了 94.62%的水平,但还是没有我们使用嵌入法特征选择过后的 96%高,有没有什么办法能够提高模型的表现呢?
- 突发奇想,特征数量已经不足原来的 3%,换模型怎么样?
在之前的建模过程中,因为计算量太大,所以我们一直使用随机森林,但事实上,我们知道 KNN 的效果比随机森林更好,KNN 在未调参的状况下已经达到 96%的准确率,而随机森林在未调参前只能达到 93%,这是模型本身的限制带来的,这个数据使用 KNN 效果就是会更好。现在我们的特征数量已经降到不足原来的 3%,可以使用 KNN 了吗?
from sklearn.neighbors import KNeighborsClassifier as KNN
# KNN() :默认k=5
cross_val_score(KNN(),X_dr,y,cv=5).mean() # 0.9674282203473394
- KNN 的 k 值学习曲线
score = []
for i in range(10):
X_dr = PCA(21).fit_transform(X)
once = cross_val_score(KNN(i+1),X_dr,y,cv=5).mean() # 交叉验证
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(10),score)
plt.show()
cross_val_score(KNN(3),X_dr,y,cv=5).mean() # 0.9681423303482433
%%timeit
cross_val_score(KNN(3),X_dr,y,cv=5).mean()
'''
28.5 s ± 746 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
'''
可以发现,原本 785 列的特征被我们缩减到 21 列之后,用 KNN 跑出了目前位置这个数据集上最好的结果。再进行更细致的调整,我们也许可以将 KNN 的效果调整到 98%以上。PCA 为我们提供了无限的可能,终于不用再因为数据量太庞大而被迫选择更加复杂的模型了!