Nature综述:古菌的多样性、生态功能和进化史
![]()
论文信息
原文标题:Diversity, ecology and evolution of Archaea
发表期刊:Nature Microbiology
发表时间:2020.05
第一作者:Baker, Brett J
第一单位:University of Texas System & University of Texas Austin
论文链接:
https://doi.org/10.1038/s41564-020-0715-z
编译:李婷 云南大学国际河流与生态安全研究院
摘要
相比于细菌,我们对古菌的认识仍十分有限。在已有研究中,微生物学家主要依靠纯菌株培养和单基因多样性调查来探究古菌。然而,在目前已发现的27个古菌门中,只有6个门的古菌有代表性物种已成功获得分离培养。基因测序技术和生物信息学的不断发展正在彻底改变有关古菌的认知。基于宏基因组测序获得的未培养古菌类群的基因组促成了新古菌门的发现,这些古菌在全球范围内广泛分布,是地球上的主要生物之一。本综述总结了现有基因组分析、富集研究以及原位分析技术在揭示古菌的代谢和生态功能等方面的研究进展。在此基础上,本文还讨论了探究古菌多样性对生命树构建和古菌早期进化认识的贡献,并提出了真核生物起源的新见解。
引言
所有单细胞非真核微生物在最初都被归类为“原核生物”或后期提出的“Monera”。这些早期的分类学研究根据形态学将细菌和古菌归为一类微生物。随着核酸序列的比较和系统发育分析的发展,我们对微生物的进化亲缘性有了初步了解。这些方法表明,生命应划分为真核生物、细菌和古菌三个界。对古菌的进一步研究揭示了古菌与真核生物在遗传和生化方面的许多相似之处,这使得人们认为古菌与真核生物的进化关系比与细菌的关系更为密切。古菌和真核生物都具有某些细菌缺乏的特征,例如组蛋白、复杂RNA聚合酶和以蛋氨酸起始翻译。随着新古菌类群的不断发现,这些界之间的相似之处越来越多。细菌、古菌和真核生物三者有其独特的遗传、生化和细胞特性。事实上,与细菌相比,古菌可能与真核生物有更多的共同点,如基因组结构、转录和翻译机制,以及它们在细胞壁中缺乏肽聚糖。
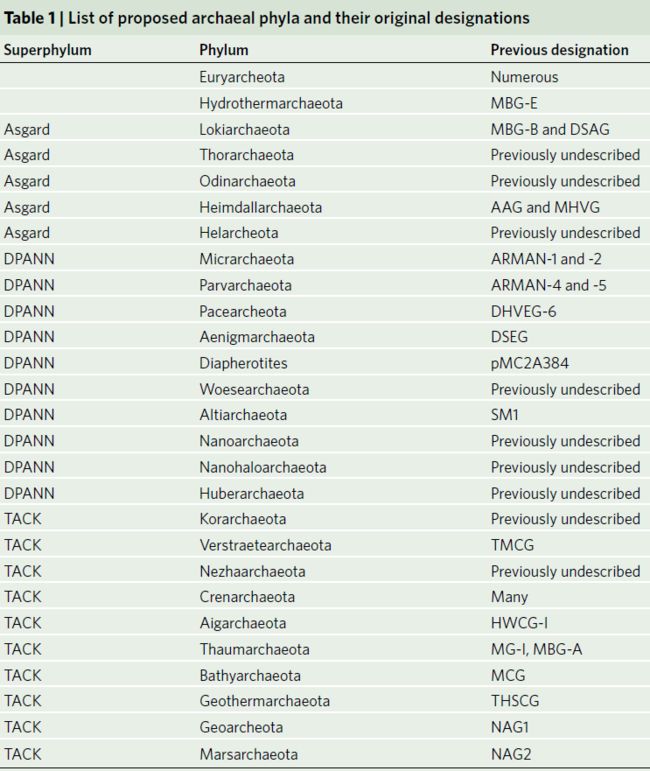
PCR等技术的发展和应用从环境中扩增到大量的16S rRNA基因序列,从而发现了大量未培养的微生物类群,并发现我们对微生物的认识常常受到尚未培养微生物的限制。最初,古菌的rRNA基因分为两个主要的门:广古菌门和泉古菌门。而进一步的研究则发现了不属于这两个门的深层进化分支。DNA测序技术和生物信息学的不断发展使快速重建环境基因组成为可能,最新的相关进展包括了对古菌中20个门的描述。除在rRNA基因多样性调查中发现的基因组外,古菌基因组的重建还促成了几个新分类群的发现,这些类群十分常见却未被发现。这些基因组通过多蛋白系统基因组学对多样性和进化史进行了更有力的比较,并对未分离培养古菌的代谢功能进行了更详细的解析。从目前已获得的古菌基因组数量来看,在对大量微生物类群进行系统的采样研究之前,充分认识古菌的多样性仍有很长的路要走(图1)。通过环境基因组技术所拼接出的古菌基因组数量仍远多于基于纯菌株培养所获得的古菌基因组数量。但是,已有研究基于环境基因组技术将几个古菌门合并为被称为“超级门”的分类群(表1)。目前有已有三个古菌超级门:Asgard、DPANN和 TACK。其中DPANN包括:丙盐古菌门 (Diapherotrites)、小古菌门(Parvarchaeota)、谜古菌门(Aenigmarchaeota)、纳盐古菌门(Nanohaloarchaeota)和纳古菌门(Nanoarchaeota);TACK包括:奇古菌门(Thaumarchaeota)、曙古菌门(Aigarchaeota)、泉古菌门(Crenarchaeota)和初古菌门(Korarchaeota)(表1)。实际上,古菌可被划分为Asgard、DPANN、TACK和广古菌门,而广古菌门则不属于超级门。本文将对这几个古菌类群进行详细的介绍。
除完善有关古菌多样性的认识之外,这些从环境基因组中拼接出的古菌基因组还增强了对古菌代谢和生态作用的理解:如发现了古菌在碳和营养循环中的作用(图2、3)。对生命树进行更多的基因组取样也促进了有关古菌进化史及其在多细胞生命起源中作用的认知,以下将对此进行更详细的讨论。
表1. 古菌超级门及门分类对应表。
图1. 古菌基因组多样性。
图2. 古菌的生态功能
图3. 古菌潜在的代谢功能
广古菌门(Euryarchaeota)
目前,广古菌门含有数量最大,种类最多的已培养古菌类群,主要包括:产甲烷菌、盐杆菌纲(嗜盐)、热原体纲(嗜酸)及嗜热的古丸菌纲、甲烷火菌纲、热球菌纲。其中极端微生物的发现,增进了对此广古菌门的认识,因为它们为生理学研究和基因组分析提供了许多已培养的代表性类群。同时,这些古菌的发现也大幅提升了我们对极端条件下的生物适应性的理解。近年来,原位地球化学和组学研究表明,广古菌不仅参与了甲烷的生产,而且还参与了厌氧甲烷的氧化。此外,这些古菌也参与了其它短链碳氢化合物的厌氧氧化,这表明这些微生物在生物地球化学循环中的作用比想象的要多。
广古菌门的新类群
虽然古菌被认为是微生物生物圈的稀有成员,但某些未培养的广古菌已被证明是生态系统的主要成员。例如,海洋II类和III类古菌(MG-II和MG-III;现称为Poseidoniales和Pontarchaea)是海洋微生物群落的重要组分。SAGMEG(Hadesarchaea)广泛分布于陆地和海底环境中。研究表明,这些重要的类群对全球生物地球化学循环十分重要,突出表明了SAGMEG基因组和其它类群基因组的拼接提升了对广古菌门生物多样性和生态学的理解。
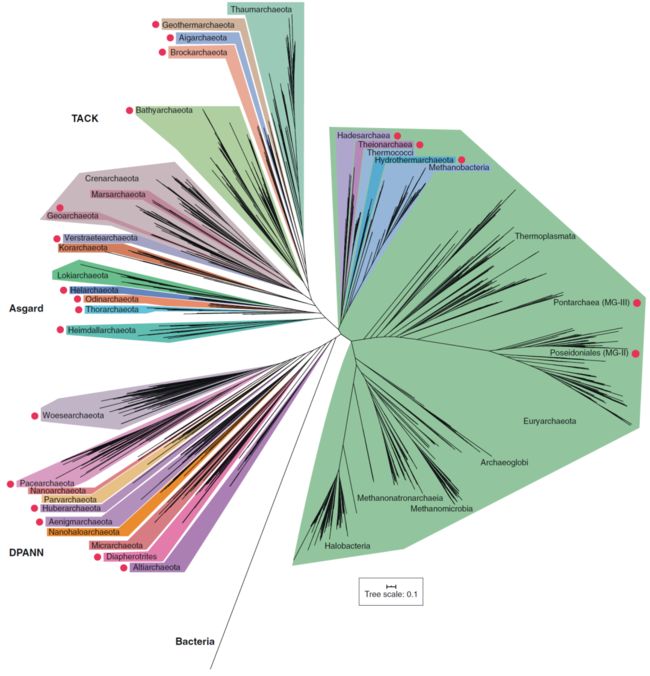
目前已获得几个属于广古菌门的基因组数据。例如,海洋底栖生物E组(MBG-E),被认为是一个单独的门,现在被命名为Hydrothermarchaeota。然而,它们在生命树中的位置仍然存在争议,并且系统发育分析表明它们很可能隶属于广古菌门(图4),甚至与DPANN分支关系密切。其它的广古菌门类群,如Hadesarchaea和Theionarchaea,被认为是不同的门而不是纲。尽管如此,这些类群与其它广古菌群具有代谢共性。同样,以前的研究提出将Altiarchaeales包括在广古菌内,最近已证明它们构成了Altiarchaeota门,并被认为与DPANN古菌关系密切。广古菌内还存在许多其它基于rRNA基因的未培养分支,特别是来自海洋水域和沉积物的分支,并且很快会有基因组数据。
图4. 最新古菌系统发育树
广古菌门的生态作用
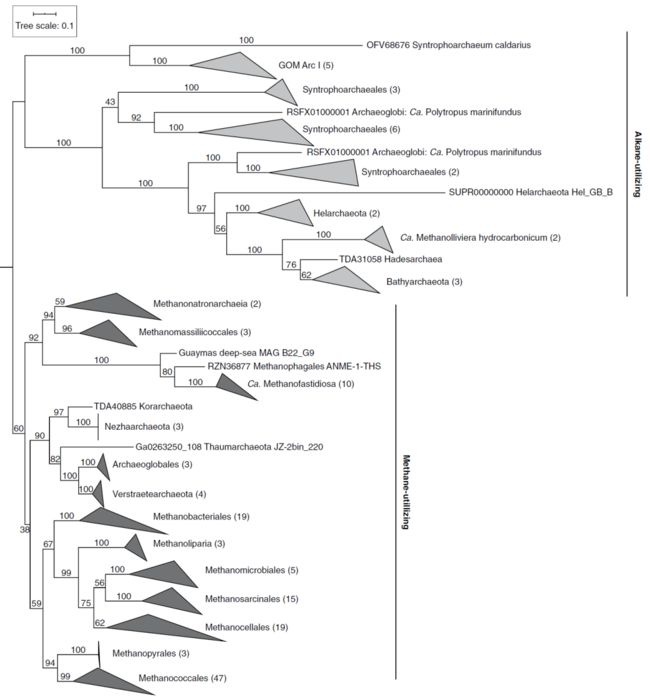
广古菌门在传统上被称为产甲烷菌,但某些广古菌也参与了厌氧甲烷氧化,这些类群被称为厌氧甲烷氧化菌(ANME)。ANME利用核心代谢途径在反方向产生甲烷,但这依赖细菌的作用,通常是硫酸盐还原菌、反硝化菌以及硝酸盐还原菌,使其在能量上处于有利地位。除它们的甲烷代谢能力外,广古菌也能降解其它碳氢化合物。例如,GoM-Arc87包含了与产甲烷类似的基因编码途径,包括甲基辅酶M还原酶(MCR)的同源基因。GoM-Arc87的富集物和HotSeep-1菌群的细菌参与了丁烷的厌氧氧化。这是第一个表明MCR蛋白具有比甲烷更广的底物特异性的实验证据(图5)。而未培养的GoM-Arc1古菌的基因组拼接表明这些微生物具有MCR基因,但在系统遗传学上与参与丁烷或甲烷氧化的微生物不同(图5)。研究表明:GoM-Arc1缺乏丁烷氧化途径,但含有一种新的类似于产甲烷的途径。最近有研究表明,“Candidatus Methanoliparia”同时包含甲烷和非甲烷烷烃类型的MCR基因,表明这些古菌能够通过歧化作用从烷烃代谢中生产甲烷。广古菌除了参与碳氢化合物的转化外,通常还参与硫、氮和铁的循环。例如具有亚硝酸盐和硫还原作用的Hadesarchaea和Theionarchaea等新类群。
图5. 古菌MCR蛋白多样性
此外,其它一些广古菌类群还参与了有机物降解,表明广古菌在自然界中占据着各种不同的生态位。DHVE2能够降解蛋白质以获得能量;MBG-D也被称为DHVE1,含有高含量的胞外肽酶,其活性可直接在缺氧的海洋沉积物中测定。MG-II和MG-III的基因组已从各种宏基因组重建中获得,可能参与了某些高分子有机化合物的循环。部分MG-II生物体含有视紫红质,它们可以利用光来促进能量的产生或促进底物的运输,并且还可以降解蛋白质。
DPANN
Karl Stetter发现了附着在Ignicoccus上的小原始细胞,叫做Nanoarchaeum equitans。遗传分析表明,该物种属于一个新的门:纳古菌门(Nanoarchaeota)。N. equitans的基因组是已知最小的基因组之一,缺乏脂质、辅因子、氨基酸和核苷酸生物合成基因等许多必需基因,这表明N. equitans是一个共生体,依赖于宿主。几年后,从其它小型古菌中发现了包含新的16S rRNA基因的DNA片段。对这些细胞的进行研究分析表明,它们比其它古菌小得多,而且与纳古菌相似,被称为ARMAN。而用于多样性调查的通用PCR引物则忽略了ARMAN。对ARMAN进行宏基因组筛选,得到了3个接近完整的基因组。这种基因组大小和其特性类似于在N. equitans,因此它们在某种程度上也可能是共生体。
ARMAN和纳古菌门被证明是DPANN的一部分。DPANN包括丙盐古菌门 (Diapherotrites)、小古菌门(Parvarchaeota)、谜古菌门(Aenigmarchaeota)、纳盐古菌门(Nanohaloarchaeota)和纳古菌门(Nanoarchaeota),对DPANN进行更广泛的基因组取样,大大扩展了已知DPANN的多样性,包括Woesearchaeota、Pacearchaeota、Huberarchaeota和几个未鉴定的古门。系统发育多样性的增加还表明,ARMAN实际上是两个截然不同的门,即Micrarchaeota和Parvarchaeota,因此将Micrarchaeota作为DPANN古菌的新门。然而,最初生命树并不包括DPANN古菌,随着系统发育学的不断发展,Altiarchaeota 被认为是DPANN的分支,尽管其确切位置仍存在争议。而DPANN古菌的分支位置将影响围绕古菌来源所展开的讨论。
DPANN的生态功能
以前,由于DPANN独特的rRNA基因组成,在多样性调查中一直被所忽略。最初发现时,认为其仅存在于极端的酸性和水热环境,然而,随着进一步的研究发现它们在自然界中无处不在。DPANN在不同环境中的存在表明,它们在生态环境中扮演着重要的角色。由于DPANN古菌有限的遗传和代谢能力,因此它们必须依赖与其它古菌的相互作用来获得必需的生物分子。尽管DPANN在代谢上受到限制,但具有通过糖酵解、β氧化和其它途径利用有机化合物(如碳和脂质)的能力。
简化的基因组和共生生活方式
在DPANN基因组中发现了一些被认为是共生关系的特征。除了基因组小,缺乏多种生物合成途径外,它们的16S rRNA和转移RNA基因中也有独特的内含子,平均基因长度相对较短,这可能是基因组简化的结果。对生物膜中ARMAN进行显微镜观察显示,它们偶尔与属于热原体目(Thermoplasmatales)的古菌有联系。我们已经获得了一种稳定的含有Micrarchaeota和热原体目的共培养物,有利于进一步了解这些相互作用。对这种共培养物的宏转录分析表明,ARMAN主要使用氨基酸,在DPANN中还确定了其它的相互作用关系。
在天然生物膜,热原体目细胞形成长附属物,穿透ARMAN的细胞壁,但其连接的目的尚不清楚。此外,ARMAN含有内部的管状膜结合结构,表明它们可能具有某种形式的分隔。最近,在I. hospitalis中也发现了一种与N. equitans直接接触的内膜系统。除了在ARMAN和其它古菌之间形成联系的附属物外,Altiarchaeota还创造了独特的带倒钩的铁丝网附属物,这些钩似乎与生物膜形成的附属物有关。这些观察结果表明,DPANN与其它古菌之间具有不同的相互作用模式,值得进一步研究。此外,矿井生物膜中的ARMAN有大量的感染病毒,并且许多病毒附着在细胞上时具有两种形态。而最常被感染的ARMAN细胞是那些与其它古菌有接触的细胞。这表明,ARMAN与寄主古菌之间可能存在无法解决的病毒防御相互作用。此外,最近发现,未培养的Micrarchaeota和Parvarchaeota包含CRISPR相关蛋白9(CAS9)病毒防御系统,这可能导致重要的生物技术应用。
TACK
嗜热和嗜酸的泉古菌(Crenarchaeota)是最容易培养的古菌,因此研究最为广泛存在。然而,对自然群落的16S rRNA基因多样性调查发现了一些未培养的、与泉古菌门相关的分支谱系,包括MG-1古菌、MCG古菌、MBG-A和MBG-E,以及TMCG。由于它们在中温沉积物和海水中的广泛分布,它们通常被称为“中温泉古菌”。随着系统发育的不断完善,一些谱系被划分为新门。例如,MG-1现在称为奇古菌门(Thaumarchaeota)。除了泉古菌和奇古菌外,初古菌(Korarchaeota)和曙古菌(Aigarchargeota)的基因组已经被证明是单系的,这四个门组成了TACK的主要部分。TACK的其它组成包括深古菌门(Bathyarchaeota)(原MCG)、Hydrothermarchaeota(原MBG-E)、Geothermarchaeota(原THSCG)和Verstraetearchaeota(原TMCG)。
泉古菌(Crenarchaeota)
泉古菌包含了一些最早从黄石国家公园温泉中分离出来Sulfolobus属的古菌,随后,从高温的陆地和海洋地区获得了更多的培养物。基于16S rRNA基因系统发育分析以及其没有组蛋白的特点,最初将泉古菌归类为与其它古菌不同的属,但是后来的研究在某些泉古菌中发现了组蛋白。大部分泉古菌是厌氧异养菌,利用蛋白质和糖,而少部分则是硫(氧化和还原)循环化学自养生物。研究最为广泛的泉古菌是Sulfolobus spp,现已成为研究超嗜热菌和病毒相互作用的最常用古菌。这些谱系大部分是在富含硫的高温环境中培养的,因此它们主要是嗜热菌超和嗜热菌。Sulfolobus中的可培养部分构成了泉古菌的主要谱系,但NAG1除外。最初,NAG1被认为是一个新的古菌门,被命名为地古菌(Geoarchaeota);然而,深入的系统基因组学研究表明,它们实际上是热变形菌目(Thermoproteales)的分支。
奇古菌门(Thaumarchaeota)
MG-I最初被认为是泉古菌Crenarchaeota的成员,然而,系统发育分析表明它们是一个独特的门,称为奇古菌门Thaumarchaeota。基于宏基因组分析发现了某些MG-I的DNA片段含有氨单加氧酶基因,这表明它们参与了氨氧化亚硝酸盐(硝化的第一步)的过程。而该组共生成员Cenarchaeum symbiosum的近乎完整的基因组显示,它们包含一个完整的碳固定途径,现已证明是能量效率最高的碳固定途径之一。此外,一些MG-I能够氧浓度较低的情况下氧化氨,奇古菌能从尿素和氰酸盐中获得氨。因此,奇古菌受到气候变化的影响,因为它们的活动与N2O的产生密切相关。此外,它们还可以合成甲基膦酸盐。氨氧化奇古菌被认为是数量最多的古菌之一,因为它们在深海和土壤中占很大比例。尽管它们具有相同的核心化学自养代谢,但可以适应各种环境条件,从淡水到盐度超高的咸水,从pH 3.5到pH 8.7,从北极到高温环境。关于奇古菌氨氧化的生物机制仍存在疑问,包括羟胺(NH2OH)氧化的酶和中间产物一氧化氮(NO)的作用。在氨氧化过程中,特别是在低氧环境中,这些中间产物的命运可能对N2O的生成起着重要作用。
深古菌门(Bathyarchaeota)
尽管对海洋沉积物的研究不如海水,但发现海洋沉积物中含有相当多的未培养古菌谱系。在近岸和海洋海底环境中,MCG在古菌DNA库中占主导地位。MCG的第一个功能性遗传信息是基于fosmid克隆文库,该文库表明该群体编码细菌叶绿素合成基因。然而,进一步研究表明,这并非是MCG的一般特征,同时也表明这个群体的某些成员可能有光驱动新陈代谢的能力。在不断的研究分析中,MCG被确定为一门,深古菌门(Bathyarchaeota)。深古菌群可在缺氧的海洋、缺氧的淡水和高温的温泉中存在且数量丰富、种类繁多。由于它们在数量上的优势,认为它们和奇异古菌一样,是地球上数量最多的古菌群之一。
通过宏基因组学,已经从海洋和淡水沉积物中发现了深古菌亚群的基因组。深古菌使矿物质碎屑矿化,这可能使蛋白质降解与氢产生结合。它们的生理活动与蛋白质和纤维素的降解以及二氧化碳的固定有关。此外,一些谱系包含了Mcr基因,这是Mcr基因第一次出现在广古菌群之外的类群。研究还发现Mcr蛋白也参与了丁烷氧化过程,因此深古菌可能可以氧化除甲烷以外的短链碳氢化合物。目前,以木质素和无机碳为碳源,已富集了8组深海古菌群,说明它们能够利用难降解的有机物。深古菌群的高度多样性表明,在这个未培养的门内将会不断有新的发现。
TACK生命树的发展
从厌氧纤维素消化池和深层陆地煤层中进行的宏基因组重建导致了未培养TMCG的恢复,名为Ca.Methanomethylicus spp。对其进行基因组的系统生物学分析表明,它们是一个独特的门,名为Verstraetearchaeota。而Methanomethylicus具有参与甲基营养型甲烷生产的途径,说明古菌碳氢化合物循环的能力存在于广古菌之外。Korarchaeum cryptofilum是初古菌门(Korarchaeota)的第一个进行了基因组重建的成员。近年来,通过宏基因组分析,初古菌基因组的数量和多样性都有所增加。此外,初古菌的某些基因组含有Mcr基因,这些基因被认为与甲烷循环与硫酸盐还原有关。另外一个新的门是曙古菌(Aigarchargeota),其第一个基因组重建的是Caldiarchaeum subterraneum,这表明该门主要与含氧温泉群落有关。基于基因含量,它们可能利用一系列细胞外聚合物,因此在循环溶解有机碳方面十分重要。
Asgard
在全球海洋沉积物多样性调查中发现的另一个广泛分布的古生物群是深海古生物群(DSAG67),也称为MBG-B44,后来被命名为洛基古菌门(Lokiarchaeota)。洛基古菌门的系统发育分析表明,它与真核生物是单系的,这意味着它们在系统树中相关,并且这些古菌和真核生物可能具有共同的祖先。真核生物在古菌中的位置表明,第一批真核细胞是从古菌的祖先那里获得的。进一步支持这一点的是,洛基古菌门包含多种基因,编码与真核肌动蛋白同源的蛋白质,转运所需的核内分选复合物(ESCRT)-I和III复合物以及泛素修饰体系统。一些真核特征蛋白(ESPs),如ESCRT-III复合物的成分,也在其它门中发现,特别是在TACK古菌中。但是,只有非常零散的分布,没有洛基古菌门中的完整。因此,有人认为洛基古菌门可能是真核生物的祖先的后代。
在发现洛基古菌门之后,从河口沉积物中发现了与其相关的新门的基因组,并将其命名为雷神古菌门(Thorarchaeota)。最近,从包括温泉和地下水在内的其它厌氧沉积物中,发现了与洛基古菌门和雷神古菌门相似的基因组,即白神古菌门(Heimdallarchaeota)和奥丁古菌门(Odinarchaeota)。与其它古菌一样,由于与常用PCR引物的不匹配,这些生物在传统的群落研究中也常常被忽视。这些门被命名为Asgard。最近,发现了另一门名为Helarchaeota的基因组。根据对Asgard群落的宏基因组分析,它们属于低丰度(<1%)种群。与洛基古菌门一样,这些新谱系包含多种涉及细胞骨架形成、转运、翻译、转录和降解途径的ESPs。
随着这些ESPs的了解,Asgard提供了关于构成细胞膜的双脂结构进化的见解。古菌的一个识别特征是在它们的脂膜中存在醚键,而在细菌和真核生物中则是酯键。这种差异被称为“脂质分裂”,并引发了许多关于真核细胞进化的讨论,以及细菌和真核细胞的酯链膜独立进化的可能性。然而,最近对洛基古菌门和其它古生门进行的分析表明,这些途径可用于细菌型和真核型酯连接脂质膜的生产,并支持Asgard祖先产生第一个真核细胞的假设。但是,这些Asgard蛋白的确切功能仍处于未知状态。
关于古菌的进化史和它们在早期真核生物进化中作用的研究发现,Asgard古菌的祖先是与蛋白质细菌共生相互作用的古宿主,后来变成了线粒体,形成了早期真核细胞。因此,研究Asgard的生态和代谢能力有利于了解这种共生互动作用的本质。Asgard基因组的代谢途径的比较分析表明,门与门之间存在明显的代谢差异。最近,已经获得了一种洛基古菌(名为Ca. Prometheoarchaeum syntrophicum MK-D1)和Halodesulfovibrio的共培养物。基因组分析和稳定同位素实验证实存在甲酸盐和氢的共生交换。有趣的是,这种浓缩培养物的显微镜检查表明Ca. P. syntrophicum形成新型的附属物,这些附属物似乎缠绕着它们的配体,这可能提供了一种捕获α-蛋白质杆菌祖先线粒体的机制。白神古菌门(Heimdallarchaeota)是另一种与真核生物关系更密切的谱系,它可能是异养生物,也可能依赖于氢、电子或简单的碳化合物的交换。显而易见的是共营养现象不仅限于洛基古菌和白神古菌。例如,Helarchaeota中似乎存在厌氧烷烃氧化,这表明许多Asgard古菌参与了营养相互作用。研究这些相互作用的机制将增进我们对导致真核生物起源的生物学事件的理解。
展望
最早发现的古菌是产甲烷菌或来自其它嗜热环境的古菌类群。因此,我们对古菌代谢作用的理解仅限于产甲烷和硫呼吸。而且最初古菌基因组研究仅限于广古菌门和泉古菌门中的培养代表。然而,最近技术的进步和长期富集研究揭示了未培养生物圈的功能潜力,挑战了关于某个分类单元具有某些功能,以及特定基因组与功能关联程度的假设。最近,基因组分类学数据库(GTDB)的提出,将系统基因组学与分类学定量结合起来,可以将新的基因组放置到分类学结构中。基于新基因组与其最近亲属之间的相对进化距离,从而提供了统一分类层次的可能性。尽管在常用的分类数据库中采用了GTDB,但在获得更深入的基因组样本之前,古门的位置应该被视为是初步的。目前古菌基因组取样的有限性意味着当前的分类体系可能会随着未来更大的取样而显著改变。
尽管在过去的十年里,对古菌的多样性的研究进展迅猛,但是我们仍然需要对自然界中的古菌进行更广泛的基因组研究。正如从宏基因组中拼接的大量基因组一样,肯定还有一些基因组由于rRNA调查的偏差还没有被检测出来。此外,8个门已经通过宏基因组拼接鉴定出来,这些门以前不为人所知,也不为rRNA序列所表征。其中一些由于PCR引物没有针对它们而没有被检测到,而另一些已经被恢复,但是很少有代表性,因此没有给出系统发育命名。这表明rRNA多样性调查忽略了或没有描述自然界存在的大量生物多样性。近年来,从宏群落中重建基因组显著增加了未培育谱系中可用基因组的数量(图4)。这将促进我们对古菌生物学的理解,而且这种分类取样的增加也可能为古菌生物学的进化提供新见解。
近年来,已经在新发现类群的基因组中确定了大量关键生态过程的代谢途径(图3)。代谢途径的存在并不总是等同于活性,可能难以准确地将功能与基因联系起来。因此,需要进行更多的研究以提供原位生理功能测定,包括将大量样品中测得的功能基因与功能联系起来。目前已发展出多种方法来量化群落的活性,包括DNA稳定同位素探针(DNA-SIP)、转录组学、代谢组学和蛋白质组学。基因组学、转录组学、代谢组学和酶分析可以为基因信息预测的功能提供强有力的支持。另一种类似于微放射自显影的新技术称为“生物正交非标准氨基酸标记”(BONCAT),它可以跟踪合成氨基酸在新合成蛋白质的掺入,从而揭示活性细胞。由于活性细胞是荧光的,因此可以将显微镜技术与靶向rRNA的荧光原位杂交(FISH)或荧光激活细胞分选技术(BONCAT-FACS)相结合。此方法提供了一种潜在的高通量技术来确定表型(生长特性和底物使用),并在群落水平上将其与基因型联系起来。将群落水平的基因组重建和活性分析结合,可以在分类水平上提供与微生物介导过程的多样性和代谢途径的联系。
为新基因进行功能注释是解决新古菌类群生态作用的另一大挑战。从环境基因组中扩增的古菌基因的异源表达取得了成功,这显示了此技术的应用前景。古菌生理学的进一步发展将增进我们对生物地球化学循环的理解,并使我们能够更准确地模拟微生物在生态系统中碳和能量的流动。我们获取基因组的能力不断加强以及追踪自然群落活动工具的不断发展,将继续促进古菌生态学和进化史的发展。鉴于近年的研究进展,古菌系统发育树的构建及新类群的发现必将有新发展。
Brett J. Baker, Valerie De Anda, Kiley W. Seitz, Nina Dombrowski, Alyson E. Santoro & Karen G. Lloyd. (2020). Diversity, ecology and evolution of Archaea. Nature Microbiology 5, 887-900, doi: https://doi.org/10.1038/s41564-020-0715-z
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]() 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读