什么是基因测序,为什么需要云计算
【摘要】 唐老师写的技术入门文章,保证小白也能看懂。本文带你领略过去这100年中人类对基因的认识从无到有,并到当下即将进入寻常百姓家的发展历程,以及说明为什么基因测序和云计算会扯上关系。另,本文定位只是科普类文章,目的是带你入门基因测序技术,所以内容篇幅都不会很长。如果你对某个环节的细节感兴趣,你可以继续查找相关的资料深入学习。
1 什么是基因
1.1 探索遗传因子
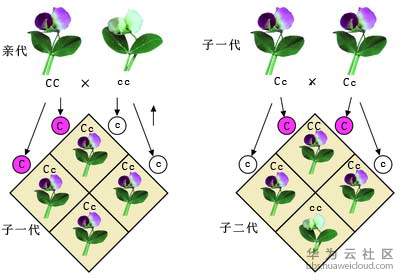
孟德尔种植豌豆的故事相信大家都有听说过,他在1856年至1863年的8年间种下了约5000株豌豆植株并进行杂交实验。孟德尔发现,当纯品系的黄色豌豆和纯品系的绿色豌豆交配时,他们的后代总是产生黄色豌豆。然而,再下一代,绿色豌豆重新出现,绿黄比例为1:3。
他在论文中提出:生物的所有性状都是通过遗传因子来传递的,遗传因子是一些独立的遗传单位。不过直到1900年时,他的研究才被人们所重视,人类开始意识到,也许有一种“遗传因子”在控制者生命的特性。所以在接下来的百年中,科学家们都在寻找这个“遗传因子”的秘密。
1.2 遗传因子与染色体
随后的1879年,德国的科学家在细胞核内发现了一种可以被碱性红色染料染色的“微粒状特殊物质”,也就是现在的“染色体”。此后,科学家们在了解了染色体与细胞分裂的关系后,开始意识到染色体可能是遗传的重要物质。
1903年,美国细胞学家萨顿在实验中发现:染色体的行为与孟德尔的遗传因子的行为是平行的,只要假定遗传因子在染色体上,孟德尔所提出的分离定律和自由组合定律的机制就可以得到合理的解释。那么,遗传因子是否真的存在于染色体上呢?这一推论很快就被美国生物学家摩尔根在1909以实验结果证实。并且实验进一步表明,一条染色体上可以有多个遗传因子。同时,科学家们开始把遗传因子命名为基因。
1.3 DNA双螺旋
虽然DNA在细胞核中很早就被发现,但证明其为遗传物质的决定性实验是1944年艾弗里的肺炎双球菌转化实验。该实验明确证实:DNA是遗传信息的载体。随后1952年赫希和蔡斯进一步证明遗传物质是DNA而不是蛋白质。
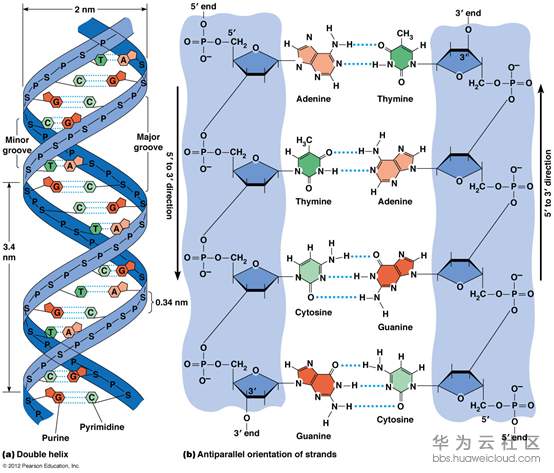
然而那个时候,虽然人们已经知道了脱氧核糖核酸(DNA)可能是遗传物质,但是对于DNA的结构,以及它如何在生命活动中发挥作用的机制还不甚了解。1953年,美国分子生物学家詹姆斯·沃森和英国物理学家佛朗西斯·克里克根据威尔金斯和富兰克林所进行的X射线衍射分析,提出了著名的DNA双螺旋结构模型,进一步说明基因载体就是DNA。
1.4 基因
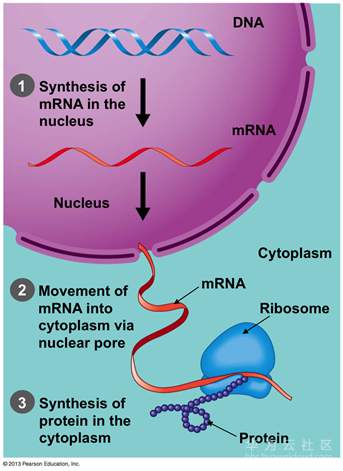
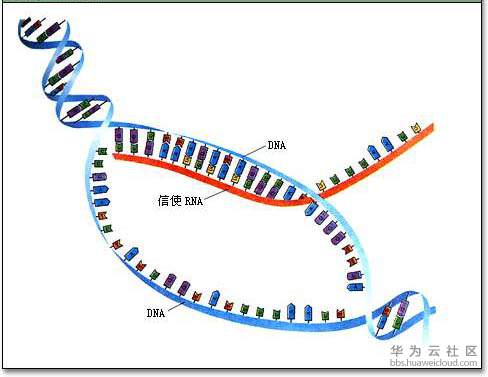
在证明DNA由无数碱基对组成的双螺旋后,那么不同的碱基对顺序,是否就代表了能够影响生物不同的特性呢。 事实结果也确实证明了这一点,这个过程就是基因的转录过程,也就是用基因编码顺序,来控制翻译从而得到不同的蛋白质。
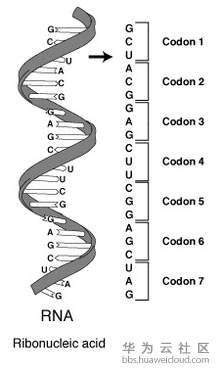
这里插一个有意思的过程,DNA在细胞核内,而蛋白质在细胞核外。那么这种翻译是怎么进行的呢?所以科学家推断肯定有一种可以传递密码的东西,能从细胞核里面跑到细胞核外面。这个信使也就是RNA了,而且RNA是一个单链结构。
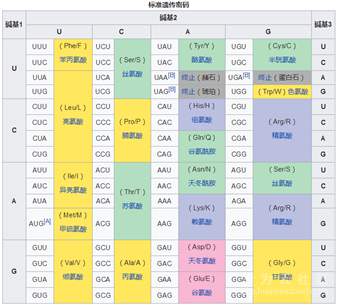
而最最神奇的,就是这个RNA翻译为蛋白质的过程。科学家证实3个编码为一组,可以对应一种氨基酸,用来控制蛋白质的种类(蛋白质由一个或多个由氨基酸残基组成的长链条组成)。
因为有4种符号,又是3个一组,所以总共也就是有64种编码。这个编码表就是上帝的密码表了
PS,如果换个角度看这个密码表,你会发现跟咱们八卦确实有那么一丢丢像(比如双螺旋,64挂)
好了,知道了基因就是神奇的密码,它可以控制生物的特性。接下来的工作就是要破解这个密码,也就是第一步得先得到DNA的具体序列。于是当前的工作重点就跑到了如何测定每种生物的DNA序列。
2 基因怎么测序
2.1 桑格测序
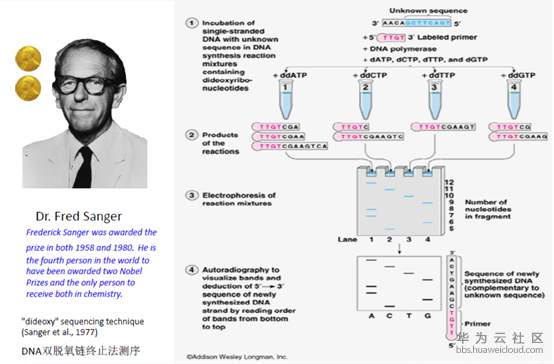
最早的DNA测序技术,由英国生物化学家弗雷德里克·桑格(Frederick Sanger)于1975年发明的。具体过程太技术化,简单概括就是一个一个按需顺序进行检测。这种检测方法可以做到精度非常高(达99.999%),读长(一次最长检测的DNA长度)很长(1000bp)。可以成为后续其他基因检测仪的判断标准,人类基因组计划(HGP)主要就是使用该测序方法完成的。
拿了两块诺贝尔奖的桑格
但是这种测序方法也有很明显的缺点,就是成本昂贵,测序速度也比较慢。这也是为什么人类基因组计划需要美、英、日、德、法、中等国耗时10年时间才完成人类基因图谱的绘制。
2.2 基因参考组
人类基因组计划(Human Genome Project, HGP)是一项规模宏大,跨国跨学科的科学探索工程。其宗旨在于测定组成人类染色体中所包含的30亿个碱基对组成的DNA序列,从而绘制人类基因组图谱,并且找出所有基因在染色体上的位置,达到破译人类遗传信息的最终目的。
该计划于2003年宣布完成,意义巨大。
2.3 二代测序技术
因为不同的人与人之间的基因序列只有不到1%差异。当我们已经有了一份完整的人类基因图谱,那么其他人的基因序列都差不多的。所以就出现了鸟枪法测序(又称散-弹-枪法)。该法的思路独特,好像树林里停了一大群鸟,很多人乱枪射击,在很短的时间内,就可以将林子中的大部分鸟打中。
“鸟枪法”有点类似人们玩的拼图游戏。拼图游戏是将一个完整的画面分成杂乱无章的碎块,然后重新拼装复原。而“鸟枪法”则是先将整个基因组打乱,切成随机碎片,然后测定每个小片段序列,最终利用计算机对这些切片进行排序和组装,并确定它们在基因组中的正确位置。
由于可以将目标先拆分小粒度,然后分段得出结果,最后合并结果。这就好比是大数据中的并行计算,直接导致的结果就是测序性能大大提升。经过不断的技术开发和改进,第二代测序技术开始诞生了。
第二代测序技术在大幅提高了测序速度的同时,还大大地降低了测序成本,并且保持了高准确性,以前完成一个人类基因组的测序需要3年时间,而使用二代测序技术则仅仅需要1周,但其序列读长方面比起第一代测序技术则要短很多,大多只有100bp-150bp。
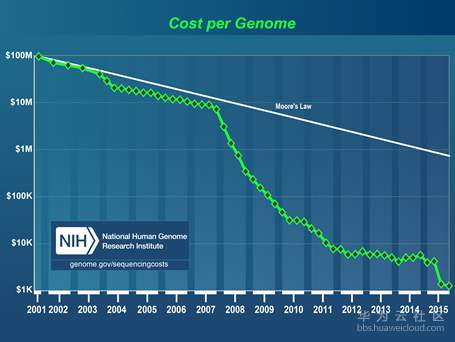
二代测序技术最大的价值在于它极大的降低了测序的成本,是的基因测序技术普及并开始进入普通消费者的视野。
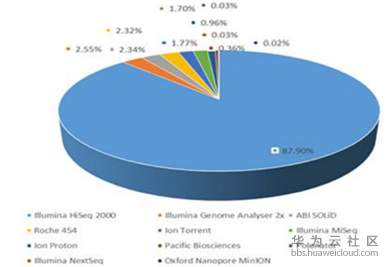
而二代测序技术中,属Illumina公司的测序仪,市场占有率最高,属于绝对的控制地位。
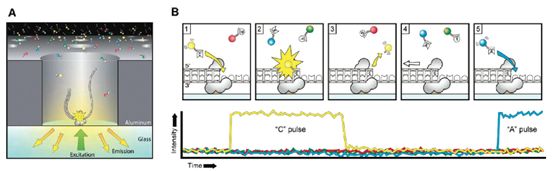
这里有个介绍Illumina测序原理的视频(3分钟),强烈推荐打开看一看,对理解二代测序原理将有很大的帮助:http://v.youku.com/v_show/id_XNzEzNzk1NTA0.html
2.4 三代测序技术
虽然目前测序市场还是以二代测序技术为主要占有者,但是新的测序方法也不短出现。以PacBio公司的SMRT和Oxford Nanopore Technologies的纳米孔单分子测序技术为标志,被称之为第三代测序技术。与前两代相比,最大的特点就是 单分子测序,测序过程无需进行PCR扩增,超长读长,以PacBio SMRT技术的测序读长为例,平均达到10Kb-15Kb,是二代测序技术的100倍以上。
第二代测序技术的优点是通量大大提升,成本大大减低,使得昔日王榭堂前燕,可以飞入寻常百姓家。总之,只有变成白菜价,才能真正对大众有意义;但它的缺点是所引入PCR过程会在一定程度上增加测序的错误率,并且具有系统偏向性,同时读长也比较短。第三代测序技术是为了解决第二代所存在的缺点而开发的,它的根本特点是单分子测序,不需要任何PCR的过程,这是为了能有效避免因PCR偏向性而导致的系统错误,同时提高读长,但这个技术还不是很成熟,需要再进化,成本也偏高。
3 云计算与基因测序
3.1 测序重组的计算
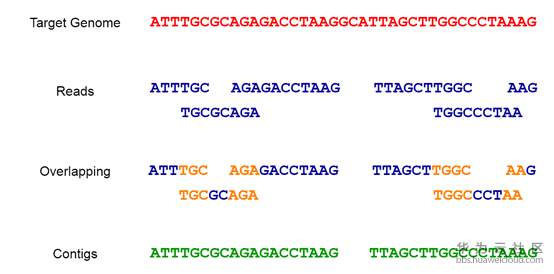
回到当前最普遍的二代测序技术。从测序原理可以知道,整个过程就是先目标打碎,然后重新拼接还原的过程。好比给你一副拼图,先打散,再照着封面参考图重新把图拼起来。听着很比较简单,我们仔细打开来看一看。
-
拼图片数巨大。基因链的长度是非常的长的,30亿bp。而每个小片段只有150bp,也就是一条链就会有至少1千万片的片段。
-
好几副拼图凑一起。测序过程中,需要将同一条链,进行复制,这样打碎后还原,可以保证结果可以覆盖目标基因链的绝大部分。
-
重叠的拼图片。由于测序仪的1和2步骤,导致会存在大量重复片段
-
缺少部分拼图片。测序化学试剂原因,测序后期的精度降低,导致部分低质量的结果是要被丢弃,不能参与还原过程的。
-
存在干扰拼图片。由于每一个人的基因肯定会有不同,所以目标基因与参考组肯定会存在差异。另外,采样的时候,也许会有其他细菌杂志干扰。
-
拼图本身就存在重复。同一条基因链中,本身就存在大量的重复序列。
以上整个过程,好比给你一座堆满了拼图片的大山,让你拼一幅图出来。这个工作量,你懂的。这里找了一个比较合适图,各位感受一下:
于是,重点来了。还原整个拼图的过程,就是使用计算机,开始各种算法,各种运算。
这里我们打开局部组装的细节看一看:
一开始由测序仪随机打断,然后软件进行“对齐”,对齐之后,需要“去重”,去重之后,再进行“合并”。最终得到想要测序的目标序列的顺序。
同时,关于拼图还原的方法,也是百家争艳。你喜欢先拼局部,我喜欢全图先找位置。各种软件各种版本,纷至沓来。。。
站在更高的角度,对于当前基因测序情况的总结就是:利用各种软件进行各种运算,来处理海量的基因数据。
3.2 传统计算方法
首先,一个人30X的覆盖深度的全基因组测序仪下机数据,可以到200G。中间运算过程中,还会变大至600G,整个重组计算过程大概需要20多个小时。所以每个基因测序都是一个极其消耗计算资源的过程。随着基因测序技术的普及,这个测序业务量也是在逐年增长。
早期为了运行如此大规模的计算存储过程,一般有两种方式。
1. 采购高性能计算机,提高单机性能往上垒。
2. 购买服务器集群,安装HPC管理软件。利用HPC多任务并发提升计算能力。
然而实际使用上,基因厂商通常会面临不少问题:
首先,不管是直接购买物理机或者自建HPC集群,成本都是非常高的。在前期测序业务还么有开展时,就需要投入大量资金进行设备采购维护,无疑提高了一个测序公司的风险成本。
其次,由于测序业务量本身是存在波动的,这会导致服务器的数量不能很好的控制。服务器不足则无法满足业务高峰时的测序任务。如果采购可以满足最大峰值的集群,那么在业务量不能时刻保持高峰时,就会存在很大的成本浪费。
最后一个就是运维成本。一个测序业务作为主要发展方向的公司,却需要投入大量人力去维护计算环境。包括软件的安装&升级,故障的定位,环境的恢复等等。这些都无疑增大了运营成本。
3.3 云上计算方法
随着云计算技术的进步,目前公有云平台已经可以为测序公司提供大规模且极易扩展的数据计算分析能力。而云计算带给测序厂商的,不仅是随时可以扩展或者随时可以销毁的资源,还减少了厂商对于前期硬件设备的投入,以及计算环境日益增长的运维成本。
目前厂商将部分测序业务搬迁放在云上执行,已经是一种非常普遍的现象。同时,越来越多的专注于基因测序的公有云也不断出现,比如DNAnexus、Illumina’s BaseSpace。其中又有一部分是采用云上云的方式,也就是在基础公有云上构建领域公有云。
比如SevenBridge的Cancer Genomics Cloud,就是构建在谷歌云之上的。
可以看到,随着云计算价格的逐步降低,基因测序技术的运算过程运行在云端将是一个越来越大的趋势。
3.4 容器技术
随着以Docker为代表的容器技术的崛起和普及,这种轻量级虚拟化技术,迅速席卷全球,为传统软件的安装部署带了了革命性的变革。
Docker容器技术使得应用程序可以在几乎任何地方以相同的方式运行。开发人员在自己笔记本上创建并测试好的容器,无需任何修改就能够在生产系统的虚拟机、物理服务器或公有云主机上运行。
即:容器使软件具备了超强的可移植能力。它可以很好的解决测序软件的各种版本运行在各种环境下的问题。比如,同一个VM上同时运行两个不同版本的GATK软件。这在传统的VM模型中是不具备的,这就给基因测序构建复杂业务场景,提供了便捷性。因为环境如果损坏,可以通过容器技术,轻松的恢复。
同时,整个测序结果,也可以很方便的重现。计算过程的可复现,这在科研领域是非常重要的。传统模型下,由于各种指定版本的软件安装,加上拥有复杂依赖关系的业务流程,要重现一个科研结果,是很困难的事情。
另外,以谷歌开源的Kubernetes为代表的调度平台也使得在大规模集群中管理Docker变得简单。被Facebook创始人马克·扎克伯格称为可与“人类基因组计划”媲美的,并获得扎克伯格基金会的资助的国际科研项目,人类细胞图谱计划(Human Cell Atlas,HCA)在2016年启动。
该项目所设想的是系统地描绘人体细胞图谱,对人体中所有(约37万亿个)细胞进行分类和测序,并通过这把钥匙,来加深对疾病诊断、监测、治疗的了解。这是一个比HGP项目在数据量和计算量都大得多的工程,目前已经有多个子项目开始使用Kubernetes作为其基础设施的底座来构建测序计算流程。
相信在未来,基于Docker容器和Kubernetes平台等新技术技术,会在基因测序领域逐步普及并发挥出更大的价值。
4 相关术语
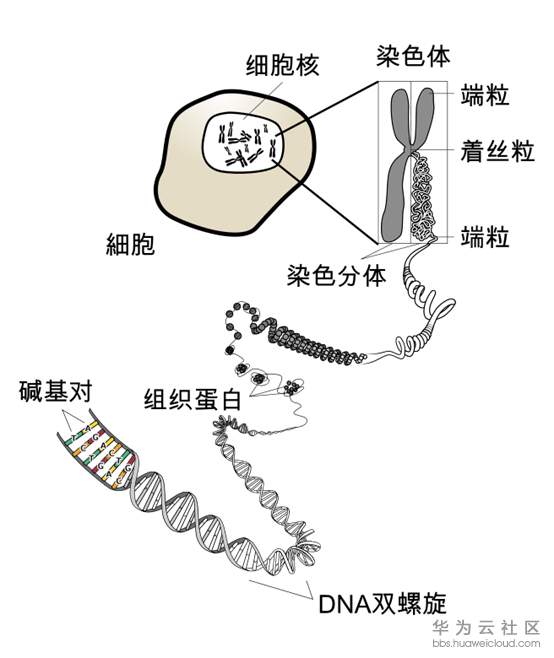
4.1 染色体
染色体是细胞核中载有遗传信息的物质,在显微镜下呈圆柱状或杆状,主要由DNA和蛋白质组成,在细胞发生有丝分裂时期容易被碱性染料着色,因此而得名。
参考:https://zh.wikipedia.org/zh-hans/%E6%9F%93%E8%89%B2%E4%BD%93
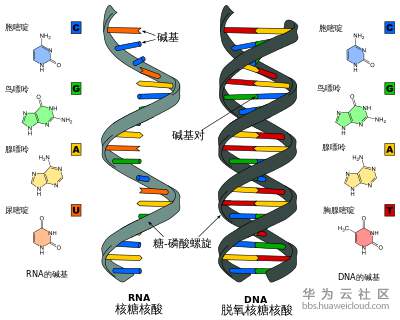
4.2 DNA
脱氧核醣核酸(英语:deoxyribonucleic acid,缩写:DNA)
人类的整个DNA是由30亿个碱基对组成。
参考:https://zh.wikipedia.org/wiki/%E8%84%B1%E6%B0%A7%E6%A0%B8%E7%B3%96%E6%A0%B8%E9%85%B8
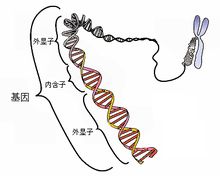
4.3 基因
基因是指控制生物性状的遗传信息,通常由DNA序列来承载。基因也可视作基本遗传单位,亦即一段具有功能性的DNA或RNA序列。也就是说并不是所有序列都叫基因,基因片段只占DNA序列总长的不到3%。
参考:https://zh.wikipedia.org/wiki/%E5%9F%BA%E5%9B%A0
最后,私货时间:
华为云618大促火热进行中,全场1折起,免费抽主机,消费满额送P30 Pro,点此抢购。
来源:华为云社区 作者:tsjsdbd