数学建模--相关性分析及Python实现
写在前面:
笔记为自行整理,内容出自课程《数学建模学习交流》,主讲人:清风
相关系数只是用来衡量两个变量线性相关程度的指标,因此,使用相关系数衡量相关性前需要确认变量间是线性相关的。
相关系数
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
%matplotlib inline
# 支持显示中文
plt.rcParams['font.family'] = ['sans-serif']
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus']=False
data = pd.read_excel('eighth_girl.xlsx')
data.head()
| 身高 | 体重 | 肺活量 | 50米跑 | 立定跳远 | 坐位体前屈 | |
|---|---|---|---|---|---|---|
| 0 | 155 | 51 | 1687 | 9.7 | 158 | 9.3 |
| 1 | 158 | 52 | 1868 | 9.3 | 162 | 9.6 |
| 2 | 160 | 59 | 1958 | 9.9 | 178 | 9.5 |
| 3 | 163 | 59 | 1756 | 9.7 | 183 | 10.1 |
| 4 | 165 | 60 | 1575 | 9.0 | 156 | 10.4 |
data.info()
RangeIndex: 591 entries, 0 to 590
Data columns (total 6 columns):
身高 591 non-null int64
体重 591 non-null int64
肺活量 591 non-null int64
50米跑 591 non-null float64
立定跳远 591 non-null int64
坐位体前屈 591 non-null float64
dtypes: float64(2), int64(4)
memory usage: 27.8 KB
统计信息
dsc = data.describe()
dsc
| 身高 | 体重 | 肺活量 | 50米跑 | 立定跳远 | 坐位体前屈 | |
|---|---|---|---|---|---|---|
| count | 591.000000 | 591.000000 | 591.000000 | 591.000000 | 591.000000 | 591.000000 |
| mean | 156.003384 | 46.783418 | 2333.233503 | 10.792014 | 166.825719 | 9.496616 |
| std | 7.389410 | 5.031473 | 350.436154 | 1.310873 | 16.813587 | 2.938186 |
| min | 135.000000 | 16.000000 | 1450.000000 | 7.800000 | 52.000000 | 0.500000 |
| 25% | 151.000000 | 44.000000 | 2109.000000 | 9.800000 | 156.000000 | 7.800000 |
| 50% | 157.000000 | 47.000000 | 2391.000000 | 10.700000 | 167.000000 | 9.600000 |
| 75% | 161.000000 | 50.000000 | 2570.000000 | 11.500000 | 178.000000 | 11.600000 |
| max | 171.000000 | 65.000000 | 3272.000000 | 15.000000 | 205.000000 | 17.500000 |
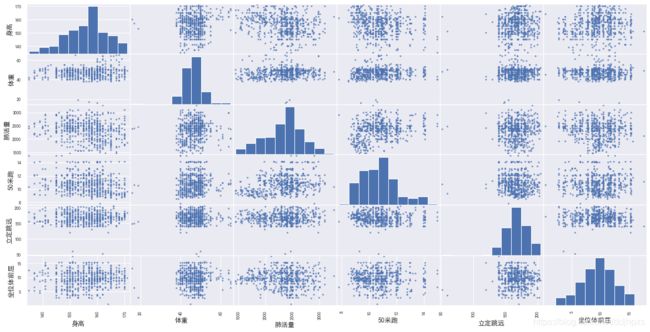
相关系数矩阵图
pd.plotting.scatter_matrix(data, figsize=(20,10), alpha=0.75)
plt.show()
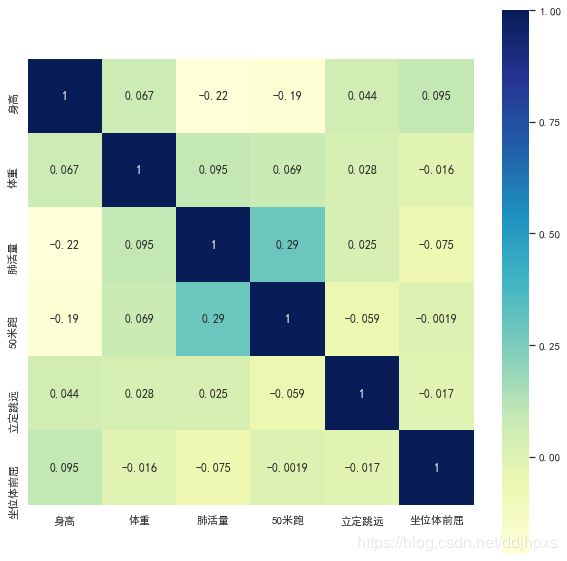
相关系数矩阵
cor = data.corr() # 默认method='pearson'
cor
| 身高 | 体重 | 肺活量 | 50米跑 | 立定跳远 | 坐位体前屈 | |
|---|---|---|---|---|---|---|
| 身高 | 1.0000 | 0.0665 | -0.2177 | -0.1920 | 0.0440 | 0.0951 |
| 体重 | 0.0665 | 1.0000 | 0.0954 | 0.0685 | 0.0279 | -0.0161 |
| 肺活量 | -0.2177 | 0.0954 | 1.0000 | 0.2898 | 0.0248 | -0.0749 |
| 50米跑 | -0.1920 | 0.0685 | 0.2898 | 1.0000 | -0.0587 | -0.0019 |
| 立定跳远 | 0.0440 | 0.0279 | 0.0248 | -0.0587 | 1.0000 | -0.0174 |
| 坐位体前屈 | 0.0951 | -0.0161 | -0.0749 | -0.0019 | -0.0174 | 1.0000 |
import seaborn as sns
sns.set(font='SimHei') # 支持中文显示
可视化矩阵
fig, ax = plt.subplots(figsize = (10,10))
# cor:相关系数矩阵
# cmap:颜色
# xticklabels:显示x轴标签
# yticklabels:显示y轴标签
# annot:方块中显示数据
# square:方块为正方形
sns.heatmap(cor, cmap='YlGnBu', xticklabels=True, yticklabels=True,
annot=True, square=True)
相关系数的假设检验
第一步:提出原假设和 H 0 H_0 H0和备择假设 H 1 H_1 H1;假设已得到一个皮尔逊相关系数 r r r,要想检验它是否显著的异于0,可以设定: H 0 : r = 0 , H 1 : r ≠ 0 H_0:r=0,H_1:r{\ne}0 H0:r=0,H1:r=0
第二步:在原假设成立的条件下,利用要检验的量构造出一个符合某一分布的统计量。对于皮尔逊相关系数 r r r而言,可以构造统计量:
t = r n − 2 1 − r 2 t=r\sqrt{\frac{n-2}{1-r^2}} t=r1−r2n−2
可以证明, t t t是服从自由度为 n − 2 n-2 n−2的 t t t分布。
第三步:将要检验的值带入这个统计量中,可以得到一个特性的值。假设计算出的相关系数为0.5,样本为30,则可以得到 t ∗ = 0.5 30 − 2 1 − 0. 5 2 = 3.05505 t^*=0.5\sqrt{\frac{30-2}{1-0.5^2}}=3.05505 t∗=0.51−0.5230−2=3.05505
第四步:根据 t t t分布的定义可以得到置信水平为0.95的 t t t分布的置信区间。做比较即可。
from scipy import stats
np.set_printoptions(suppress=True) # 不使用用科学计数法
pd.set_option('display.float_format',lambda x : '%.4f' % x) # 保留小数点后4位有效数字
# 0.975分位数
tp = stats.t.isf(1-0.975, 28)
x = np.linspace(-5,5,100)
y = stats.t.pdf(x, 28)
plt.plot(x,y)
plt.vlines(-tp, 0, stats.t.pdf(-tp, 28), colors='orange')
plt.vlines(tp, 0, stats.t.pdf(tp, 28), colors='orange')
plt.fill_between(x, 0, y, where=abs(x)>tp, interpolate=True, color='r')
更简便的方法:求p值
# 自定义求解p值矩阵的函数
def my_pvalue_pearson(x):
col = x.shape[1]
col_name = x.columns.values
p_val = []
for i in range(col):
for j in range(col):
p_val.append(stats.pearsonr(x[col_name[i]], x[col_name[j]])[1])
p_val = pd.DataFrame(np.array(p_val).reshape(col, col), columns=col_name, index=col_name)
p_val.to_csv('p_val_pearson.csv') # 此处实则为多此一举,目的是借助带有excel格式的数据使得输出更美观
p_val = pd.read_csv('p_val_pearson.csv', index_col=0)
return p_val
my_pvalue_pearson(data)
| 身高 | 体重 | 肺活量 | 50米跑 | 立定跳远 | 坐位体前屈 | |
|---|---|---|---|---|---|---|
| 身高 | 0.0000 | 0.1061 | 0.0000 | 0.0000 | 0.2859 | 0.0208 |
| 体重 | 0.1061 | 0.0000 | 0.0204 | 0.0960 | 0.4978 | 0.6963 |
| 肺活量 | 0.0000 | 0.0204 | 0.0000 | 0.0000 | 0.5469 | 0.0687 |
| 50米跑 | 0.0000 | 0.0960 | 0.0000 | 0.0000 | 0.1542 | 0.9637 |
| 立定跳远 | 0.2859 | 0.4978 | 0.5469 | 0.1542 | 0.0000 | 0.6728 |
| 坐位体前屈 | 0.0208 | 0.6963 | 0.0687 | 0.9637 | 0.6728 | 0.0000 |
将p值与0.05做比较即可
正态分布检验
Jarque-Bera检验(n>30)
x = stats.norm.rvs(2, 3, 100)
skewness = stats.skew(x) # 偏度
kurtosis = stats.kurtosis(x) # 峰度
jbtext = stats.jarque_bera(x)
print('偏度为:',skewness)
print('峰度为:',kurtosis)
print('J-B值:',jbtext[0])
print('p-value:',jbtext[1])
偏度为: 0.05657245566213502
峰度为: -0.135509867406316
J-B值: 0.1298528963460595
p-value: 0.9371363889360985
def my_jbtext(x):
col_name = x.columns.values
col_cnt = x.shape[1]
h_mat = np.zeros(col_cnt)
p_mat = np.zeros(col_cnt)
for i in range(col_cnt):
p_val = stats.jarque_bera(data[col_name[i]])[1]
p_mat[i] = p_val
if p_val >= 0.05:
h_mat[i] = 0 # 通过原假设
else:
h_mat[i] = 1 # 拒绝原假设
print(h_mat)
print(p_mat) # 各列的p值
my_jbtext(data)
[1. 1. 1. 1. 1. 1.]
[0.00602083 0. 0.00853 0. 0. 0.03949912]
1代表拒绝原假设,0代表接受
Shapiro-wilk检验(3<=n<=50)
stats.shapiro(data['身高']) # 单个变量
(0.9839125275611877, 4.196843292447738e-06)
def my_shaptext(x):
col_name = x.columns.values
col_cnt = x.shape[1]
h_mat = np.zeros(col_cnt)
p_mat = np.zeros(col_cnt)
for i in range(col_cnt):
p_val = stats.shapiro(data[col_name[i]])[1]
p_mat[i] = p_val
if p_val >= 0.05:
h_mat[i] = 0 # 通过原假设
else:
h_mat[i] = 1 # 拒绝原假设
print(h_mat)
print(p_mat) # 各列的p值
my_shaptext(data)
[1. 1. 1. 1. 1. 1.]
[0.0000042 0. 0.00000207 0. 0. 0.00005767]
Q-Q图(大样本时使用)
stats.probplot(data['身高'], dist="norm", plot=plt)
plt.show()
斯皮尔曼相关系数
定义: X X X和 Y Y Y为两组数据,其斯皮尔曼相关系数:
r s = 1 − 6 ∑ i = 1 n d i 2 n ( n 2 − 1 ) r_s=1-\frac{6\sum_{i=1}^n{d_{i}^{2}}}{n\left( n^2-1 \right)} rs=1−n(n2−1)6∑i=1ndi2
其中, d i d_i di为 X i X_i Xi和 Y i Y_i Yi之间的等级差。(一个数的等级是指,将其所在的一列数按照从小到大的顺序排列后,这个数作在的位置;若有相同的数值,则他们的等级为所在位置的算数平均)
可以证明: r s r_s rs位于 − 1 -1 −1和 1 1 1之间。
data.corr(method='spearman')
| 身高 | 体重 | 肺活量 | 50米跑 | 立定跳远 | 坐位体前屈 | |
|---|---|---|---|---|---|---|
| 身高 | 1.0000 | 0.0301 | -0.2430 | -0.1990 | 0.0624 | 0.1099 |
| 体重 | 0.0301 | 1.0000 | 0.1305 | 0.0898 | 0.0216 | -0.0488 |
| 肺活量 | -0.2430 | 0.1305 | 1.0000 | 0.2626 | 0.0219 | -0.0801 |
| 50米跑 | -0.1990 | 0.0898 | 0.2626 | 1.0000 | -0.0910 | -0.0029 |
| 立定跳远 | 0.0624 | 0.0216 | 0.0219 | -0.0910 | 1.0000 | -0.0399 |
| 坐位体前屈 | 0.1099 | -0.0488 | -0.0801 | -0.0029 | -0.0399 | 1.0000 |
斯皮尔曼相关系数的假设检验
# 自定义求解p值矩阵的函数
def my_pvalue_spearman(x):
col = x.shape[1]
col_name = x.columns.values
p_val = []
for i in range(col):
for j in range(col):
p_val.append(stats.spearmanr(x[col_name[i]], x[col_name[j]])[1])
p_val = pd.DataFrame(np.array(p_val).reshape(col, col), columns=col_name, index=col_name)
p_val.to_csv('p_val_spearman.csv') # 此处实则为多此一举,目的是借助带有excel格式的数据使得输出更美观
p_val = pd.read_csv('p_val_spearman.csv', index_col=0)
return p_val
my_pvalue_spearman(data)
| 身高 | 体重 | 肺活量 | 50米跑 | 立定跳远 | 坐位体前屈 | |
|---|---|---|---|---|---|---|
| 身高 | 0.0000 | 0.4647 | 0.0000 | 0.0000 | 0.1295 | 0.0075 |
| 体重 | 0.4647 | 0.0000 | 0.0015 | 0.0290 | 0.5996 | 0.2362 |
| 肺活量 | 0.0000 | 0.0015 | 0.0000 | 0.0000 | 0.5944 | 0.0517 |
| 50米跑 | 0.0000 | 0.0290 | 0.0000 | 0.0000 | 0.0270 | 0.9436 |
| 立定跳远 | 0.1295 | 0.5996 | 0.5944 | 0.0270 | 0.0000 | 0.3330 |
| 坐位体前屈 | 0.0075 | 0.2362 | 0.0517 | 0.9436 | 0.3330 | 0.0000 |
斯皮尔曼相关系数和皮尔逊相关系数的选择:
1.连续数据,正态分布,线性关系,用pearson相关系数是最恰当,当然用spearman相关系数也可以, 就是效率没有pearson相关系数高。
2.上述任一条件不满足,就用spearman相关系数,不能用pearson相关系数。
3.两个定序数据之间也用spearman相关系数,不能用pearson相关系数。
定序数据是指仅仅反映观测对象等级、顺序关系的数据,是由定序尺度计量形成的,表现为类别,可以进行排序,属于品质数据。
例如:优、良、差;
我们可以用1表示差、 2表示良、 3表示优,但请注意,用2除以1得出的2并不
代表任何含义。定序数据最重要的意义代表了一组数据中的某种逻辑顺序。
注:斯皮尔曼相关系数的适用条件比皮尔逊相关系数要广,只要数据满足单调关系
(例如线性函数、指数函数、对数函数等)就能够使用。