ML:非监督学习之 聚类_从KMeans到GMM高斯混合聚类
本文节选于《Python Data Science Handbook》by Jake VanderPlas 2016-11-17 First Edition
In Depth: Gaussian Mixture Model
k-means 聚类模型简单、易于理解,但是也因此无法处理比较复杂的聚类问题。尤其是它的非概率论本质,和它用简单的distance-from-cluster-center/中心点距离来进行聚类方法,导致在许多实际情况下模型效果不佳。本文要讨论的GMM/Gaussian Mixture Model可以看做是k-means的拓展,但是克服了KMeans缺点的有力工具。
Motivating GMM: Weaknesses of k-Means
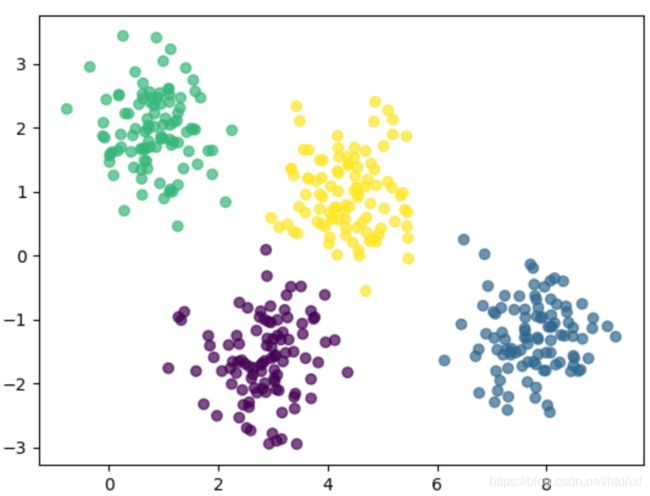
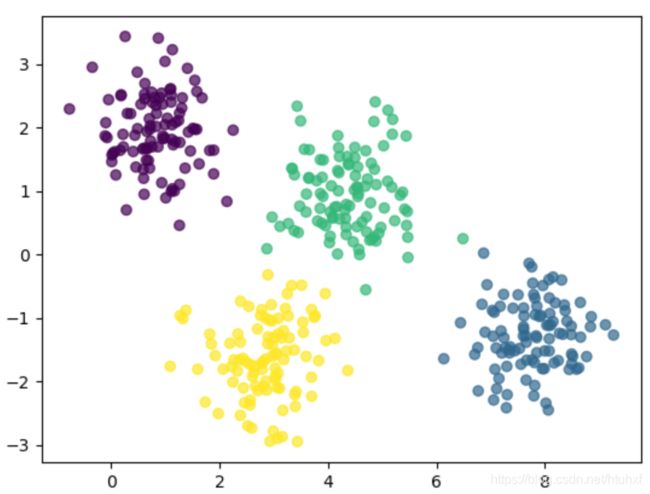
我们首先看些k-means的不足,然后想想增强聚类模型的方向。在(书的)前边我们看到,KMeans对于well-seperated的数据分类效果很棒。如下的例子,KMeans的分类结果和肉眼观察的非常接近:
# 首先生成一些数据
from sklearn.datasets import make_blobs
X, y_true = make_blobs(n_samples=400, centers=4, cluster_std=0.6, random_state=0)
X = X[:, ::-1] # 翻转坐标轴使做出来的图更直观(不翻转也差不多)
from sklearn.cluster import KMeans
kmeans = KMeans(4, random_state=0)
labels = kmeans.fit_predict(X) # .fit_predict(X) 等价于 .fit(X).predict(X)
plt.scatter(X[:,0], X[:, 1], c=labels, alpha=0.7)
plt.show()
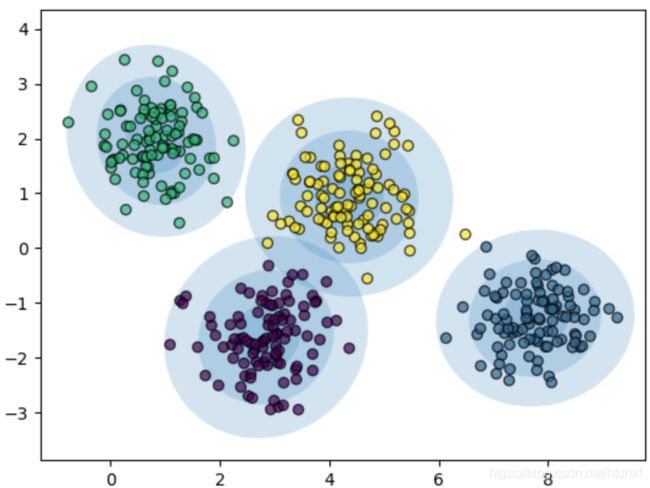
 如上图直观上,一些位置的数据点比其他的分类更确切一些,但是,中间的2个clusters之间的点就有些模糊,似乎归到无论哪边都说得通。同时,KMean又给不出cluster里每个点分到哪一类的概率数据,因此我们就得弄个能满足我们要求的分析工具(或许通过bootstrap来估计出来这些概率)。
如上图直观上,一些位置的数据点比其他的分类更确切一些,但是,中间的2个clusters之间的点就有些模糊,似乎归到无论哪边都说得通。同时,KMean又给不出cluster里每个点分到哪一类的概率数据,因此我们就得弄个能满足我们要求的分析工具(或许通过bootstrap来估计出来这些概率)。
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
from scipy.spatial.distance import cdist
import matplotlib.pyplot as plt
def plot_kmeans(kmeans, X, n_cluster=4, rseed=0, ax=None):
labels = kmeans.fit_predict(X)
# 先画个散点图
ax = ax or plt.gca()
ax.axis('equal')
ax.scatter(X[:, 0], X[:, 1],
c=labels, alpha=0.7, ec='black', lw=1,
cmap='viridis', s=40, zorder=2) # zorder,计算机顺序用语,值越小越先画
# 在画个圈圈
centers = kmeans.cluster_centers_
radii = [cdist(X[labels == i], [center]).max()
for i, center in enumerate(centers)]
for c, r in zip(centers, radii):
ax.add_patch(plt.Circle(c, r,
fc='#CCCCCC', ec='black',
lw=3, alpha=0.5, zorder=1))
X, y_true = make_blobs(n_samples=400, centers=4, cluster_std=0.6, random_state=0)
X = X[:, ::-1]
kmeans = KMeans(n_clusters=4, random_state=0)
plot_kmeans(kmeans, X)
plt.show()
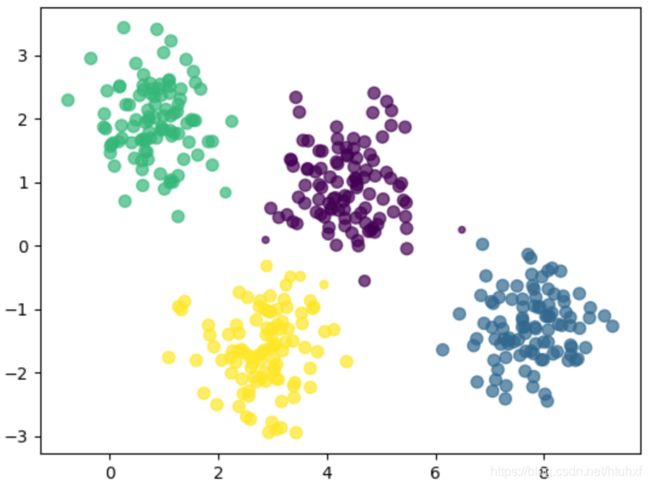
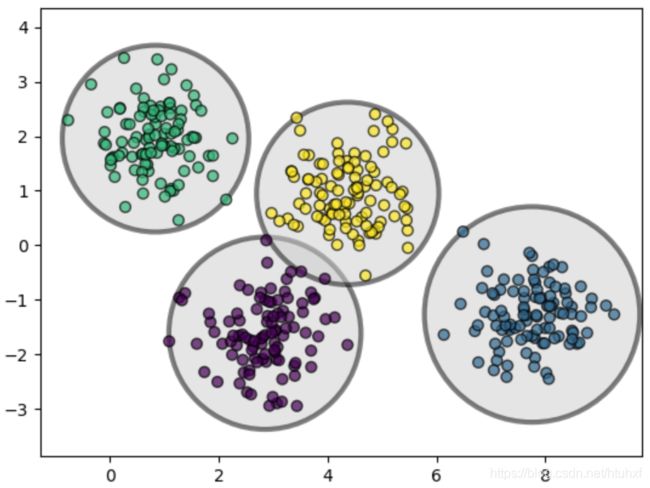 上图很明显展示了KMeans是按圆圈子进行聚类的,也最适合离散分布呈现圆形的数据点。但是其他的形状就不行了,如矩形、椭圆啥的就。如下边这个例子:
上图很明显展示了KMeans是按圆圈子进行聚类的,也最适合离散分布呈现圆形的数据点。但是其他的形状就不行了,如矩形、椭圆啥的就。如下边这个例子:
import numpy as np
rng = np.random.RandomState(13)
X_stretched = np.dot(X, rng.randn(2, 2))
kmeans = KMeans(n_clusters=4, random_state=0)
plot_kmeans(kmeans, X_stretched)
plt.show()
 上图直观显示了非圆形分布数据点并没有被聚好类;也形象地再次印证了在聚类上KMeans就会画圈圈这一招;
上图直观显示了非圆形分布数据点并没有被聚好类;也形象地再次印证了在聚类上KMeans就会画圈圈这一招;
所以KMeans的两个缺点:一是适用的范围窄,二是无法给出每个数据点被聚到该某一类的概率;
我们自然会想到:如果KMeans的圆圈边界是椭圆而不局限于圆,并且是通过对比数据点到所有中心点的距离作为聚类的依据、而不是仅仅关注最近的中心是哪个,该有多好!其实满足这俩条件的模型是有的,它名字叫Guassian Mixture Models/高斯混合模型。
Generalizing E-M: Gaussian Mixture Models/GM
Gaussian Mixture模型用来对存在多维高斯概率分布特征数据进行建模。KMeans可以算是GM的一种简单情形:
from sklearn.mixture import GaussianMixture
import matplotlib.pyplot as plt
from sklearn.datesets import make_plobs
X, y_ture = make_plobs(n_samples=400, centers=4,
cluster_std=0.6, random_state=0)
X = X[:, ::-1]
gm = Gaussian.Mixture(n_components=4)
labels = gm.fit_predict(X)
plt.scatter(X[:, 0], X[:, 1], c=labels, alpha=0.7)
plt.show()
 除此之外,GM模型内含计算每个点聚到该类中心点的概率功能,
除此之外,GM模型内含计算每个点聚到该类中心点的概率功能,predict_proba,应用如下:
probs = gm.predict_proba(X)
print(probs[:5])
# 结果如下
[[1.75162717e-22 4.69238090e-01 2.76240973e-07 5.30761633e-01]
[4.71110558e-15 1.97106146e-17 9.99999999e-01 9.22826700e-10]
[3.07981606e-17 2.34875746e-14 9.99999998e-01 2.09565089e-09]
[5.00285665e-10 5.27592131e-11 9.15972926e-05 9.99908402e-01]
[5.28028479e-14 3.86146363e-18 9.99999998e-01 1.69687673e-09]]
并且我们能够可视化每个点概率的大小
size = 50 * probs.max()
plt.scatter(X[:0], X[:1], s=size, c=labels, alpha=0.7)
plt.show()
 GM的算法原理和KMeans是很相似的:使用expectation-maximization/EM/期望最大化为目标进行如下步骤:
GM的算法原理和KMeans是很相似的:使用expectation-maximization/EM/期望最大化为目标进行如下步骤:
1. Choose starting guesses for the location and shape
2. 满足条件之前进行如下循环:
a. 期望/E step:对于每一个数据点,find weights encoding the probability of membership in each cluster
b. 最大化/M step:对于每一个类,update its location, normalization, and shape based on all data points, making use of the weights
和KMeans相比它的最大特点是不同如下:
from matplot.patches import Ellipse
import matplot.pyplot as plt
from sklearn.mixture import GaussianMixture
from sklearn.datesets import make_blobs
import numpy as np
def draw_ellipse(position, covariance, ax=None, **kwargs):
ax = ax or plt.gca()
if covariance.shape == (2, 2):
U, s, Vt = np.linalg.svd(covariance)
angle = np.degrees(np.arctan2(U[1, 0], U[0, 0]))
width, height = 2 * np.sqrt(s)
else:
angle=0
width, height = 2 * np.sqrt(covariance)
for nsig in range(1, 4):
ax.add_patch(Ellipse(position, nsig * width, nsig * height,
angle, ##kwargs))
def plot_gm(gm, X, label=True):
labels = gm.fit_predict(X)
if label:
plt.scatter(X[:, 0], X[:, 1], c=labels, alpha=0.7, zorder=2,
ec='black')
else:
plt.scatter(X[:, 0], X[:, 1], alpha=0.7, zorder=2,
ec='black')
plt.axis('equal')
w_factor = 0.2 / gm.weights_.max()
for pos, covar, w in zip(gm.means_, gm.covariances_, gm.weights_):
draw_ellipse(pos, covar, alpha=w*w_factor)
X, y_true = make_blobs(n_samples=400, centers=4, cluster_std=0.6, random_state=0)
X = X[:, ::-1]
gm = GaussianMixture(n_components=4, random_states=42)
plot_gm(gm, X)
plt.show()
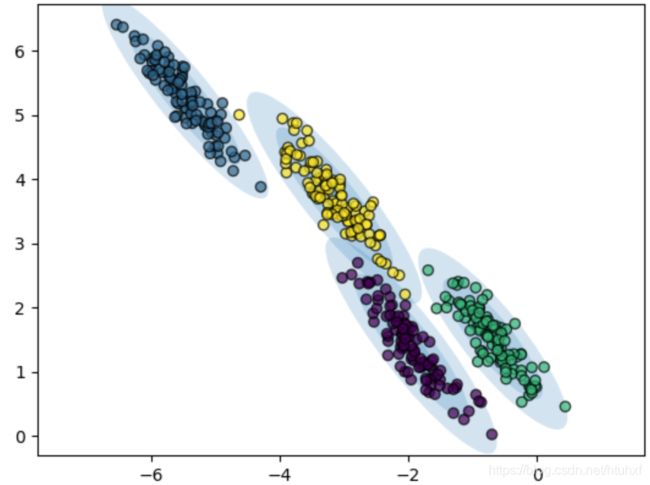
类似的,我们可以用GM模型再来模拟一下上边长条分布的数据,会发现效果好多了:
rng = np.random.RandomState(13)
X_stretched = np.dot(X, rng.randn(2, 2))
gm = GaussianMixture(n_components=4, random_state=0)
plot_gm(gm, X_stretched)
plt.show()
Choosing the covariance type
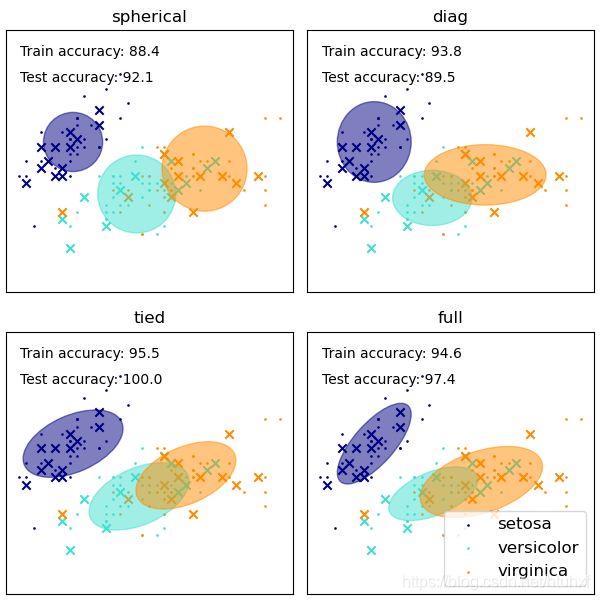
如果看sklearn.mixture.Gaussian()会发现有个选项 c o v a r i a n c e _ t y p e covariance\_type covariance_type的4个参数 { ‘ f u l l ’ ( d e f a u l t ) , ‘ t i e d ’ , ‘ d i a g ’ , ‘ s p h e r i c a l ’ } \{‘full’ (default), ‘tied’, ‘diag’, ‘spherical’\} {‘full’(default),‘tied’,‘diag’,‘spherical’}:它们代表着不同的自由度,即决定画的椭圆朝向。
spherical:每个类的圈圈仅仅是方差不一样(表现出来就是圆的半径不同),图像和KMeans有点像(见下图),最简单、最快;
diag:每个类的圈圈的协方差矩阵都是自由独立的的对角阵,意味着圈圈的半径都平行于坐标轴但长度是自由的(见下图);
tied:每个类的圈圈的协方差矩阵都一样,意味着圈圈大小、半径都一样,只是不要求半径方向和坐标轴平行,例如斜着的椭圆(见下图);
full:每个类的圈圈的协方差矩阵都是自由独立的,意味着圈圈可以半径长度、和坐标轴的方向都是自由独立的(见下图)
convariance_type对应着线性变换。简单来说对角阵只是在原坐标轴方向上进行的拉伸缩短、非对角阵可以看做是拉伸缩短基础上对坐标轴也进行了旋转;
想进一步了解的可以看3Blue1Brown的非常棒非常易懂的视频展示《线性代数的本质》链接在此。
 #### GM as Density Estimation
#### GM as Density Estimation
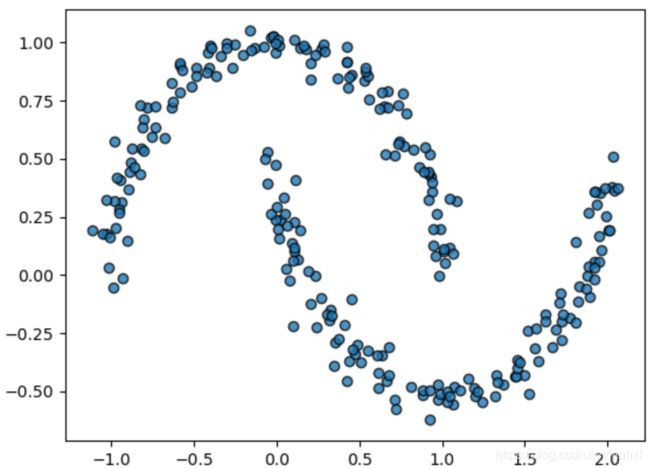
虽然GM经常被当做一种聚类的算法,但实质上它是基于density estimation/密度估计的算法。什么意思呢?就是模型对数据点的拟合技术上说不是聚类模型,而是一般意义上用于描述数据点分布的概率模型。举个栗子:
from sklearn.datasets import make_moons
import matplotlib.pyplot as plt
Xmoon, ymoon = make_moons(250, noise=0.05, random_state=0 )
plt.scatter(Xmoon[:, 0], Xmoon[:, 1], alpha=0.8, ec='black')
plt.show()
 如果我们直接设定
如果我们直接设定n_compenents=2,结果并不理想:
from sklearn.datasets import make_moons
Xmoon, ymoon = make_moons(250, noise=0.05, random_state=0)
gm = GaussianMixture(n_components=2, random_state=0)
plot_gm(gm, Xmoon)
plt.show()
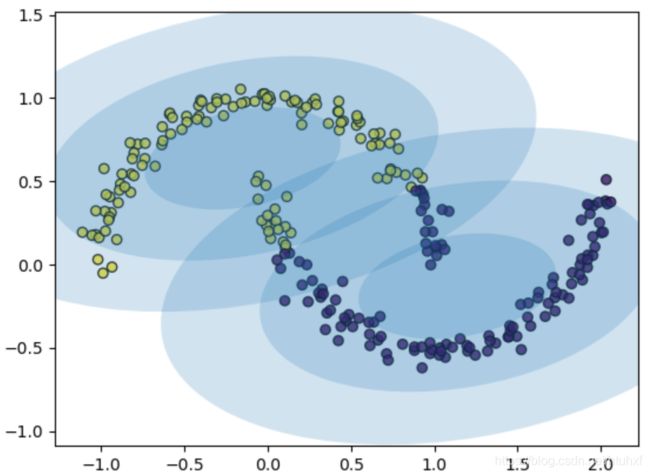 但是,如果忽略标签,并且用更大的
但是,如果忽略标签,并且用更大的n_components,会发现:
gm = GaussianMixture(n_components=18, random_state=0)
plot_gm(gm, Xmoon, label=False)
plt.show()
 上图并不是为了尽心聚类,而是拟合数据点的分布。它此时是一个分布模型,就是说我们可以用它产生类似原数据分布的新数据点,例如:
上图并不是为了尽心聚类,而是拟合数据点的分布。它此时是一个分布模型,就是说我们可以用它产生类似原数据分布的新数据点,例如:
gm = GaussianMixture(n_components=18, random_state=0)
gm.fit(Xmoon)
Xnew, ynew = gm.sample(500)
plt.scatter(Xnew[:, 0], Xnew[:, 1], ec='black', alpha=0.8)
plt.show()
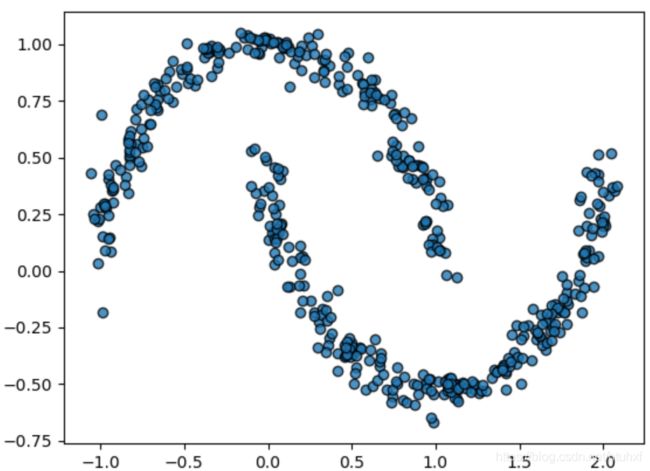 对于任意的多维分布数据点集合,GM是一个方便灵活的的拟合模型。
对于任意的多维分布数据点集合,GM是一个方便灵活的的拟合模型。
How many components?
事实上GM作为一个generative model,提供了一些个确定最佳n_components的方法。Generative model本质上是一个拟合数据的概率分布模型, 所以我们可以轻易地量化数据点在模型里的 l i k l i h o o d liklihood liklihood,并通过cross-validation避免过拟合。另一种修正过拟合的途径是通过模型评判标准:Akaike information criterion(AIC)或者Bayesian information criterion(BIC)。Scikit-Learn的GM estimator内置AIC和BIC标准,可以非常方便的使用:
n_components = np.arange(1, 21)
models = [GaussianMixture(n, random_state=0).fit(Xmoon) for n in n_components]
plt.plot(n_components, [m.aic(Xmoon) for m in models], label='AIC')
plt.plot(n_components, [m.bic(Xmoon) for m in models], label='BIC')
plt.xlabel('n_components')
plt.legend()
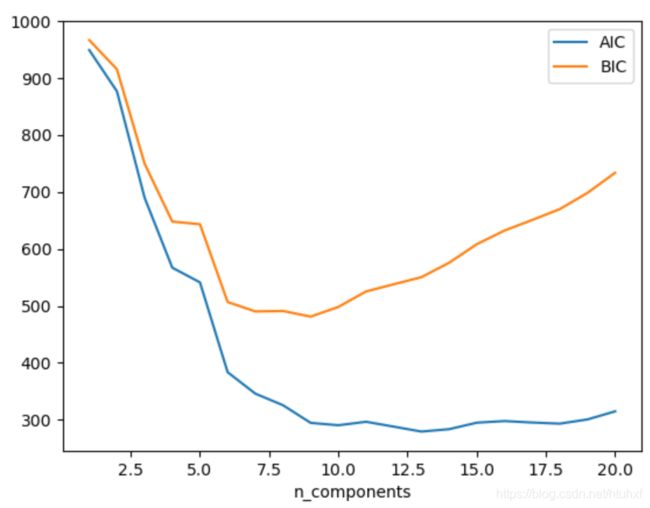 AIC和BIC本质上是“熵”,即值越小越好,一般形式如下。
AIC和BIC本质上是“熵”,即值越小越好,一般形式如下。
A I C = 2 k − 2 ln ( L ) AIC=2k - 2\ln(L) AIC=2k−2ln(L)
B I C = k ln ( n ) − 2 ln ( L ) BIC=k\ln(n)-2\ln(L) BIC=kln(n)−2ln(L)
其中 k k k为模型参数个数, n n n为样本数量, L L L为极大似然函数估计。
AIC和BIC相同之处都是 L L L越大越好,模型参数 k k k越少,但是惩罚项的侧重不同,即当 n > e 2 n>e^2 n>e2即样本不小于7的时候,BIC的惩罚度更大,因为当1)其他条不变情况下,任何模型的拟合度都随着 n n n的增大而增大,所以倾向于选择使用样本数较少的模型;2)只改变 k k k同时 k k k不小于7的情况下,AIC的结果增速较小。
需要再次强调的是,AIC和BIC选择的n_components仅针对GM作为density estimator,而不是作为聚类算法时的优选结果。不少人都更倾向于把GM作为一个density estimator,只有在简单数据集有保证的情况下用于聚类。