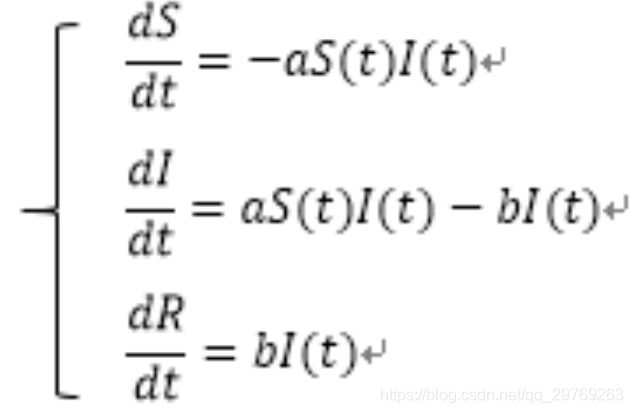- 机器学习与深度学习间关系与区别
ℒℴѵℯ心·动ꦿ໊ོ꫞
人工智能学习深度学习python
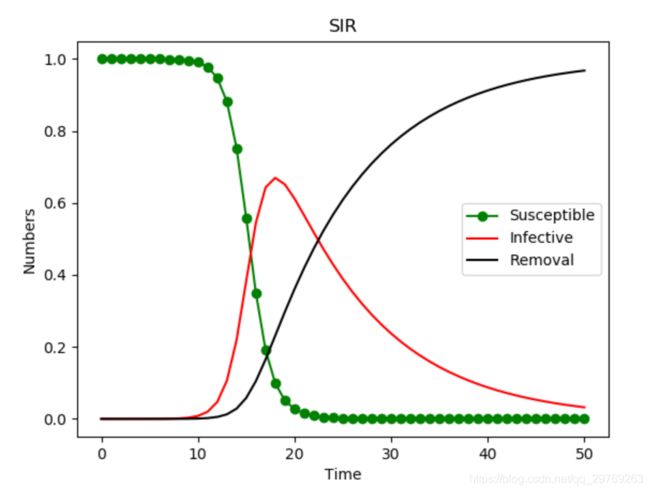
一、机器学习概述定义机器学习(MachineLearning,ML)是一种通过数据驱动的方法,利用统计学和计算算法来训练模型,使计算机能够从数据中学习并自动进行预测或决策。机器学习通过分析大量数据样本,识别其中的模式和规律,从而对新的数据进行判断。其核心在于通过训练过程,让模型不断优化和提升其预测准确性。主要类型1.监督学习(SupervisedLearning)监督学习是指在训练数据集中包含输入
- OC语言多界面传值五大方式
Magnetic_h
iosui学习objective-c开发语言
前言在完成暑假仿写项目时,遇到了许多需要用到多界面传值的地方,这篇博客来总结一下比较常用的五种多界面传值的方式。属性传值属性传值一般用前一个界面向后一个界面传值,简单地说就是通过访问后一个视图控制器的属性来为它赋值,通过这个属性来做到从前一个界面向后一个界面传值。首先在后一个界面中定义属性@interfaceBViewController:UIViewController@propertyNSSt
- 理解Gunicorn:Python WSGI服务器的基石
范范0825
ipythonlinux运维
理解Gunicorn:PythonWSGI服务器的基石介绍Gunicorn,全称GreenUnicorn,是一个为PythonWSGI(WebServerGatewayInterface)应用设计的高效、轻量级HTTP服务器。作为PythonWeb应用部署的常用工具,Gunicorn以其高性能和易用性著称。本文将介绍Gunicorn的基本概念、安装和配置,帮助初学者快速上手。1.什么是Gunico
- 30天风格练习-DAY2
黄希夷
Day2(重义)在一个周日/一周的最后一天,我来到位于市中心/市区繁华地带的一家购物中心/商场,中心内人很多/熙熙攘攘。我注意到/看见一个独行/孤身一人的年轻女孩/,留着一头引人注目/长过腰际的头发,上身穿一件暗红色/比正红色更深的衣服/穿在身体上的东西。走下扶梯的时候,她摔倒了/跌向地面,在她正要站起来/让身体离开地面的时候,过长/超过一般人长度的头发被支撑身体/躯干的手掌压/按在下面,她赶紧用
- 消息中间件有哪些常见类型
xmh-sxh-1314
java
消息中间件根据其设计理念和用途,可以大致分为以下几种常见类型:点对点消息队列(Point-to-PointMessagingQueues):在这种模型中,消息被发送到特定的队列中,消费者从队列中取出并处理消息。队列中的消息只能被一个消费者消费,消费后即被删除。常见的实现包括IBM的MQSeries、RabbitMQ的部分使用场景等。适用于任务分发、负载均衡等场景。发布/订阅消息模型(Pub/Sub
- Python数据分析与可视化实战指南
William数据分析
pythonpython数据
在数据驱动的时代,Python因其简洁的语法、强大的库生态系统以及活跃的社区,成为了数据分析与可视化的首选语言。本文将通过一个详细的案例,带领大家学习如何使用Python进行数据分析,并通过可视化来直观呈现分析结果。一、环境准备1.1安装必要库在开始数据分析和可视化之前,我们需要安装一些常用的库。主要包括pandas、numpy、matplotlib和seaborn等。这些库分别用于数据处理、数学
- 每日一题——第八十四题
互联网打工人no1
C语言程序设计每日一练c语言
题目:编写函数1、输入10个职工的姓名和职工号2、按照职工由大到小顺序排列,姓名顺序也随之调整3、要求输入一个职工号,用折半查找法找出该职工的姓名#define_CRT_SECURE_NO_WARNINGS#include#include#defineMAX_EMPLOYEES10typedefstruct{intid;charname[50];}Empolyee;voidinputEmploye
- WPF中的ComboBox控件几种数据绑定的方式
互联网打工人no1
wpfc#
一、用字典给ItemsSource赋值(此绑定用的地方很多,建议熟练掌握)在XMAL中:在CS文件中privatevoidBindData(){DictionarydicItem=newDictionary();dicItem.add(1,"北京");dicItem.add(2,"上海");dicItem.add(3,"广州");cmb_list.ItemsSource=dicItem;cmb_l
- git常用命令笔记
咩酱-小羊
git笔记
###用习惯了idea总是不记得git的一些常见命令,需要用到的时候总是担心旁边站了人~~~记个笔记@_@,告诉自己看笔记不丢人初始化初始化一个新的Git仓库gitinit配置配置用户信息gitconfig--globaluser.name"YourName"gitconfig--globaluser.email"
[email protected]"基本操作克隆远程仓库gitclone查看
- python os.environ
江湖偌大
python深度学习
os.environ['TF_CPP_MIN_LOG_LEVEL']='0'#默认值,输出所有信息os.environ['TF_CPP_MIN_LOG_LEVEL']='1'#屏蔽通知信息(INFO)os.environ['TF_CPP_MIN_LOG_LEVEL']='2'#屏蔽通知信息和警告信息(INFO\WARNING)os.environ['TF_CPP_MIN_LOG_LEVEL']='
- Python中os.environ基本介绍及使用方法
鹤冲天Pro
#Pythonpython服务器开发语言
文章目录python中os.environos.environ简介os.environ进行环境变量的增删改查python中os.environ的使用详解1.简介2.key字段详解2.1常见key字段3.os.environ.get()用法4.环境变量的增删改查和判断是否存在4.1新增环境变量4.2更新环境变量4.3获取环境变量4.4删除环境变量4.5判断环境变量是否存在python中os.envi
- Pyecharts数据可视化大屏:打造沉浸式数据分析体验
我的运维人生
信息可视化数据分析数据挖掘运维开发技术共享
Pyecharts数据可视化大屏:打造沉浸式数据分析体验在当今这个数据驱动的时代,如何将海量数据以直观、生动的方式展现出来,成为了数据分析师和企业决策者关注的焦点。Pyecharts,作为一款基于Python的开源数据可视化库,凭借其丰富的图表类型、灵活的配置选项以及高度的定制化能力,成为了构建数据可视化大屏的理想选择。本文将深入探讨如何利用Pyecharts打造数据可视化大屏,并通过实际代码案例
- 直抒《紫罗兰永恒花园外传》
雷姆的黑色童话
没看过《紫罗兰永恒花园》的我莫名的看完了《紫罗兰永恒花园外传》,又莫名的被故事中的姐妹之情狠狠地感动了的一把。感动何在:困苦中相依为命的姐妹二人被迫分离,用一个人的自由换取另一个人的幸福。之后,虽相隔不知几许依旧心心念念彼此牵挂。这种深深的姐妹情谊就是令我为之动容的所在。贝拉和泰勒分别影片开始,海天之间一个孩童凭栏眺望,手中拿着折旧的信纸。镜头一转,挑灯伏案的薇尔莉特正在打字机前奋笔疾书。这些片段
- Python教程:一文了解使用Python处理XPath
旦莫
Python进阶python开发语言
目录1.环境准备1.1安装lxml1.2验证安装2.XPath基础2.1什么是XPath?2.2XPath语法2.3示例XML文档3.使用lxml解析XML3.1解析XML文档3.2查看解析结果4.XPath查询4.1基本路径查询4.2使用属性查询4.3查询多个节点5.XPath的高级用法5.1使用逻辑运算符5.2使用函数6.实战案例6.1从网页抓取数据6.1.1安装Requests库6.1.2代
- python os.environ_python os.environ 读取和设置环境变量
weixin_39605414
pythonos.environ
>>>importos>>>os.environ.keys()['LC_NUMERIC','GOPATH','GOROOT','GOBIN','LESSOPEN','SSH_CLIENT','LOGNAME','USER','HOME','LC_PAPER','PATH','DISPLAY','LANG','TERM','SHELL','J2REDIR','LC_MONETARY','QT_QPA
- linux中sdl的使用教程,sdl使用入门
Melissa Corvinus
linux中sdl的使用教程
本文通过一个简单示例讲解SDL的基本使用流程。示例中展示一个窗口,窗口里面有个随机颜色快随机移动。当我们鼠标点击关闭按钮时间窗口关闭。基本步骤如下:1.初始化SDL并创建一个窗口。SDL_Init()初始化SDL_CreateWindow()创建窗口2.纹理渲染存储RGB和存储纹理的区别:比如一个从左到右由红色渐变到蓝色的矩形,用存储RGB的话就需要把矩形中每个点的具体颜色值存储下来;而纹理只是一
- LLM 词汇表
落难Coder
LLMsNLP大语言模型大模型llama人工智能
Contextwindow“上下文窗口”是指语言模型在生成新文本时能够回溯和参考的文本量。这不同于语言模型训练时所使用的大量数据集,而是代表了模型的“工作记忆”。较大的上下文窗口可以让模型理解和响应更复杂和更长的提示,而较小的上下文窗口可能会限制模型处理较长提示或在长时间对话中保持连贯性的能力。Fine-tuning微调是使用额外的数据进一步训练预训练语言模型的过程。这使得模型开始表示和模仿微调数
- 读《人世间》有感
一0一
这个寒假,就如同朋友圈中的一段话:一闭眼,一睁眼假期还有5天,在一闭眼一睁眼假期还有12天;再一闭眼一睁眼假期还有20天;不敢睡,不敢睡啊……受疫情影响,这个假期变得漫长又煎熬,我也无时无刻不关注着疫情的变化。当然这样的一个假期,我还真得要感谢周翔,因为他有个爱看书的习惯,所以家里有不少他看过的书,可以让我随意挑选,因此也让我的假期不至于那么无所事事。这次我选了一本梁晓声的《人世间》,作为一名语文
- 2020-04-12每天三百字之连接与替代
冷眼看潮
不知道是不是好为人师,有时候还真想和别人分享一下我对某些现象的看法或者解释。人类社会不断发展进步的过程,就是不断连接与替代的过程。人类发现了火并应用火以后,告别了茹毛饮血的野兽般的原始生活(火烧、烹饪替代了生食)人类用石器代替了完全手工,工具的使用使人类进步一大步。类似这样的替代还有很多,随着科技的发展,有更多的原始的事物被替代,代之以更高效、更先进的技术。在近现代,汽车替代了马车,高速公路和铁路
- 探索OpenAI和LangChain的适配器集成:轻松切换模型提供商
nseejrukjhad
langchaineasyui前端python
#探索OpenAI和LangChain的适配器集成:轻松切换模型提供商##引言在人工智能和自然语言处理的世界中,OpenAI的模型提供了强大的能力。然而,随着技术的发展,许多人开始探索其他模型以满足特定需求。LangChain作为一个强大的工具,集成了多种模型提供商,通过提供适配器,简化了不同模型之间的转换。本篇文章将介绍如何使用LangChain的适配器与OpenAI集成,以便轻松切换模型提供商
- 使用Faiss进行高效相似度搜索
llzwxh888
faisspython
在现代AI应用中,快速和高效的相似度搜索是至关重要的。Faiss(FacebookAISimilaritySearch)是一个专门用于快速相似度搜索和聚类的库,特别适用于高维向量。本文将介绍如何使用Faiss来进行相似度搜索,并结合Python代码演示其基本用法。什么是Faiss?Faiss是一个由FacebookAIResearch团队开发的开源库,主要用于高维向量的相似性搜索和聚类。Faiss
- python是什么意思中文-在python中%是什么意思
编程大乐趣
Python中%有两种:1、数值运算:%代表取模,返回除法的余数。如:>>>7%212、%操作符(字符串格式化,stringformatting),说明如下:%[(name)][flags][width].[precision]typecode(name)为命名flags可以有+,-,''或0。+表示右对齐。-表示左对齐。''为一个空格,表示在正数的左侧填充一个空格,从而与负数对齐。0表示使用0填
- 使用Apify加载Twitter消息以进行微调的完整指南
nseejrukjhad
twittereasyui前端python
#使用Apify加载Twitter消息以进行微调的完整指南##引言在自然语言处理领域,微调模型以适应特定任务是提升模型性能的常见方法。本文将介绍如何使用Apify从Twitter导出聊天信息,以便进一步进行微调。##主要内容###使用Apify导出推文首先,我们需要从Twitter导出推文。Apify可以帮助我们做到这一点。通过Apify的强大功能,我们可以批量抓取和导出数据,适用于各类应用场景。
- 如何部分格式化提示模板:LangChain中的高级技巧
nseejrukjhad
langchainjava服务器python
标题:如何部分格式化提示模板:LangChain中的高级技巧内容:如何部分格式化提示模板:LangChain中的高级技巧引言在使用大型语言模型(LLM)时,提示工程是一个关键环节。LangChain提供了强大的提示模板功能,让我们能更灵活地构建和管理提示。本文将介绍LangChain中一个高级特性-部分格式化提示模板,这个技巧可以让你的提示管理更加高效和灵活。什么是部分格式化提示模板?部分格式化提
- 有舍才有得
_清净_
为什么经常讲放下?放下就是让你要舍得、舍去。喜舍心就是把自己喜欢的,用慈悲心喜舍出去。这就锻炼了你们在人间,学会放下原本不舍得的东西或一些事物,学会舍出去,学会帮助别人,学会多付出。你今天付出了慈悲心、喜舍心,以后会得到更多的缘助力。缘助力是什么?——贵人缘啊。今天没有付出,不懂得付出,什么都只会想到自己,那你也得不到缘助力。慈悲喜舍就是用慈悲心去帮助别人,用喜舍心去付出,最后也会得到别人回报。别
- Day1笔记-Python简介&标识符和关键字&输入输出
~在杰难逃~
Pythonpython开发语言大数据数据分析数据挖掘
大家好,从今天开始呢,杰哥开展一个新的专栏,当然,数据分析部分也会不定时更新的,这个新的专栏主要是讲解一些Python的基础语法和知识,帮助0基础的小伙伴入门和学习Python,感兴趣的小伙伴可以开始认真学习啦!一、Python简介【了解】1.计算机工作原理编程语言就是用来定义计算机程序的形式语言。我们通过编程语言来编写程序代码,再通过语言处理程序执行向计算机发送指令,让计算机完成对应的工作,编程
- python八股文面试题分享及解析(1)
Shawn________
python
#1.'''a=1b=2不用中间变量交换a和b'''#1.a=1b=2a,b=b,aprint(a)print(b)结果:21#2.ll=[]foriinrange(3):ll.append({'num':i})print(11)结果:#[{'num':0},{'num':1},{'num':2}]#3.kk=[]a={'num':0}foriinrange(3):#0,12#可变类型,不仅仅改变
- 人工智能时代,程序员如何保持核心竞争力?
jmoych
人工智能
随着AIGC(如chatgpt、midjourney、claude等)大语言模型接二连三的涌现,AI辅助编程工具日益普及,程序员的工作方式正在发生深刻变革。有人担心AI可能取代部分编程工作,也有人认为AI是提高效率的得力助手。面对这一趋势,程序员应该如何应对?是专注于某个领域深耕细作,还是广泛学习以适应快速变化的技术环境?又或者,我们是否应该将重点转向AI无法轻易替代的软技能?让我们一起探讨程序员
- 每日算法&面试题,大厂特训二十八天——第二十天(树)
肥学
⚡算法题⚡面试题每日精进java算法数据结构
目录标题导读算法特训二十八天面试题点击直接资料领取导读肥友们为了更好的去帮助新同学适应算法和面试题,最近我们开始进行专项突击一步一步来。上一期我们完成了动态规划二十一天现在我们进行下一项对各类算法进行二十八天的一个小总结。还在等什么快来一起肥学进行二十八天挑战吧!!特别介绍小白练手专栏,适合刚入手的新人欢迎订阅编程小白进阶python有趣练手项目里面包括了像《机器人尬聊》《恶搞程序》这样的有趣文章
- Python快速入门 —— 第三节:类与对象
孤华暗香
Python快速入门python开发语言
第三节:类与对象目标:了解面向对象编程的基础概念,并学会如何定义类和创建对象。内容:类与对象:定义类:class关键字。类的构造函数:__init__()。类的属性和方法。对象的创建与使用。示例:classStudent:def__init__(self,name,age,major):self.name&#
- java杨辉三角
3213213333332132
java基础
package com.algorithm;
/**
* @Description 杨辉三角
* @author FuJianyong
* 2015-1-22上午10:10:59
*/
public class YangHui {
public static void main(String[] args) {
//初始化二维数组长度
int[][] y
- 《大话重构》之大布局的辛酸历史
白糖_
重构
《大话重构》中提到“大布局你伤不起”,如果企图重构一个陈旧的大型系统是有非常大的风险,重构不是想象中那么简单。我目前所在公司正好对产品做了一次“大布局重构”,下面我就分享这个“大布局”项目经验给大家。
背景
公司专注于企业级管理产品软件,企业有大中小之分,在2000年初公司用JSP/Servlet开发了一套针对中
- 电驴链接在线视频播放源码
dubinwei
源码电驴播放器视频ed2k
本项目是个搜索电驴(ed2k)链接的应用,借助于磁力视频播放器(官网:
http://loveandroid.duapp.com/ 开放平台),可以实现在线播放视频,也可以用迅雷或者其他下载工具下载。
项目源码:
http://git.oschina.net/svo/Emule,动态更新。也可从附件中下载。
项目源码依赖于两个库项目,库项目一链接:
http://git.oschina.
- Javascript中函数的toString()方法
周凡杨
JavaScriptjstoStringfunctionobject
简述
The toString() method returns a string representing the source code of the function.
简译之,Javascript的toString()方法返回一个代表函数源代码的字符串。
句法
function.
- struts处理自定义异常
g21121
struts
很多时候我们会用到自定义异常来表示特定的错误情况,自定义异常比较简单,只要分清是运行时异常还是非运行时异常即可,运行时异常不需要捕获,继承自RuntimeException,是由容器自己抛出,例如空指针异常。
非运行时异常继承自Exception,在抛出后需要捕获,例如文件未找到异常。
此处我们用的是非运行时异常,首先定义一个异常LoginException:
/**
* 类描述:登录相
- Linux中find常见用法示例
510888780
linux
Linux中find常见用法示例
·find path -option [ -print ] [ -exec -ok command ] {} \;
find命令的参数;
- SpringMVC的各种参数绑定方式
Harry642
springMVC绑定表单
1. 基本数据类型(以int为例,其他类似):
Controller代码:
@RequestMapping("saysth.do")
public void test(int count) {
}
表单代码:
<form action="saysth.do" method="post&q
- Java 获取Oracle ROWID
aijuans
javaoracle
A ROWID is an identification tag unique for each row of an Oracle Database table. The ROWID can be thought of as a virtual column, containing the ID for each row.
The oracle.sql.ROWID class i
- java获取方法的参数名
antlove
javajdkparametermethodreflect
reflect.ClassInformationUtil.java
package reflect;
import javassist.ClassPool;
import javassist.CtClass;
import javassist.CtMethod;
import javassist.Modifier;
import javassist.bytecode.CodeAtt
- JAVA正则表达式匹配 查找 替换 提取操作
百合不是茶
java正则表达式替换提取查找
正则表达式的查找;主要是用到String类中的split();
String str;
str.split();方法中传入按照什么规则截取,返回一个String数组
常见的截取规则:
str.split("\\.")按照.来截取
str.
- Java中equals()与hashCode()方法详解
bijian1013
javasetequals()hashCode()
一.equals()方法详解
equals()方法在object类中定义如下:
public boolean equals(Object obj) {
return (this == obj);
}
很明显是对两个对象的地址值进行的比较(即比较引用是否相同)。但是我们知道,String 、Math、I
- 精通Oracle10编程SQL(4)使用SQL语句
bijian1013
oracle数据库plsql
--工资级别表
create table SALGRADE
(
GRADE NUMBER(10),
LOSAL NUMBER(10,2),
HISAL NUMBER(10,2)
)
insert into SALGRADE values(1,0,100);
insert into SALGRADE values(2,100,200);
inser
- 【Nginx二】Nginx作为静态文件HTTP服务器
bit1129
HTTP服务器
Nginx作为静态文件HTTP服务器
在本地系统中创建/data/www目录,存放html文件(包括index.html)
创建/data/images目录,存放imags图片
在主配置文件中添加http指令
http {
server {
listen 80;
server_name
- kafka获得最新partition offset
blackproof
kafkapartitionoffset最新
kafka获得partition下标,需要用到kafka的simpleconsumer
import java.util.ArrayList;
import java.util.Collections;
import java.util.Date;
import java.util.HashMap;
import java.util.List;
import java.
- centos 7安装docker两种方式
ronin47
第一种是采用yum 方式
yum install -y docker
- java-60-在O(1)时间删除链表结点
bylijinnan
java
public class DeleteNode_O1_Time {
/**
* Q 60 在O(1)时间删除链表结点
* 给定链表的头指针和一个结点指针(!!),在O(1)时间删除该结点
*
* Assume the list is:
* head->...->nodeToDelete->mNode->nNode->..
- nginx利用proxy_cache来缓存文件
cfyme
cache
user zhangy users;
worker_processes 10;
error_log /var/vlogs/nginx_error.log crit;
pid /var/vlogs/nginx.pid;
#Specifies the value for ma
- [JWFD开源工作流]JWFD嵌入式语法分析器负号的使用问题
comsci
嵌入式
假如我们需要用JWFD的语法分析模块定义一个带负号的方程式,直接在方程式之前添加负号是不正确的,而必须这样做:
string str01 = "a=3.14;b=2.71;c=0;c-((a*a)+(b*b))"
定义一个0整数c,然后用这个整数c去
- 如何集成支付宝官方文档
dai_lm
android
官方文档下载地址
https://b.alipay.com/order/productDetail.htm?productId=2012120700377310&tabId=4#ps-tabinfo-hash
集成的必要条件
1. 需要有自己的Server接收支付宝的消息
2. 需要先制作app,然后提交支付宝审核,通过后才能集成
调试的时候估计会真的扣款,请注意
- 应该在什么时候使用Hadoop
datamachine
hadoop
原帖地址:http://blog.chinaunix.net/uid-301743-id-3925358.html
存档,某些观点与我不谋而合,过度技术化不可取,且hadoop并非万能。
--------------------------------------------万能的分割线--------------------------------
有人问我,“你在大数据和Hado
- 在GridView中对于有外键的字段使用关联模型进行搜索和排序
dcj3sjt126com
yii
在GridView中使用关联模型进行搜索和排序
首先我们有两个模型它们直接有关联:
class Author extends CActiveRecord {
...
}
class Post extends CActiveRecord {
...
function relations() {
return array(
'
- 使用NSString 的格式化大全
dcj3sjt126com
Objective-C
格式定义The format specifiers supported by the NSString formatting methods and CFString formatting functions follow the IEEE printf specification; the specifiers are summarized in Table 1. Note that you c
- 使用activeX插件对象object滚动有重影
蕃薯耀
activeX插件滚动有重影
使用activeX插件对象object滚动有重影 <object style="width:0;" id="abc" classid="CLSID:D3E3970F-2927-9680-BBB4-5D0889909DF6" codebase="activex/OAX339.CAB#
- SpringMVC4零配置
hanqunfeng
springmvc4
基于Servlet3.0规范和SpringMVC4注解式配置方式,实现零xml配置,弄了个小demo,供交流讨论。
项目说明如下:
1.db.sql是项目中用到的表,数据库使用的是oracle11g
2.该项目使用mvn进行管理,私服为自搭建nexus,项目只用到一个第三方 jar,就是oracle的驱动;
3.默认项目为零配置启动,如果需要更改启动方式,请
- 《开源框架那点事儿16》:缓存相关代码的演变
j2eetop
开源框架
问题引入
上次我参与某个大型项目的优化工作,由于系统要求有比较高的TPS,因此就免不了要使用缓冲。
该项目中用的缓冲比较多,有MemCache,有Redis,有的还需要提供二级缓冲,也就是说应用服务器这层也可以设置一些缓冲。
当然去看相关实现代代码的时候,大致是下面的样子。
[java]
view plain
copy
print
?
public vo
- AngularJS浅析
kvhur
JavaScript
概念
AngularJS is a structural framework for dynamic web apps.
了解更多详情请见原文链接:http://www.gbtags.com/gb/share/5726.htm
Directive
扩展html,给html添加声明语句,以便实现自己的需求。对于页面中html元素以ng为前缀的属性名称,ng是angular的命名空间
- 架构师之jdk的bug排查(一)---------------split的点号陷阱
nannan408
split
1.前言.
jdk1.6的lang包的split方法是有bug的,它不能有效识别A.b.c这种类型,导致截取长度始终是0.而对于其他字符,则无此问题.不知道官方有没有修复这个bug.
2.代码
String[] paths = "object.object2.prop11".split("'");
System.ou
- 如何对10亿数据量级的mongoDB作高效的全表扫描
quentinXXZ
mongodb
本文链接:
http://quentinXXZ.iteye.com/blog/2149440
一、正常情况下,不应该有这种需求
首先,大家应该有个概念,标题中的这个问题,在大多情况下是一个伪命题,不应该被提出来。要知道,对于一般较大数据量的数据库,全表查询,这种操作一般情况下是不应该出现的,在做正常查询的时候,如果是范围查询,你至少应该要加上limit。
说一下,
- C语言算法之水仙花数
qiufeihu
c算法
/**
* 水仙花数
*/
#include <stdio.h>
#define N 10
int main()
{
int x,y,z;
for(x=1;x<=N;x++)
for(y=0;y<=N;y++)
for(z=0;z<=N;z++)
if(x*100+y*10+z == x*x*x
- JSP指令
wyzuomumu
jsp
jsp指令的一般语法格式: <%@ 指令名 属性 =”值 ” %>
常用的三种指令: page,include,taglib
page指令语法形式: <%@ page 属性 1=”值 1” 属性 2=”值 2”%>
include指令语法形式: <%@include file=”relative url”%> (jsp可以通过 include