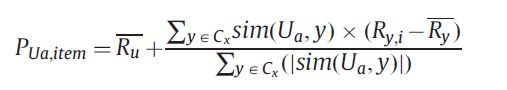基于遗传算法和Kmeans的电影推荐算法
本文参考文献:https://www.sciencedirect.com/science/article/pii/S1045926X14000901
1.基于用户的协同滤波算法
在我的前一篇文章中我介绍了一种基于用户喜好和电影类型进行推荐的算法。这次我来介绍一种将遗传算法和Kmeans结合的电影推荐算法。
我们知道基于用户的协同滤波算法是一种十分热门的算法。这种算法的目的是找到跟你相似的用户,然后向你推荐这些用户喜欢的物品。可以看出来这里的关键是找到相似的用户。这其实可以看作一个聚类问题,即把同一类型的用户分成同一类,然后在进行推荐时就可以只在同一类的用户中计算推荐的物品。
因此我们首先将推荐问题转化为聚类问题。
2.遗传算法和Kmeans
Kmeans是非常著名的无监督方法,可以将样品分为不同的类。但是这种方法很受初始点选择的影响。如果我们选择了合适的初始点,那么聚类的效果会好很多。
问题来了: Kmeans选择初始点时是随机的,我们怎么去挑选适合的初始点呢?
于是就利用到遗传算法。遗传算法是一种模拟物种进化的算法,一开始我们会有不同的个体,并且有一个Objective function。Objective function可以计算出每个个体对环境的适应值,如果适应值高,则有更大的机率遗传到下一代。
因此在这里,假设我们要将用户分为5类,则每个个体表示Kmeans的初始点(p1,p2,p3,p4,p5),而Objective function计算的是利用该个体进行Kmeans分类的效果。
通过遗传算法不同的演变,我们就可以找到最适合的Kmeans初始点。
3.算法实施步骤
(1)PCA降维
一般在进行聚类运算前,我们都会作降维操作。因此我们首先构建用户-电影评分表,然后作PCA降维。具体降到多少维,一般取保留95%以上的信息的维度即可。
完成降维后我们会得到这样的一张表。其中Index是用户ID(这里显示Index是movieId, 不要在意这个细节。),columns即是维度。
(2)遗传算法计算起始点
利用遗传算法计算最佳起始点。
假设我们打算将用户分为13类。并且假设一共有50个个体,则我们随机生成50个个体(p1,p2,p3.....p50)。其中每一个个体都由13个点组成。Objective function(p1)计算的是所有数据点到最近的p1的初始点的距离的和。也就是说我们计算每个数据点和与之最近的p1中的初始点的距离,然后把所有的距离加起来。所以如果算出来的结果越小,说明这个个体选择的初始点划分效果更好,适应度更高。
大体介绍完成了,我们开始介绍遗传的过程。 在遗传过程中我们依次进行以下操作:
1.利用Objective function计算每个个体的适应程度。
2.进行选择操作。即按照一定概率选择个体,被选中的个体直接遗传到下一代。其中适应度越高的个体被选中的几率越大。总共选择50个,即部分个体会重复。
3.进行交叉操作。按照一定概率选择多对个体。然后按照下图的方式,利用每队个体Ci1和Ci2创造出新的一对个体Ci1‘和Ci2’,接着利用Objective function对比这四个个体,并留下适应度高的两个。
4.变异操作。按照一定概率选择个体,被选中的个体进行如下变异。
5.重复以上操作直至迭代一定的次数。该次数由自己设定。
最后我们会得到50个个体,然后我们计算出适应度最高的那个就是我们希望求得的最佳Kmeans起始点。(在本人实践过程中最后得到的所有个体的值是一样的。)
(3)进行推荐
最后我们将利用计算出来的起始点放入Kmeans模型中对所有用户进行聚类。然后利用协同滤波算法进行推荐。下面公式计算用户Ua对item i的评分。其中Cx表示用户Ua属于的类,Ru表示用户Ua的平均评分。
整个流程图如下。
附上本人写的代码。这里我做的是基于物品的协同滤波,代码比较凌乱,有一些语句可能也用不上。
from sklearn.decomposition import PCA
import numpy as np
import pandas as pd
from sklearn.metrics import mean_squared_error
from sklearn.cluster import KMeans
from sklearn.model_selection import train_test_split
from sklearn.metrics import pairwise_distances
def fresh_property(population_current):
sum = 0
for i in range(Population_size):
# print(i)
population_current[i][1]['fitness'] = objective_function(population_current[i])
sum += population_current[i][1]['fitness']
population_current[0][1]['rate_fit'] = population_current[0][1]['fitness'] / sum
population_current[0][1]['cumm_fit'] = population_current[0][1]['rate_fit']
for i in range(Population_size):
population_current[i][1]['rate_fit'] = population_current[i][1]['fitness'] / sum
population_current[i][1]['cumm_fit'] = population_current[i][1]['rate_fit'] + population_current[i-1][1]['cumm_fit']
print('finish_fresh')
def objective_function(individual):
distance = 0
for index in pca_d:
distance += (np.subtract(individual[0],pca_d[index])**2).sum(axis=1).min()
return distance
def select(population_current,population_next):
for i in range(Population_size):
# print('select',i)
rand = np.random.rand(1)
if rand <= population_current[0][1]['cumm_fit']:
population_next[i] = population_current[0]
else:
for j in range(Population_size):
if population_current[j][1]['cumm_fit'] <= rand and population_current[j+1][1]['cumm_fit'] >= rand:
population_next[i] = population_current[j+1]
break
def crossover(population_next):
for i in range(Population_size):
# print('crossover',i)
rand = np.random.rand(1)
if rand <= Probability_crossover:
rand_cluster = np.random.randint(Cluster_number)
p1_num = np.random.randint(Population_size)
p2_num = np.random.randint(Population_size)
p1 = population_next[p1_num]
p2 = population_next[p2_num]
c1 = p1
c2 = p2
c1[0] = np.vstack([p1[0][:rand_cluster,:],p2[0][rand_cluster:,:]])
c2[0] = np.vstack([p2[0][:rand_cluster,:],p1[0][rand_cluster:,:]])
test_c = [[],[]]
test_c[0].extend([objective_function(c1),objective_function(c2),objective_function(p1),objective_function(p2)])
test_c[1].extend([c1,c2,p1,p2])
population_next[p1_num] = test_c[1][test_c[0].index(min(test_c[0]))]
test_c[1] = test_c[1][:test_c[0].index(min(test_c[0]))] + test_c[1][test_c[0].index(min(test_c[0]))+1:]
test_c[0].remove(min(test_c[0]))
population_next[p2_num] = test_c[1][test_c[0].index(min(test_c[0]))]
def mutation(population_next):
for i in range(Population_size):
# print('mutation')
rand = np.random.rand(1)
if rand <= Probability_mutation:
mutation_array = np.ones([Cluster_number,Dimension_number])
for k in range(Cluster_number):
rand_pick = np.random.randint(Population_size)
mutation_array[k] = population_next[rand_pick][0][k]
if objective_function([mutation_array]) < objective_function(population_next[i]):
population_next[i][0] = mutation_array
def user_predict(train_data,label,euc,test_data,k,user_mean,movie_mean):
prediction_set = []
new = 0
last = 0
for i in range(len(test_data)):
user = int(test_data.iloc[i].userId)
movie = int(test_data.iloc[i].movieId)
new = user
if movie not in train_data.index:
prediction_set.append(user_mean[user])
continue
if new != last:
cluster_label = label.loc[movie].label
mean = movie_mean[movie]
k_similar_index = []
if len(label[label['label'] == cluster_label]) < k:
k_similar_index.extend(euc[movie][label[label['label'] == cluster_label].index].index)
else:
k_similar_index.extend(euc[movie][label[label['label'] == cluster_label].index].sort_values(ascending=True)[:k].index)
add_up = 0
add_down = 0
for similar_index in k_similar_index:
if train_data[user][similar_index] != 0:
similar_mean = movie_mean[similar_index]
add_up = add_up + euc[movie][similar_index] * (train_data[user][similar_index] - similar_mean)
add_down = add_down + abs(euc[movie][similar_index])
if add_down == 0:
prediction = mean
else:
prediction = mean+add_up/add_down
if(prediction > 5):
prediction = 5
if(prediction < 0):
predition = 0
prediction_set.append(prediction)
last = new
return prediction_set
error_set = []
#ga_file = open('ga','w')
#设置参数
Population_size = 40
Cluster_number = 10
Dimension_number = 500
iteration_num = 100
Probability_crossover = 0.5
Probability_mutation = 0.0001;
train,test = train_test_split(rating,test_size = 0.2,random_state=0)
test = test.sort_index()
item_based = train.pivot_table(index='movieId', columns='userId', values='rating')
user_mean = item_based.mean(axis=0)
movie_mean = item_based.mean(axis=1)
item_based = item_based.fillna(0)
#PCA降维
pca = PCA(n_components=Dimension_number)
pca_data = pd.DataFrame(pca.fit_transform(item_based),index=item_based.index)
min_max = []
min_max.append(pca_data.max())
min_max.append(pca_data.min())
pca_data = pca_data.T.to_dict()
pca_d = {}
for i in pca_data:
pca_d[i] = list(pca_data[i].values())
#cluster_number设置为10
cluster_num=10
#初始化个体
population_current = []
population_next = []
for i in range(Population_size):
gene_array = np.array([])
for j in range(Dimension_number):
gene = np.random.uniform(min_max[0][j],min_max[1][j],(Cluster_number,1))
if len(gene_array) == 0:
gene_array = gene
else:
gene_array = np.hstack([gene_array,gene])
population_current.append([gene_array,{'rate_fit':0,'cumm_fit':0,'fitness':0}])
population_next = population_current[:]
fresh_property(population_current)
#开始进行遗传操作
for i in range(iteration_num):
print('iteration',i)
select(population_current,population_next)
crossover(population_next)
mutation(population_next)
fresh_property(population_next)
population_current = population_next[:]
#利用计算出来的初始点进行聚类
kmeans = KMeans(n_clusters=cluster_num,init=population_next[0][0])
kmeans.fit(pca_data)
label = pd.DataFrame(kmeans.labels_,index = item_based.index,columns=['label'])
route = str(cluster_num)+'.csv'
label.to_csv(route)
'''
euc = pd.DataFrame(pairwise_distances(item_based,metric='euclidean'),index=item_based.index,columns=item_based.index)
error = np.sqrt(mean_squared_error(test.rating,user_predict(item_based,label,euc,test,100,user_mean,movie_mean)))
ga_file.write('cluseter_num ')
ga_file.write(str(cluster_num))
ga_file.write(': ')
ga_file.write(str(error))
ga_file.write('\n')
ga_file.close()
'''