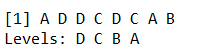R-数据清洗(附代码,图片)
数据清洗是将原始的数据进行整理和规范,以达到数据分析人员使用要求的数据。这个过程很重要,也很花费时间。现将当前学到的方式总结,欢迎大家互相交流。
1.缺失值处理
在R中,当原始数据中存在缺失值时,该缺失值用NA表示,如下图有一个缺失值。
birth=read.csv("chds_births.csv",header = TRUE)
head(birth)

若某一列数据缺失过多(>30%),那么这一列实际上就已经丢掉了很多关键的信息,可以从数据集中直接去掉这一列。若缺失的不是很多数据,则需要对该列进行填充。
1.1 缺失率计算
1.查看某列缺失情况
#查看某列是否缺失
is.na(birth$gestation)
该函数返回一个BOOL类型的数组,若缺失,则对应位置为TRUE,否则为FALSE.
2.查看数据集缺失情况
#检查数据集内是否含有缺失值
anyNA(birth)
若填充完毕后,想要查看该数据集是否还有缺失值,则该函数可以进行检验。若还有缺失值,则返回TRUE,否则返回FALSE.
3.计算缺失率
#查看缺失率
sapply(birth,f unction(df){sum(is.na(df)/nrow(birth))})

如上图所示,计算了每列的缺失比率。我们发现fht这一列缺失率高达39.8%,这一列原则上可以删除。
4.缺失情况可视化
方法一:
#缺失率可视化
library(Rcpp)
library(Amelia)
missmap(birth,main="missing map")
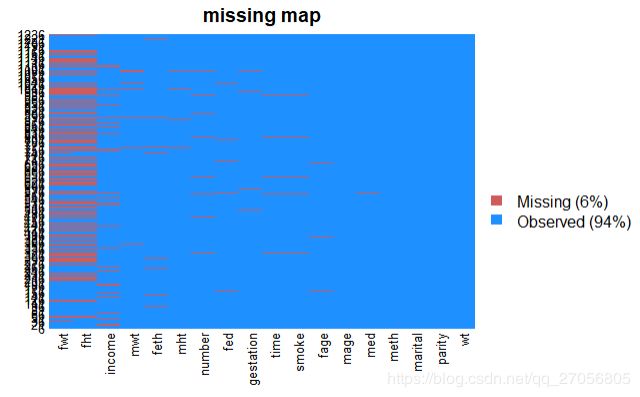
当然R也提供可视化缺失率的包,在可视化之前需要安装Rcpp包和Amelia包。先安装Rccp包,再安装Amelia包。如上面的代码可见。从上图也可以看出fwt和fht两个变量缺失率很高。
方法二:
这个可视化比上一种方法易于理解。
VIM包内的aggr()函数会生成缺失个数(或缺失率)的柱状图,显示效果更直观。
prop:TRUE表示显示缺失率,FALSE表示显示缺失个数
numbers: 默认FALSE,表示删除数值型标签。
library(VIM)
aggr(birth,prop=TRUE,numbers=TRUE)
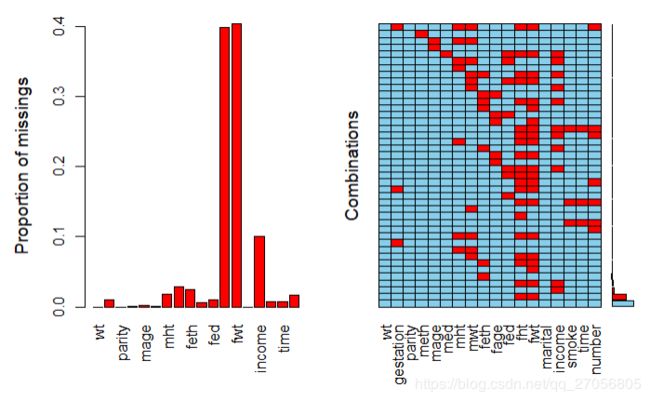
(貌似Rstudio3.5不支持直接下载,需要先下载安装包,然后本地安装)
1.2 缺失值填充
对于numeric数据类型而言,最常用的方法是用均值或中间值进行插补,而对于分类变量则用众数进行插补。
- 用均值进行插补
#对wt列用均值进行插补,na.rm=TRUE表示忽略缺失值,该参数必须写上
births$wt[is.na(birth$wt)]=mean(birth$wt,na.rm = TRUE)
- 用中间值进行插补
births$wt[is.na(birth$wt)]=median(birth$wt,na.rm = TRUE)
- 用众数进行插补
对于int型或者分类变量,需要用众数进行插补。
由于R中没有直接求众数的函数,因此需要自己写。当然你可以把下面代码封装成一个函数,达到更加通用的目的。
table=sort(birth$gestation,decreasing=TRUE)
mode=as.data.frame(table)[1,1]
birth$gestation[is.na(birth$gestation)]=as.numeric(as.character(mode))
- 直接删掉缺失值
birth=na.omit(birth)
2.数据类型转化
有时候数据加载后,数据类型并不是我们想要的类型,这时候就需要进行数据类型转化。常用的几个数据类型转化函数如下:
2.1 基本类型转化
#转化成数值型(R中没有转化成int型的函数)
as.numeric()
#转化成字符型
as.character()
#转化成数据集
as.data.frame()
#转化成矩阵
as.matrix()
#转化成BOOL类型
as.logical()
#转化成因子型
as.factor()
#但更常用的是直接使用factor()函数
factor(x,levels=c(),labels=c(),ordered=FALSE)
这里重点讲解一下因子型的转化。因子是R语言中表示分类变量的一种数据类型,因子变量是不能像数值型变量进行加减乘除操作的,尽管它有时看起来就是1和0组成的。
分类变量可以分为有序变量和无序变量。像名族,国家,性别这样的变量是无序变量,因为我们不关注它的顺序。像学历等级,疾病治疗情况这样的变量,可以看做有序分类变量,因为他代表了一定的方向性。
factor()函数的强大功能:
1.有序变量和无序变量是通过factor()函数的ordered参数来设定的。
2.可以为因子的水平重新命名。(有时候水平太抽象不直观)
factor(x,levels=c(),labels=c(),ordered=FALSE)
x: 待转化的向量;
levels: x中原始的取值。
labels: 取代原始列中取值的一个替代名称。注意:levels内的变量和labels内的变量是一一对应的。
ordered: 用于将x变量是否设置为有序变量。
country=c("中国","美国","中国","日本")
str(country)
country1=factor(country,levels = c("美国","中国","日本"),labels = c(1,2,3))
str(country1)

这里我们看出country原来是字符变量。通过factor函数后转化成因子变量。由于我们通过labels参数为三个国家编号为:美国-1,中国-2,日本-3,所以最后是以编号显示国家的。同时也可以看出levels和labels参数是一一对应的。
2.2 哑变量(dummay variable)生成
哑变量采用model.matrix(formula,data)函数生成。该函数使用前需要满足以下几个条件:
1.参数formula的~后面的参数类型必须是factor类型,且是需要转化的目标列。
2.~前面的是需要被排除的列。
3.后面需要添加一个-1,原因是该函数会返回截距项列,而该列是不需要的。-1表示不返回截距列。
4.该函数返回值后需要通过as.data.frame()转化成数据集格式。
5.data数据必须是数据集格式。
#y1,y2是data中不需要转化的列,x1,x2,x3是需要转化且为factor的列,-1表示不返回截距列
model.matrix(y1+y2+..~x1+x2+x3...-1,data=data)
2.3 变量的重编码(字符型变量转化成分类变量)
重编码即按区间将一系列数据分成若干组。如将>=90分为优秀,80-90为良好等情况。
score=c(94,23,56,78,42,60,90,82)
#breaks为划分的依据,若为整数,表示划分成几组。
#labels为划分后标签,right默认为TRUE,即左开右闭,
#当为FALSE时,为左闭右开。这样90分就可以划分到A等级了。
#返回值类型为factor类型,这很酷。
score=cut(score,breaks=c(0,60,80,90,100),labels = c("D","C","B","A"),right = FALSE)
score
3.高相关性变量诊断与剔除
在构建模型,尤其是线性回归模型过程中,对变量的要求是很高的。变量之间不能是高相关性的,不能是低方差,不能是强共线性的。若存在这些情况,需要将对应变量进行剔除。
R中的caret包可以完成上述三种情况变量的剔除。
library(caret)
#这里我们选用三列numeric数据进行测试
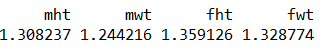
data=birth[,names(birth)%in%c("mht","mwt","fht","fwt")]
head(data)
#先计算data的相关系数
cor=cor(data)
cor
#该函数会返回需要剔除的列的列索引(即剔除相关性强的两列中的一列,保留另一列)
#cutoff是剔除的阈值,即相关性达到该数后会被剔除。
index=findCorrelation(cor,cutoff=0.45)
index
#输出要剔除的列的名称
names(data)[index]


由上图可知,fht和fwt之间相关性为0.482>0.45,这里将第四列fwt返回,表示可以剔除。
4. 低方差变量诊断与剔除
利用caret包内的nearZeroVar()函数查找低方差的列
library(caret)
#这里令y的方差为0
data=data.frame(x=c(1,2,3,4,5,6),y=c(1,1,1,1,1,1))
zerovar=nearZeroVar(data)
zerovar
#删除方差较小的列
newdata=data[,-zerovar]
5.共线性变量诊断与剔除
方法一:
利用caret包内的findLinearCombos()函数查找并剔除高共线性变量。
library(caret)
#同上,该函数返回可以直接剔除的共线性变量的列索引
findLinearCombos(data)
方法二:
car包内的vif(model)函数也能够检测多重共线性。但是该函数的参数必须是lm()或glm()函数生成的模型。该函数返回值为每列的vif值,若vif>4就表示该列存在严重的多重共线性,需要删除。
library(car)
fit=lm(wt~mht+mwt+fht+fwt,data=birth)
vif(fit)